+ データを開く
データを開く
- 基本情報
基本情報
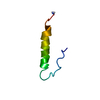
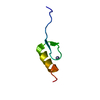
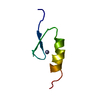
| 登録情報 | データベース: PDB / ID: 1bhi | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | STRUCTURE OF TRANSACTIVATION DOMAIN OF CRE-BP1/ATF-2, NMR, 20 STRUCTURES | ||||||
 要素 要素 | CRE-BP1 | ||||||
 キーワード キーワード | DNA-BINDING REGULATORY PROTEIN / CRE BINDING PROTEIN / ATF-2 / TRANSCRIPTIONAL ACTIVATION DOMAIN /  ZN FINGER (ジンクフィンガー) ZN FINGER (ジンクフィンガー) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報abducens nucleus development / hypoglossal nucleus development / detection of cell density / H4 histone acetyltransferase complex / growth plate cartilage chondrocyte proliferation / facial nucleus development / growth plate cartilage chondrocyte differentiation / positive regulation of cardiac muscle myoblast proliferation / cellular response to anisomycin / positive regulation of transforming growth factor beta2 production ...abducens nucleus development / hypoglossal nucleus development / detection of cell density / H4 histone acetyltransferase complex / growth plate cartilage chondrocyte proliferation / facial nucleus development / growth plate cartilage chondrocyte differentiation / positive regulation of cardiac muscle myoblast proliferation / cellular response to anisomycin / positive regulation of transforming growth factor beta2 production /  cAMP response element binding / cellular lipid metabolic process / cAMP response element binding / cellular lipid metabolic process /  leucine zipper domain binding / cAMP response element binding protein binding / histone H2B acetyltransferase activity / positive regulation of mitochondrial membrane permeability involved in apoptotic process / cellular response to leucine starvation / brainstem development / histone H4 acetyltransferase activity / NK T cell differentiation / vacuole organization / neurofilament cytoskeleton organization / apoptotic process involved in development / NGF-stimulated transcription / intrinsic apoptotic signaling pathway in response to hypoxia / mitotic intra-S DNA damage checkpoint signaling / motor neuron apoptotic process / response to osmotic stress / hepatocyte apoptotic process / outflow tract morphogenesis / Activation of the AP-1 family of transcription factors / p38MAPK cascade / Response of EIF2AK4 (GCN2) to amino acid deficiency / white fat cell differentiation / BMP signaling pathway / adipose tissue development / cis-regulatory region sequence-specific DNA binding / hematopoietic progenitor cell differentiation / leucine zipper domain binding / cAMP response element binding protein binding / histone H2B acetyltransferase activity / positive regulation of mitochondrial membrane permeability involved in apoptotic process / cellular response to leucine starvation / brainstem development / histone H4 acetyltransferase activity / NK T cell differentiation / vacuole organization / neurofilament cytoskeleton organization / apoptotic process involved in development / NGF-stimulated transcription / intrinsic apoptotic signaling pathway in response to hypoxia / mitotic intra-S DNA damage checkpoint signaling / motor neuron apoptotic process / response to osmotic stress / hepatocyte apoptotic process / outflow tract morphogenesis / Activation of the AP-1 family of transcription factors / p38MAPK cascade / Response of EIF2AK4 (GCN2) to amino acid deficiency / white fat cell differentiation / BMP signaling pathway / adipose tissue development / cis-regulatory region sequence-specific DNA binding / hematopoietic progenitor cell differentiation /  histone acetyltransferase activity / JNK cascade / transcription initiation-coupled chromatin remodeling / negative regulation of angiogenesis / liver development / Regulation of PTEN gene transcription / promoter-specific chromatin binding / TP53 Regulates Transcription of DNA Repair Genes / RNA polymerase II transcription regulatory region sequence-specific DNA binding / peptidyl-threonine phosphorylation / Heme signaling / mRNA transcription by RNA polymerase II / Transcriptional activation of mitochondrial biogenesis / response to organic cyclic compound / cellular response to virus / protein import into nucleus / RNA polymerase II transcription regulator complex / positive regulation of DNA-binding transcription factor activity / histone acetyltransferase activity / JNK cascade / transcription initiation-coupled chromatin remodeling / negative regulation of angiogenesis / liver development / Regulation of PTEN gene transcription / promoter-specific chromatin binding / TP53 Regulates Transcription of DNA Repair Genes / RNA polymerase II transcription regulatory region sequence-specific DNA binding / peptidyl-threonine phosphorylation / Heme signaling / mRNA transcription by RNA polymerase II / Transcriptional activation of mitochondrial biogenesis / response to organic cyclic compound / cellular response to virus / protein import into nucleus / RNA polymerase II transcription regulator complex / positive regulation of DNA-binding transcription factor activity /  Circadian Clock / sequence-specific double-stranded DNA binding / site of double-strand break / cellular response to oxidative stress / HATs acetylate histones / DNA-binding transcription activator activity, RNA polymerase II-specific / Estrogen-dependent gene expression / in utero embryonic development / mitochondrial outer membrane / RNA polymerase II-specific DNA-binding transcription factor binding / DNA-binding transcription factor activity, RNA polymerase II-specific / positive regulation of protein phosphorylation / RNA polymerase II cis-regulatory region sequence-specific DNA binding / DNA-binding transcription factor activity / protein heterodimerization activity / DNA damage response / Circadian Clock / sequence-specific double-stranded DNA binding / site of double-strand break / cellular response to oxidative stress / HATs acetylate histones / DNA-binding transcription activator activity, RNA polymerase II-specific / Estrogen-dependent gene expression / in utero embryonic development / mitochondrial outer membrane / RNA polymerase II-specific DNA-binding transcription factor binding / DNA-binding transcription factor activity, RNA polymerase II-specific / positive regulation of protein phosphorylation / RNA polymerase II cis-regulatory region sequence-specific DNA binding / DNA-binding transcription factor activity / protein heterodimerization activity / DNA damage response /  クロマチン / positive regulation of gene expression / regulation of DNA-templated transcription / regulation of transcription by RNA polymerase II / クロマチン / positive regulation of gene expression / regulation of DNA-templated transcription / regulation of transcription by RNA polymerase II /  protein kinase binding / negative regulation of transcription by RNA polymerase II / protein homodimerization activity / positive regulation of transcription by RNA polymerase II / protein kinase binding / negative regulation of transcription by RNA polymerase II / protein homodimerization activity / positive regulation of transcription by RNA polymerase II /  核質 / identical protein binding / 核質 / identical protein binding /  metal ion binding / metal ion binding /  細胞核 / 細胞核 /  細胞質 細胞質類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  溶液NMR / 溶液NMR /  SIMULATED ANNEALING, ENERGY MINIMIZATION SIMULATED ANNEALING, ENERGY MINIMIZATION | ||||||
 データ登録者 データ登録者 | Nagadoi, A. / Nakazawa, K. / Uda, H. / Maekawa, T. / Ishii, S. / Nishimura, Y. | ||||||
 引用 引用 |  ジャーナル: J.Mol.Biol. / 年: 1999 ジャーナル: J.Mol.Biol. / 年: 1999タイトル: Solution structure of the transactivation domain of ATF-2 comprising a zinc finger-like subdomain and a flexible subdomain. 著者: Nagadoi, A. / Nakazawa, K. / Uda, H. / Okuno, K. / Maekawa, T. / Ishii, S. / Nishimura, Y. #1:  ジャーナル: Genes Dev. / 年: 1990 ジャーナル: Genes Dev. / 年: 1990タイトル: Erratum. Transcription Factor ATF Cdna Clones: An Extensive Family of Leucine Zipper Proteins Able to Selectively Form DNA-Binding Heterodimers 著者: Hai, T.W. / Liu, F. / Coukos, W.J. / Green, M.R. #2:  ジャーナル: Genes Dev. / 年: 1989 ジャーナル: Genes Dev. / 年: 1989タイトル: Transcription Factor ATF Cdna Clones: An Extensive Family of Leucine Zipper Proteins Able to Selectively Form DNA-Binding Heterodimers 著者: Hai, T.W. / Liu, F. / Coukos, W.J. / Green, M.R. #3:  ジャーナル: Embo J. / 年: 1989 ジャーナル: Embo J. / 年: 1989タイトル: Leucine Zipper Structure of the Protein Cre-BP1 Binding to the Cyclic AMP Response Element in Brain 著者: Maekawa, T. / Sakura, H. / Kanei-Ishii, C. / Sudo, T. / Yoshimura, T. / Fujisawa, J. / Yoshida, M. / Ishii, S. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1bhi.cif.gz 1bhi.cif.gz | 258.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1bhi.ent.gz pdb1bhi.ent.gz | 226.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1bhi.json.gz 1bhi.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/bh/1bhi https://data.pdbj.org/pub/pdb/validation_reports/bh/1bhi ftp://data.pdbj.org/pub/pdb/validation_reports/bh/1bhi ftp://data.pdbj.org/pub/pdb/validation_reports/bh/1bhi | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質・ペプチド | 分子量: 4336.971 Da / 分子数: 1 断片: ZINC FINGER-LIKE SUBDOMAIN OF THE TRANSACTIVATION DOMAIN 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: CDNA / 生物種 (発現宿主): Escherichia coli / 発現宿主: Homo sapiens (ヒト) / 遺伝子: CDNA / 生物種 (発現宿主): Escherichia coli / 発現宿主:   Escherichia coli BL21 (大腸菌) / 株 (発現宿主): BL21 / 参照: UniProt: P15336 Escherichia coli BL21 (大腸菌) / 株 (発現宿主): BL21 / 参照: UniProt: P15336 |
|---|
-実験情報
-実験
| 実験 | 手法:  溶液NMR 溶液NMR |
|---|---|
| NMR実験の詳細 | Text: THE STRUCTURES OF N- AND C-TERMINI OF THE PEPTIDE FRAGMENT (MET 1 - PRO 6 AND LYS 36 - GLY 38) WERE DISORDERED AMONG THE 20 STRUCTURES. |
- 試料調製
試料調製
| 試料状態 | pH: 6.3 / 温度: 300 K |
|---|---|
結晶化 | *PLUS 手法: その他 / 詳細: NMR |
-NMR測定
| NMRスペクトロメーター | タイプ: Bruker DMX 600 / 製造業者: Bruker / モデル : DMX 600 / 磁場強度: 600 MHz : DMX 600 / 磁場強度: 600 MHz |
|---|
- 解析
解析
| NMR software |
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法:  SIMULATED ANNEALING, ENERGY MINIMIZATION / ソフトェア番号: 1 SIMULATED ANNEALING, ENERGY MINIMIZATION / ソフトェア番号: 1 | |||||||||
| NMRアンサンブル | コンフォーマー選択の基準: THE 20 FINAL STRUCTURES EXHIBITED NO DISTANCE RESTRAINT VIOLATIONS GREATER THAN 0.1 ANGSTROMS AND DIHEDRAL ANGLE VIOLATIONS GREATER THAN 6.0 DEGREES. 計算したコンフォーマーの数: 100 / 登録したコンフォーマーの数: 20 |
 ムービー
ムービー コントローラー
コントローラー








 PDBj
PDBj













