+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8661 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM reconstruction of the bacteriophage T4 isometric capsid | ||||||||||||
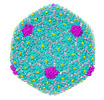
 マップデータ マップデータ | Cryo-EM reconstruction of the bacteriophage T4 isometric capsid | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード |  Bacteriophage T4 (T4ファージ) / isometric head / virus capsid assembly / triangulation numbers / size-determining mutations / capsid stabilization / capsid decoration proteins / Bacteriophage T4 (T4ファージ) / isometric head / virus capsid assembly / triangulation numbers / size-determining mutations / capsid stabilization / capsid decoration proteins /  VIRUS (ウイルス) VIRUS (ウイルス) | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |   Enterobacteria phage T4 (ファージ) Enterobacteria phage T4 (ファージ) | ||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.3 Å クライオ電子顕微鏡法 / 解像度: 3.3 Å | ||||||||||||
 データ登録者 データ登録者 | Chen Z / Sun L | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2017 ジャーナル: Proc Natl Acad Sci U S A / 年: 2017タイトル: Cryo-EM structure of the bacteriophage T4 isometric head at 3.3-Å resolution and its relevance to the assembly of icosahedral viruses. 著者: Zhenguo Chen / Lei Sun / Zhihong Zhang / Andrei Fokine / Victor Padilla-Sanchez / Dorit Hanein / Wen Jiang / Michael G Rossmann / Venigalla B Rao /  要旨: The 3.3-Å cryo-EM structure of the 860-Å-diameter isometric mutant bacteriophage T4 capsid has been determined. WT T4 has a prolate capsid characterized by triangulation numbers (T numbers) T = 13 ...The 3.3-Å cryo-EM structure of the 860-Å-diameter isometric mutant bacteriophage T4 capsid has been determined. WT T4 has a prolate capsid characterized by triangulation numbers (T numbers) T = 13 for end caps and T = 20 for midsection. A mutation in the major capsid protein, gp23, produced T=13 icosahedral capsids. The capsid is stabilized by 660 copies of the outer capsid protein, Soc, which clamp adjacent gp23 hexamers. The occupancies of Soc molecules are proportional to the size of the angle between the planes of adjacent hexameric capsomers. The angle between adjacent hexameric capsomers is greatest around the fivefold vertices, where there is the largest deviation from a planar hexagonal array. Thus, the Soc molecules reinforce the structure where there is the greatest strain in the gp23 hexagonal lattice. Mutations that change the angles between adjacent capsomers affect the positions of the pentameric vertices, resulting in different triangulation numbers in bacteriophage T4. The analysis of the T4 mutant head assembly gives guidance to how other icosahedral viruses reproducibly assemble into capsids with a predetermined T number, although the influence of scaffolding proteins is also important. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8661.map.gz emd_8661.map.gz | 12.7 GB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8661-v30.xml emd-8661-v30.xml emd-8661.xml emd-8661.xml | 20.2 KB 20.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8661.png emd_8661.png | 294.6 KB | ||
| Filedesc metadata |  emd-8661.cif.gz emd-8661.cif.gz | 7.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8661 http://ftp.pdbj.org/pub/emdb/structures/EMD-8661 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8661 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8661 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  5vf3MC  8t1xM M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8661.map.gz / 形式: CCP4 / 大きさ: 1.5 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8661.map.gz / 形式: CCP4 / 大きさ: 1.5 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM reconstruction of the bacteriophage T4 isometric capsid | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.81 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Enterobacteria phage T4
| 全体 | 名称:   Enterobacteria phage T4 (ファージ) Enterobacteria phage T4 (ファージ) |
|---|---|
| 要素 |
|
-超分子 #1: Enterobacteria phage T4
| 超分子 | 名称: Enterobacteria phage T4 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 10665 / 生物種: Enterobacteria phage T4 / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: Yes |
|---|---|
| 宿主 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| ウイルス殻 | Shell ID: 1 / 名称: gp23*-gp24*-Soc-Hoc / 直径: 860.0 Å / T番号(三角分割数): 13 |
-分子 #1: Capsid vertex protein gp24
| 分子 | 名称: Capsid vertex protein gp24 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Enterobacteria phage T4 (ファージ) Enterobacteria phage T4 (ファージ) |
| 分子量 | 理論値: 45.838574 KDa |
| 配列 | 文字列: STTTNSNSIG RPNLVALTRA TTKLIYSDIV ATQRTNQPVA AFYGIKYLNP DNEFTFKTGA TYAGEAGYVD REQITELTEE SKLTLNKGD LFKYNNIVYK VLEDTPFATI EESDLELALQ IAIVLLKVRL FSDAASTSKF ESSDSEIADA RFQINKWQTA V KSRKLKTG ...文字列: STTTNSNSIG RPNLVALTRA TTKLIYSDIV ATQRTNQPVA AFYGIKYLNP DNEFTFKTGA TYAGEAGYVD REQITELTEE SKLTLNKGD LFKYNNIVYK VLEDTPFATI EESDLELALQ IAIVLLKVRL FSDAASTSKF ESSDSEIADA RFQINKWQTA V KSRKLKTG ITVELAQDLE ANGFDAPNFL EDLLATEMAD EINKDILQSL ITVSKRYKVT GITDSGFIDL SYASAPEAGR SL YRMVCEM VSHIQKESTY TATFCVASAR AAAILAASGW LKHKPEDDKY LSQNAYGFLA NGLPLYCDTN SPLDYVIVGV VEN IGEKEI VGSIFYAPYT EGLDLDDPEH VGAFKVVVDP ESLQPSIGLL VRYALSANPY TVAKDEKEAR IIDGGDMDKM AGRS DLSVL LGVKLPKIII DE UniProtKB: Capsid vertex protein |
-分子 #2: Major capsid protein
| 分子 | 名称: Major capsid protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Enterobacteria phage T4 (ファージ) Enterobacteria phage T4 (ファージ) |
| 分子量 | 理論値: 48.728863 KDa |
| 配列 | 文字列: AEIGGDHGYN ATNIAAGQTS GAVTQIGPAV MGMVRRAIPN LIAFDICGVQ PMNSPTGQVF ALRAVYGKDP VAAGAKEAFH PMYGPDAMF SGQGAAKKFP ALAASTQTTV GDIYTHFFQE TGTVYLQASV QVTIDAGATD AAKLDAEIKK QMEAGALVEI A EGMATSIA ...文字列: AEIGGDHGYN ATNIAAGQTS GAVTQIGPAV MGMVRRAIPN LIAFDICGVQ PMNSPTGQVF ALRAVYGKDP VAAGAKEAFH PMYGPDAMF SGQGAAKKFP ALAASTQTTV GDIYTHFFQE TGTVYLQASV QVTIDAGATD AAKLDAEIKK QMEAGALVEI A EGMATSIA ELQEGFNGST DNPWNEMGFR IDKQVIEAKS RQLKAAYSIE LTQDLRAVHG MDADAELSGI LATEIMLEIN RE VVDWINY SAQVGKSGMT LTPGSKAGVF DFQDPIDIRG ARWAGESFKA LLFQIDKEAV EIARQTGRGE GNFIIASRNV VNV LASVDT GISYAAQGLA TGFSTDTTKS VFAGVLGGKY RVYIDQYAKQ DYFTVGYKGP NEMDAGIYYA PYVALTPLRG SDPK NFQPV MGFKTRYGIG INPFAESAAQ APASRIQSGM PSILNSLGKN AYFRRVYVKG I UniProtKB: Major capsid protein |
-分子 #3: Small outer capsid protein
| 分子 | 名称: Small outer capsid protein / タイプ: protein_or_peptide / ID: 3 / コピー数: 11 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Enterobacteria phage T4 (ファージ) Enterobacteria phage T4 (ファージ) |
| 分子量 | 理論値: 9.085095 KDa |
| 配列 | 文字列: MASARGYVNI KTFEQKLDGN KKIEGKEVSV AFPLYSDVHK ISGAHYQTFP SEKAAYSTVY EENQRTEWIA ANEDLWKVTG UniProtKB: Small outer capsid protein |
-分子 #4: Highly immunogenic outer capsid protein
| 分子 | 名称: Highly immunogenic outer capsid protein / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Enterobacteria phage T4 (ファージ) Enterobacteria phage T4 (ファージ) |
| 分子量 | 理論値: 40.416547 KDa |
| 配列 | 文字列: MTFTVDITPK TPTGVIDETK QFTATPSGQT GGGTITYAWS VDNVPQDGAE ATFSYVLKGP AGQKTIKVVA TNTLSEGGPE TAEATTTIT VKNKTQTTTL AVTPASPAAG VIGTPVQFTA ALASQPDGAS ATYQWYVDDS QVGGETNSTF SYTPTTSGVK R IKCVAQVT ...文字列: MTFTVDITPK TPTGVIDETK QFTATPSGQT GGGTITYAWS VDNVPQDGAE ATFSYVLKGP AGQKTIKVVA TNTLSEGGPE TAEATTTIT VKNKTQTTTL AVTPASPAAG VIGTPVQFTA ALASQPDGAS ATYQWYVDDS QVGGETNSTF SYTPTTSGVK R IKCVAQVT ATDYDALSVT SNEVSLTVNK KTMNPQVTLT PPSINVQQDA SATFTANVTG APEEAQITYS WKKDSSPVEG ST NVYTVDT SSVGSQTIEV TATVTAADYN PVTVTKTGNV TVTAKVAPEP EGELPYVHPL PHRSSAYIWC GWWVMDEIQK MTE EGKDWK TDDPDSKYYL HRYTLQKMMK DYPEVDVQES RNGYIIHKTA LETGIIYTYP UniProtKB: Highly immunogenic outer capsid protein |
-分子 #5: Highly immunogenic outer capsid protein
| 分子 | 名称: Highly immunogenic outer capsid protein / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Enterobacteria phage T4 (ファージ) Enterobacteria phage T4 (ファージ) |
| 分子量 | 理論値: 1.294587 KDa |
| 配列 | 文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK) |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | 2D array |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7 詳細: 50 mM sodium phosphate, pH 7.0, 75 mM sodium chloride, 1 mM magnesium chloride |
| グリッド | モデル: Ted Pella / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: LACEY / 支持フィルム - Film thickness: 3 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.04 kPa / 詳細: glow discharge for 60 seconds with 30 mA current |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 80 % / チャンバー内温度: 298 K / 装置: GATAN CRYOPLUNGE 3 / 詳細: Blot for 8 seconds before plunging.. |
| 詳細 | High-purity sample, volume = 50 uL, 10^12 particles |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): -3.5 µm / 最小 デフォーカス(補正後): -0.8 µm / 倍率(補正後): 18000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): -3.0 µm / 最小 デフォーカス(公称値): -1.0 µm / 倍率(公称値): 18000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): -3.0 µm / 最小 デフォーカス(公称値): -1.0 µm / 倍率(公称値): 18000 |
| 試料ステージ | 試料ホルダーモデル: OTHER / ホルダー冷却材: NITROGEN |
| 温度 | 最低: 90.0 K / 最高: 100.0 K |
| アライメント法 | Coma free - Residual tilt: 0.5 mrad |
| 詳細 | Data collection by Leginon |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 3710 pixel / デジタル化 - サイズ - 縦: 3838 pixel / デジタル化 - 画像ごとのフレーム数: 3-22 / 撮影したグリッド数: 1 / 実像数: 3000 / 平均露光時間: 9.0 sec. / 平均電子線量: 26.0 e/Å2 詳細: Images were collected in super-resolution mode at 40 frames per second. |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 19000 詳細: Approximately 1000 manually-picked particles were used for 2D classification. Templates were then generated from these 2D classes and used for automated particle picking. |
|---|---|
| 初期モデル | モデルのタイプ: NONE 詳細: EMAN was used to generate an initial model from about 1000 randomly-oriented particles. |
| 初期 角度割当 | タイプ: COMMON LINE 詳細: Randomly assigned for the particles used for initial model |
| 最終 3次元分類 | クラス数: 200 / 平均メンバー数/クラス: 15 / ソフトウェア - 名称: RELION 詳細: Relion was used for reference-free 2D classification. |
| 最終 角度割当 | タイプ: COMMON LINE / ソフトウェア - 名称: EMAN, JSPR / 詳細: EMAN and JSPR were used for refinement. |
| 最終 再構成 | 使用したクラス数: 60 / 想定した対称性 - 点群: I (正20面体型対称 ) / アルゴリズム: FOURIER SPACE / 解像度のタイプ: BY AUTHOR / 解像度: 3.3 Å / 解像度の算出法: FSC 0.143 CUT-OFF ) / アルゴリズム: FOURIER SPACE / 解像度のタイプ: BY AUTHOR / 解像度: 3.3 Å / 解像度の算出法: FSC 0.143 CUT-OFF詳細: Independent refinement of even/odd map, gold standard 使用した粒子像数: 18000 |
| 詳細 | The selected images were gain-normalized. |
 ムービー
ムービー コントローラー
コントローラー