+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7907 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
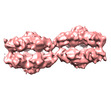
| タイトル | Two retinoschisin molecules interacting laterally forming a unit cell of a filamentous strand. | |||||||||
 マップデータ マップデータ | RS1 linear strand unit cell with two double octamer ring molecules. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Retinal adhesion protein X-linked retinoschisis Discoidin domain /  CELL ADHESION (細胞接着) CELL ADHESION (細胞接着) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報neuron to neuron synapse / retina layer formation / : /  eye development / phosphatidylinositol-3,4-bisphosphate binding / eye development / phosphatidylinositol-3,4-bisphosphate binding /  phosphatidylserine binding / photoreceptor inner segment / phosphatidylserine binding / photoreceptor inner segment /  視覚 / protein homooligomerization / 視覚 / protein homooligomerization /  細胞接着 ...neuron to neuron synapse / retina layer formation / : / 細胞接着 ...neuron to neuron synapse / retina layer formation / : /  eye development / phosphatidylinositol-3,4-bisphosphate binding / eye development / phosphatidylinositol-3,4-bisphosphate binding /  phosphatidylserine binding / photoreceptor inner segment / phosphatidylserine binding / photoreceptor inner segment /  視覚 / protein homooligomerization / 視覚 / protein homooligomerization /  細胞接着 / external side of plasma membrane / 細胞接着 / external side of plasma membrane /  extracellular space extracellular space類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
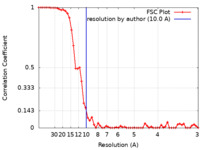
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 10.0 Å クライオ電子顕微鏡法 / 解像度: 10.0 Å | |||||||||
 データ登録者 データ登録者 | Heymann JB / Vijayasarathy C / Huang RK / Dearborn AD / Sieving PA / Steven AC | |||||||||
 引用 引用 |  ジャーナル: J Cell Biol / 年: 2019 ジャーナル: J Cell Biol / 年: 2019タイトル: Cryo-EM of retinoschisin branched networks suggests an intercellular adhesive scaffold in the retina. 著者: J Bernard Heymann / Camasamudram Vijayasarathy / Rick K Huang / Altaira D Dearborn / Paul A Sieving / Alasdair C Steven /  要旨: Mutations in the retinal protein retinoschisin (RS1) cause progressive loss of vision in young males, a form of macular degeneration called X-linked retinoschisis (XLRS). We previously solved the ...Mutations in the retinal protein retinoschisin (RS1) cause progressive loss of vision in young males, a form of macular degeneration called X-linked retinoschisis (XLRS). We previously solved the structure of RS1, a 16-mer composed of paired back-to-back octameric rings. Here, we show by cryo-electron microscopy that RS1 16-mers can assemble into extensive branched networks. We classified the different configurations, finding four types of interaction between the RS1 molecules. The predominant configuration is a linear strand with a wavy appearance. Three less frequent types constitute the branch points of the network. In all cases, the "spikes" around the periphery of the double rings are involved in these interactions. In the linear strand, a loop (usually referred to as spike 1) occurs on both sides of the interface between neighboring molecules. Mutations in this loop suppress secretion, indicating the possibility of intracellular higher-order assembly. These observations suggest that branched networks of RS1 may play a stabilizing role in maintaining the integrity of the retina. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7907.map.gz emd_7907.map.gz | 37.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7907-v30.xml emd-7907-v30.xml emd-7907.xml emd-7907.xml | 17.7 KB 17.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_7907_fsc.xml emd_7907_fsc.xml | 5.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_7907.png emd_7907.png | 93.6 KB | ||
| マスクデータ |  emd_7907_msk_1.map emd_7907_msk_1.map | 42.9 MB |  マスクマップ マスクマップ | |
| その他 |  emd_7907_half_map_1.map.gz emd_7907_half_map_1.map.gz emd_7907_half_map_2.map.gz emd_7907_half_map_2.map.gz | 39.5 MB 39.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7907 http://ftp.pdbj.org/pub/emdb/structures/EMD-7907 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7907 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7907 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7907.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7907.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RS1 linear strand unit cell with two double octamer ring molecules. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.04 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_7907_msk_1.map emd_7907_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Second half map of RS1 unit
| ファイル | emd_7907_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Second half map of RS1 unit | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: First half map of the RS1 unit cell
| ファイル | emd_7907_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | First half map of the RS1 unit cell | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Retinoschisin
| 全体 | 名称: Retinoschisin |
|---|---|
| 要素 |
|
-超分子 #1: Retinoschisin
| 超分子 | 名称: Retinoschisin / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Human retinoschisin with a honeybee mellitin leader sequence and a C-terminal 6-histidine tag |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 器官: Eye / 組織: Retina / 細胞中の位置: Intercellular space Homo sapiens (ヒト) / 器官: Eye / 組織: Retina / 細胞中の位置: Intercellular space |
| 分子量 | 理論値: 380 KDa |
-分子 #1: Retinoschisin
| 分子 | 名称: Retinoschisin / タイプ: protein_or_peptide / ID: 1 詳細: Mature human RS1 with a C-terminal hexahistidine tag 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  unidentified baculovirus (ウイルス) unidentified baculovirus (ウイルス) |
| 配列 | 文字列: STEDEGEDPW YQKACKCDCQ GGPNALWSAG ATSLDCIPEC PYHKPLGFES GEVTPDQITC SNPEQYVGWY SSWTANKARL NSQGFGCAWL SKFQDSSQWL QIDLKEIKVI SGILTQGRCD IDEWMTKYSV QYRTDERLNW IYYKDQTGNN RVFYGNS DR TSTVQNLLRP ...文字列: STEDEGEDPW YQKACKCDCQ GGPNALWSAG ATSLDCIPEC PYHKPLGFES GEVTPDQITC SNPEQYVGWY SSWTANKARL NSQGFGCAWL SKFQDSSQWL QIDLKEIKVI SGILTQGRCD IDEWMTKYSV QYRTDERLNW IYYKDQTGNN RVFYGNS DR TSTVQNLLRP PIISRFIRLI PLGWHVRIAI RMELLECVSK CAHHHHHH UniProtKB:  Retinoschisin Retinoschisin |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| ||||||||||||
| グリッド | モデル: C-flat-2/2 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: PLASMA CLEANING | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 293 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 3.23 µm / 最小 デフォーカス(補正後): 1.04 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm Bright-field microscopy / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 1-50 / 撮影したグリッド数: 1 / 実像数: 1136 / 平均露光時間: 0.25 sec. / 平均電子線量: 0.66 e/Å2 / 詳細: 30 degree tilted specimen |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z
Z Y
Y X
X