+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4734 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the core Shigella flexneri type III secretion system export gate complex SctRST (Spa24/Spa9/Spa29). Type three secretion system Type three secretion system | ||||||||||||||||||
 マップデータ マップデータ | |||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | ||||||||||||||||||
| 生物種 |   Shigella flexneri (フレクスナー赤痢菌) Shigella flexneri (フレクスナー赤痢菌) | ||||||||||||||||||
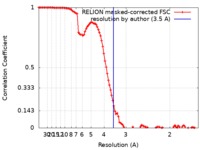
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.5 Å クライオ電子顕微鏡法 / 解像度: 3.5 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Johnson S / Kuhlen L / Deme JC / Abrusci P / Lea SM | ||||||||||||||||||
| 資金援助 |  英国, 5件 英国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2018 ジャーナル: Nat Struct Mol Biol / 年: 2018タイトル: Structure of the core of the type III secretion system export apparatus. 著者: Lucas Kuhlen / Patrizia Abrusci / Steven Johnson / Joseph Gault / Justin Deme / Joseph Caesar / Tobias Dietsche / Mehari Tesfazgi Mebrhatu / Tariq Ganief / Boris Macek / Samuel Wagner / Carol ...著者: Lucas Kuhlen / Patrizia Abrusci / Steven Johnson / Joseph Gault / Justin Deme / Joseph Caesar / Tobias Dietsche / Mehari Tesfazgi Mebrhatu / Tariq Ganief / Boris Macek / Samuel Wagner / Carol V Robinson / Susan M Lea /   要旨: Export of proteins through type III secretion systems is critical for motility and virulence of many major bacterial pathogens. Three putative integral membrane proteins (FliP, FliQ, FliR) are ...Export of proteins through type III secretion systems is critical for motility and virulence of many major bacterial pathogens. Three putative integral membrane proteins (FliP, FliQ, FliR) are suggested to form the core of an export gate in the inner membrane, but their structure, assembly and location within the final nanomachine remain unclear. Here, we present the cryoelectron microscopy structure of the Salmonella Typhimurium FliP-FliQ-FliR complex at 4.2 Å. None of the subunits adopt canonical integral membrane protein topologies, and common helix-turn-helix structural elements allow them to form a helical assembly with 5:4:1 stoichiometry. Fitting of the structure into reconstructions of intact secretion systems, combined with cross-linking, localize the export gate as a core component of the periplasmic portion of the machinery. This study thereby identifies the export gate as a key element of the secretion channel and implies that it primes the helical architecture of the components assembling downstream. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4734.map.gz emd_4734.map.gz | 70.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4734-v30.xml emd-4734-v30.xml emd-4734.xml emd-4734.xml | 24.5 KB 24.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4734_fsc.xml emd_4734_fsc.xml | 10.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4734.png emd_4734.png | 59.8 KB | ||
| マスクデータ |  emd_4734_msk_1.map emd_4734_msk_1.map | 91.1 MB |  マスクマップ マスクマップ | |
| その他 |  emd_4734_additional.map.gz emd_4734_additional.map.gz emd_4734_half_map_1.map.gz emd_4734_half_map_1.map.gz emd_4734_half_map_2.map.gz emd_4734_half_map_2.map.gz | 6.8 MB 71.3 MB 71.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4734 http://ftp.pdbj.org/pub/emdb/structures/EMD-4734 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4734 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4734 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4734.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4734.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.822 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_4734_msk_1.map emd_4734_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
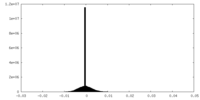
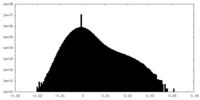
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: #1
| ファイル | emd_4734_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
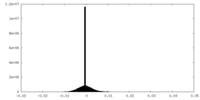
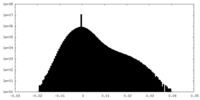
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: None
| ファイル | emd_4734_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | None | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: None
| ファイル | emd_4734_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | None | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Shigella flexneri type III secretion export gate (Spa24Spa9Spa29)
| 全体 | 名称: Shigella flexneri type III secretion export gate (Spa24Spa9Spa29) Type three secretion system Type three secretion system |
|---|---|
| 要素 |
|
-超分子 #1: Shigella flexneri type III secretion export gate (Spa24Spa9Spa29)
| 超分子 | 名称: Shigella flexneri type III secretion export gate (Spa24Spa9Spa29) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Shigella flexneri (フレクスナー赤痢菌) Shigella flexneri (フレクスナー赤痢菌) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) / 組換株: MT56 Escherichia coli (大腸菌) / 組換株: MT56 |
| 分子量 | 実験値: 191 KDa |
-分子 #1: Surface presentation of antigens protein SpaP
| 分子 | 名称: Surface presentation of antigens protein SpaP / タイプ: protein_or_peptide / ID: 1 / コピー数: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Shigella flexneri (フレクスナー赤痢菌) Shigella flexneri (フレクスナー赤痢菌) |
| 分子量 | 理論値: 24.215562 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MLSDMSLIAT LSFFTLLPFL VAAGTCYIKF SIVFVMVRNA LGLQQVPSNM TLNGIALIMA LFVMKPIIEA GYENYLNGPQ KFDTISDIV RFSDSGLMEY KQYLKKHTDL ELARFFQRSE EENADLKSAE NNDYSLFSLL PAYALSEIKD AFKIGFYLYL P FVVVDLVI ...文字列: MLSDMSLIAT LSFFTLLPFL VAAGTCYIKF SIVFVMVRNA LGLQQVPSNM TLNGIALIMA LFVMKPIIEA GYENYLNGPQ KFDTISDIV RFSDSGLMEY KQYLKKHTDL ELARFFQRSE EENADLKSAE NNDYSLFSLL PAYALSEIKD AFKIGFYLYL P FVVVDLVI SSILLALGMM MMSPITISVP IKLVLFVALD GWGILSKALI EQYINIPA |
-分子 #2: Surface presentation of antigens protein SpaR
| 分子 | 名称: Surface presentation of antigens protein SpaR / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Shigella flexneri (フレクスナー赤痢菌) Shigella flexneri (フレクスナー赤痢菌) |
| 分子量 | 理論値: 32.644086 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MDISSWFESI HVFLILLNGV FFRLAPLFFF LPFLNNGIIS PSIRIPVIFL VASGLITSGK VDIGSSVFEH VYFLMFKEII VGLLLSFCL SLPFWIFHAV GSIIDNQRGA TLSSSIDPAN GVDTSELAKF FNLFSAVVFL YSGGMVFILE SIQLSYNICP L FSQCSFRI ...文字列: MDISSWFESI HVFLILLNGV FFRLAPLFFF LPFLNNGIIS PSIRIPVIFL VASGLITSGK VDIGSSVFEH VYFLMFKEII VGLLLSFCL SLPFWIFHAV GSIIDNQRGA TLSSSIDPAN GVDTSELAKF FNLFSAVVFL YSGGMVFILE SIQLSYNICP L FSQCSFRI SNILTFLTLL ASQAVILASP VMIVLLLSEV LLGVLSRFAP QMNAFSVSLT IKSLLAIFII FICSSTIYFS KV QFFLGEH KFFTNLFVRE NLYFQGQFGS WSHPQFEKGG GSGGGSGGGS WSHPQFEK |
-分子 #3: Surface presentation of antigens protein SpaQ
| 分子 | 名称: Surface presentation of antigens protein SpaQ / タイプ: protein_or_peptide / ID: 3 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Shigella flexneri (フレクスナー赤痢菌) Shigella flexneri (フレクスナー赤痢菌) |
| 分子量 | 理論値: 9.433338 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MSDIVYMGNK ALYLILIFSL WPVGIATVIG LSIGLLQTVT QLQEQTLPFG IKLIGVSISL LLLSGWYGEV LLSFCHEIMF LIKSGV |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 8.4 mg/mL | ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||
| グリッド | モデル: Quantifoil, UltrAuFoil, R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE | ||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 実像数: 3741 / 平均露光時間: 8.0 sec. / 平均電子線量: 48.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-6r6b: |
 ムービー
ムービー コントローラー
コントローラー















 Z
Z Y
Y X
X