+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM Structure of the Helicobacter pylori dcagM PR | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード |  T4SS / T4SS /  protein translocation / protein translocation /  MEMBRANE PROTEIN (膜タンパク質) MEMBRANE PROTEIN (膜タンパク質) | ||||||||||||
| 生物種 |   Helicobacter pylori 26695 (ピロリ菌) Helicobacter pylori 26695 (ピロリ菌) | ||||||||||||
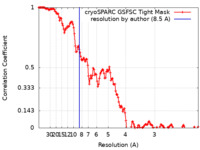
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 8.5 Å クライオ電子顕微鏡法 / 解像度: 8.5 Å | ||||||||||||
 データ登録者 データ登録者 | Roberts JR | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Life Sci Alliance / 年: 2024 ジャーナル: Life Sci Alliance / 年: 2024タイトル: Subdomains of the Cag T4SS outer membrane core complex exhibit structural independence. 著者: Jacquelyn R Roberts / Sirena C Tran / Arwen E Frick-Cheng / Kaeli N Bryant / Chiamaka D Okoye / W Hayes McDonald / Timothy L Cover / Melanie D Ohi /  要旨: The Cag type IV secretion system (Cag T4SS) has an important role in the pathogenesis of gastric cancer. The Cag T4SS outer membrane core complex (OMCC) is organized into three regions: a 14-fold ...The Cag type IV secretion system (Cag T4SS) has an important role in the pathogenesis of gastric cancer. The Cag T4SS outer membrane core complex (OMCC) is organized into three regions: a 14-fold symmetric outer membrane cap (OMC) composed of CagY, CagX, CagT, CagM, and Cag3; a 17-fold symmetric periplasmic ring (PR) composed of CagY and CagX; and a stalk with unknown composition. We investigated how CagT, CagM, and a conserved antenna projection (AP) region of CagY contribute to the structural organization of the OMCC. Single-particle cryo-EM analyses showed that complexes purified from Δ or Δ mutants no longer had organized OMCs, but the PRs remained structured. OMCCs purified from a CagY antenna projection mutant (CagYAP) were structurally similar to WT OMCCs, except for the absence of the α-helical antenna projection. These results indicate that CagY and CagX are sufficient for maintaining a stable PR, but the organization of the OMC requires CagY, CagX, CagM, and CagT. Our results highlight an unexpected structural independence of two major subdomains of the Cag T4SS OMCC. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_42393.map.gz emd_42393.map.gz | 258.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-42393-v30.xml emd-42393-v30.xml emd-42393.xml emd-42393.xml | 17.3 KB 17.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_42393_fsc.xml emd_42393_fsc.xml | 13.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_42393.png emd_42393.png | 100.3 KB | ||
| Filedesc metadata |  emd-42393.cif.gz emd-42393.cif.gz | 5.9 KB | ||
| その他 |  emd_42393_half_map_1.map.gz emd_42393_half_map_1.map.gz emd_42393_half_map_2.map.gz emd_42393_half_map_2.map.gz | 254.5 MB 254.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-42393 http://ftp.pdbj.org/pub/emdb/structures/EMD-42393 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42393 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42393 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_42393.map.gz / 形式: CCP4 / 大きさ: 274.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_42393.map.gz / 形式: CCP4 / 大きさ: 274.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_42393_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_42393_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Helicobacter pylori dcagM PR
| 全体 | 名称: Helicobacter pylori dcagM PR |
|---|---|
| 要素 |
|
-超分子 #1: Helicobacter pylori dcagM PR
| 超分子 | 名称: Helicobacter pylori dcagM PR / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Helicobacter pylori 26695 (ピロリ菌) / 株: 26695 / 細胞中の位置: membrane Helicobacter pylori 26695 (ピロリ菌) / 株: 26695 / 細胞中の位置: membrane |
-分子 #1: Cag pathogenicity island protein (Cag8)
| 分子 | 名称: Cag pathogenicity island protein (Cag8) / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Helicobacter pylori 26695 (ピロリ菌) Helicobacter pylori 26695 (ピロリ菌) |
| 配列 | 文字列: MGQAFFKKIV GCFCLGYLF L SSAIEAAA LD IKNFNRG RVK VVNKKI AYLG DEKPI TIWTS LDNV TVIQLE KDE TISYITT GF NKGWSIVP N SNHIFIQPK SVKSNLMFEK EAVNFALMT R DYQEFLKT KK LIVDAPD PKE LEEQKK ALEK EKEAK ...文字列: MGQAFFKKIV GCFCLGYLF L SSAIEAAA LD IKNFNRG RVK VVNKKI AYLG DEKPI TIWTS LDNV TVIQLE KDE TISYITT GF NKGWSIVP N SNHIFIQPK SVKSNLMFEK EAVNFALMT R DYQEFLKT KK LIVDAPD PKE LEEQKK ALEK EKEAK EQAQK AQKD KREKRK EER AKNRANL EN LTNAMSNP Q NLSNNKNLS EFIKQQRENE LDQMERLED M QEQAQANA LK QIEELNK KQA EETIKQ RAKD KINIK TDKPQ KSPE DNSIEL SPS DSAWRTN LV VRTNKALY Q FILRIAQKD NFASAYLTVK LEYPQRHEV S SVIEELKK RE EAKRQKE LIK QENLNT TAYI NRVMM ASNEQ IINK EKIREE KQK IILDQAK AL ETQYVHNA L KRNPVPRNY NYYQAPEKRS KHIMPSEIF D DGTFTYFG FK NITLQPA IFV VQPDGK LSMT DAAID PNMTN SGLR WYRVNE IAE KFKLIKD KA LVTVINKG Y GKNPLTKNY NIKNYGELER VIKKLPEVR D K |
-分子 #2: Cag pathogenicity island protein (Cag7)
| 分子 | 名称: Cag pathogenicity island protein (Cag7) / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Helicobacter pylori 26695 (ピロリ菌) Helicobacter pylori 26695 (ピロリ菌) |
| 配列 | 文字列: MNEENDKLET SKKAQQDSP Q DLSNEEAT EA NHFENLL KES KESSDH HLDN PTETQ THFDG DKSE ETQTQM DSE GNETSES SN GSLADKLF K KARKLVDNK KPFTQQKNLD EETQELNEE D DQENNEYQ EE TQTDLID DET SKKTQQ HSPQ DLSNE ...文字列: MNEENDKLET SKKAQQDSP Q DLSNEEAT EA NHFENLL KES KESSDH HLDN PTETQ THFDG DKSE ETQTQM DSE GNETSES SN GSLADKLF K KARKLVDNK KPFTQQKNLD EETQELNEE D DQENNEYQ EE TQTDLID DET SKKTQQ HSPQ DLSNE EATEA NHFE NLLKES KES SDHHLDN PT ETQTNFDG D KSEETQTQM DSEGNETSES SNGSLADKL F KKARKLVD NK KPFTQQK NLD EETQEL NEED DQENN EYQEE TQTD LIDDET SKK TQQHSPQ DL SNEEATEA N HFENLLKES KESSDHHLDN PTETQTNFD G DKSEEITD DS NDQEIIK GSK KKYIIG GIVV AVLIV IILFS RSIF HYFMPL EDK SSRFSKD RN LYVNDEIQ I RQEYNRLLK ERNEKGNMID KNLFFNDDP N RTLYNYLN IA EIEDKNP LRA FYECIS NGGN YEECL KLIKD KKLQ DQMKKT LEA YNDCIKN AK TEEERIKC L DLIKDENLK KSLLNQQKVQ VALDCLKNA K TDEERNEC LK LINDPEI REK FRKELE LQKE LQEYK DCIKN AKTE AEKNKC LKG LSKEAIE RL KQQALDCL K NAKTDEERN ECLKNIPQDL QKELLADMS V KAYKDCVS KA RNEKEKQ ECE KLLTPE ARKK LEQQV LDCLK NAKT DEERKK CLK DLPKDLQ SD ILAKESLK A YKDCVSQAK TEAEKKECEK LLTPEAKKL L EEEAKESV KA YLDCVSQ AKT EAEKKE CEKL LTPEA KKKLE EAKK SVKAYL DCV SRARNEK EK KECEKLLT P EAKKLLEQQ ALDCLKNAKT DKERKKCLK D LPKDLQKK VL AKESVKA YLD CVSQAK TEAE KKECE KLLTP EARK LLEEAK KSV KAYLDCV SQ AKTEAEKK E CEKLLTPEA RKLLEEXAKE SVKAYLDCV S QAKNEAEK KE CEKLLTL ESK KKLEEA KKSV KAYLD CVSQA KTEA EKKECE KLL TPEAKKL LE QQALDCLK N AKTEADKKR CVKDLPKDLQ KKVLAKESL K AYKDCVSK AR NEKEKKE CEK LLTPEA KKLL EEAKK SVKAY LDCV SQAKTE AEK KECEKLL TP EARKLLEE A KESVKAYKD CVSKARNEKE KKECEKLLT P EAKKLLEQ QV LDCLKNA KTE ADKKRC VKDL PKDLQ KKVLA KESV KAYLDC VSR ARNEKEK KE CEKLLTPE A KKLLEEAKE SLKAYKDCLS QARNEEERR A CEKLLTPE AR KLLEQEV KKS IKAYLD CVSR ARNEK EKKEC EKLL TPEARK FLA KQVLNCL EK AGNEEERK A CLKNLPKDL QENILAKESL KAYKDCLSQ A RNEEERRA CE KLLTPEA RKL LEQEVK KSVK AYLDC VSRAR NEKE KKECEK LLT PEARKFL AK ELQQKDKA I KDCLKNADP NDRAAIMKCL DGLSDEEKL K YLQEAREK AV ADCLAMA KTD EEKRKC QNLY SDLIQ EIQNK RTQN KQNQLS KTE RLHQASE CL DNLDDPTD Q EAIEQCLEG LSDSERALIL GIKRQADEV D LIYSDLRN RK TFDNMAA KGY PLLPMD FKNG GDIAT INATN VDAD KIASDN PIY ASIEPDI AK QYETEKTI K DKNLEAKLA KALGGNKKDD DKEKSKKST A EAKAENNK ID KDVAETA KNI SEIALK NKKE KSGEF VDENG NPID DKKKAE KQD ETSPVKQ AF IGKSDPTF V LAQYTPIEI TLTSKVDATL TGIVSGVVA K DVWNMNGT MI LLDKGTK VYG NYQSVK GGTP IMTRL MIVFT KAIT PDGVII PLA NAQAAGM LG EAGVDGYV N NHFMKRIGF AVIASVVNSF LQTAPIIAL D KLIGLGKG RS ERTPEFN YAL GQAING SMQS SAQMS NQILG QLMN IPPSFY KNE GDSIKIL TM DDIDFSGV Y DVKITNKSV VDEIIKQSTK TLSREHEEI T TSPKGGN |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.4 mg/mL |
|---|---|
| 緩衝液 | pH: 7 |
| 凍結 | 凍結剤: NITROGEN |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 60.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 Z
Z Y
Y X
X