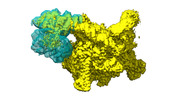
登録情報 データベース : EMDB / ID : EMD-41856タイトル Cryo-EM structure of E. coli NarL-transcription activation complex at 3.2A E coli NarL-TAC unsharpened map 複合体 : E. coli NarL transcription activation complex on the yeaR promoterタンパク質・ペプチド : x 6種DNA : x 2種RNA : x 1種リガンド : x 2種 / / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Escherichia coli (大腸菌)手法 / / 解像度 : 3.23 Å Liu B / Kompaniiets D / Wang D 資金援助 1件 Organization Grant number 国 Not funded
ジャーナル : Nucleic Acids Res / 年 : 2024タイトル : Structural basis for transcription activation by the nitrate-responsive regulator NarL.著者 : Dmytro Kompaniiets / Lina He / Dong Wang / Wei Zhou / Yang Yang / Yangbo Hu / Bin Liu / 要旨 : Transcription activation is a crucial step of regulation during transcription initiation and a classic check point in response to different stimuli and stress factors. The Escherichia coli NarL is a ... Transcription activation is a crucial step of regulation during transcription initiation and a classic check point in response to different stimuli and stress factors. The Escherichia coli NarL is a nitrate-responsive global transcription factor that controls the expression of nearly 100 genes. However, the molecular mechanism of NarL-mediated transcription activation is not well defined. Here we present a cryo-EM structure of NarL-dependent transcription activation complex (TAC) assembled on the yeaR promoter at 3.2 Å resolution. Our structure shows that the NarL dimer binds at the -43.5 site of the promoter DNA with its C-terminal domain (CTD) not only binding to the DNA but also making interactions with RNA polymerase subunit alpha CTD (αCTD). The key role of these NarL-mediated interactions in transcription activation was further confirmed by in vivo and in vitro transcription assays. Additionally, the NarL dimer binds DNA in a different plane from that observed in the structure of class II TACs. Unlike the canonical class II activation mechanism, NarL does not interact with σ4, while RNAP αCTD is bound to DNA on the opposite side of NarL. Our findings provide a structural basis for detailed mechanistic understanding of NarL-dependent transcription activation on yeaR promoter and reveal a potentially novel mechanism of transcription activation. 履歴 登録 2023年9月7日 - ヘッダ(付随情報) 公開 2024年1月17日 - マップ公開 2024年1月17日 - 更新 2024年2月21日 - 現状 2024年2月21日 処理サイト : RCSB / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報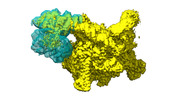
 マップデータ
マップデータ 試料
試料 キーワード
キーワード transcription (転写 (生物学)) /
transcription (転写 (生物学)) /  RNA polymerase (RNAポリメラーゼ) / transcription activation / NarL / nitrate-responsive regulator / transcription-DNA complex
RNA polymerase (RNAポリメラーゼ) / transcription activation / NarL / nitrate-responsive regulator / transcription-DNA complex 機能・相同性情報
機能・相同性情報 RNA polymerase complex / submerged biofilm formation / cellular response to cell envelope stress / cytosolic DNA-directed RNA polymerase complex / regulation of DNA-templated transcription initiation / bacterial-type flagellum assembly /
RNA polymerase complex / submerged biofilm formation / cellular response to cell envelope stress / cytosolic DNA-directed RNA polymerase complex / regulation of DNA-templated transcription initiation / bacterial-type flagellum assembly /  sigma factor activity / phosphorelay signal transduction system ...sigma factor antagonist complex / DNA-binding transcription repressor activity /
sigma factor activity / phosphorelay signal transduction system ...sigma factor antagonist complex / DNA-binding transcription repressor activity /  RNA polymerase complex / submerged biofilm formation / cellular response to cell envelope stress / cytosolic DNA-directed RNA polymerase complex / regulation of DNA-templated transcription initiation / bacterial-type flagellum assembly /
RNA polymerase complex / submerged biofilm formation / cellular response to cell envelope stress / cytosolic DNA-directed RNA polymerase complex / regulation of DNA-templated transcription initiation / bacterial-type flagellum assembly /  sigma factor activity / phosphorelay signal transduction system / bacterial-type flagellum-dependent cell motility / nitrate assimilation / transcription elongation factor complex / regulation of DNA-templated transcription elongation / transcription antitermination /
sigma factor activity / phosphorelay signal transduction system / bacterial-type flagellum-dependent cell motility / nitrate assimilation / transcription elongation factor complex / regulation of DNA-templated transcription elongation / transcription antitermination /  運動性 /
運動性 /  デオキシリボ核酸 / DNA-templated transcription initiation /
デオキシリボ核酸 / DNA-templated transcription initiation /  ribonucleoside binding / DNA-directed 5'-3' RNA polymerase activity /
ribonucleoside binding / DNA-directed 5'-3' RNA polymerase activity /  ポリメラーゼ / response to heat / protein-containing complex assembly / intracellular iron ion homeostasis /
ポリメラーゼ / response to heat / protein-containing complex assembly / intracellular iron ion homeostasis /  protein dimerization activity / transcription cis-regulatory region binding / response to antibiotic / negative regulation of DNA-templated transcription / regulation of DNA-templated transcription / magnesium ion binding /
protein dimerization activity / transcription cis-regulatory region binding / response to antibiotic / negative regulation of DNA-templated transcription / regulation of DNA-templated transcription / magnesium ion binding /  DNA binding / zinc ion binding /
DNA binding / zinc ion binding /  ATP binding /
ATP binding /  生体膜 / identical protein binding /
生体膜 / identical protein binding /  細胞質基質 /
細胞質基質 /  細胞質
細胞質
 Escherichia coli (大腸菌)
Escherichia coli (大腸菌) 単粒子再構成法 /
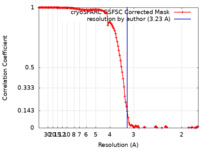
単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.23 Å
クライオ電子顕微鏡法 / 解像度: 3.23 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Nucleic Acids Res / 年: 2024
ジャーナル: Nucleic Acids Res / 年: 2024

 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_41856.map.gz
emd_41856.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-41856-v30.xml
emd-41856-v30.xml emd-41856.xml
emd-41856.xml EMDBヘッダ
EMDBヘッダ emd_41856_fsc.xml
emd_41856_fsc.xml FSCデータファイル
FSCデータファイル emd_41856.png
emd_41856.png emd-41856.cif.gz
emd-41856.cif.gz emd_41856_additional_1.map.gz
emd_41856_additional_1.map.gz emd_41856_additional_2.map.gz
emd_41856_additional_2.map.gz emd_41856_half_map_1.map.gz
emd_41856_half_map_1.map.gz emd_41856_half_map_2.map.gz
emd_41856_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-41856
http://ftp.pdbj.org/pub/emdb/structures/EMD-41856 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41856
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41856
 F&H 検索
F&H 検索 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_41856.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_41856.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 クライオ電子顕微鏡法
クライオ電子顕微鏡法 解析
解析 単粒子再構成法
単粒子再構成法 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000
Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 
 画像解析
画像解析
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X