+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
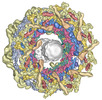
| タイトル | Double nuclear outer ring of Nup84-complexes from the yeast NPC | |||||||||||||||
 マップデータ マップデータ | full c8 ring of double Y-complexes from multibody volume | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード |  nuclear pore complex (核膜孔) / nucleocytoplasmic transport / nuclear pore complex (核膜孔) / nucleocytoplasmic transport /  nucleoporin (ヌクレオポリン) / nucleoporin (ヌクレオポリン) /  membrane protein (膜タンパク質) / membrane protein (膜タンパク質) /  translocase (輸送酵素) / translocase (輸送酵素) /  TRANSPORT PROTEIN (運搬体タンパク質) TRANSPORT PROTEIN (運搬体タンパク質) | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報mRNA export from nucleus in response to heat stress / Seh1-associated complex / positive regulation of ER to Golgi vesicle-mediated transport / protein exit from endoplasmic reticulum / COPII-coated vesicle budding / COPII-mediated vesicle transport / nuclear pore localization / nuclear pore central transport channel / telomere tethering at nuclear periphery / regulation of TORC1 signaling ...mRNA export from nucleus in response to heat stress / Seh1-associated complex / positive regulation of ER to Golgi vesicle-mediated transport / protein exit from endoplasmic reticulum / COPII-coated vesicle budding / COPII-mediated vesicle transport / nuclear pore localization / nuclear pore central transport channel / telomere tethering at nuclear periphery / regulation of TORC1 signaling / nuclear pore outer ring / COPII vesicle coat / post-transcriptional tethering of RNA polymerase II gene DNA at nuclear periphery / positive regulation of protein exit from endoplasmic reticulum / structural constituent of nuclear pore / vacuolar membrane / silent mating-type cassette heterochromatin formation / nucleocytoplasmic transport / subtelomeric heterochromatin formation /  ribosomal large subunit export from nucleus / positive regulation of TOR signaling / mRNA transport / mRNA export from nucleus / ribosomal large subunit export from nucleus / positive regulation of TOR signaling / mRNA transport / mRNA export from nucleus /  核膜孔 / 核膜孔 /  : / positive regulation of TORC1 signaling / protein export from nucleus / cell periphery / protein import into nucleus / double-strand break repair / : / positive regulation of TORC1 signaling / protein export from nucleus / cell periphery / protein import into nucleus / double-strand break repair /  核膜 / 核膜 /  核膜 / 核膜 /  chromosome, telomeric region / endoplasmic reticulum membrane / structural molecule activity / positive regulation of DNA-templated transcription / negative regulation of transcription by RNA polymerase II / chromosome, telomeric region / endoplasmic reticulum membrane / structural molecule activity / positive regulation of DNA-templated transcription / negative regulation of transcription by RNA polymerase II /  小胞体 / positive regulation of transcription by RNA polymerase II / identical protein binding 小胞体 / positive regulation of transcription by RNA polymerase II / identical protein binding類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) | |||||||||||||||
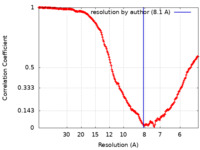
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 8.1 Å クライオ電子顕微鏡法 / 解像度: 8.1 Å | |||||||||||||||
 データ登録者 データ登録者 | Akey CW / Echeverria I / Ouch C / Fernandez-Martinez J / Rout MP | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2023 ジャーナル: Mol Cell / 年: 2023タイトル: Implications of a multiscale structure of the yeast nuclear pore complex. 著者: Christopher W Akey / Ignacia Echeverria / Christna Ouch / Ilona Nudelman / Yi Shi / Junjie Wang / Brian T Chait / Andrej Sali / Javier Fernandez-Martinez / Michael P Rout /   要旨: Nuclear pore complexes (NPCs) direct the nucleocytoplasmic transport of macromolecules. Here, we provide a composite multiscale structure of the yeast NPC, based on improved 3D density maps from ...Nuclear pore complexes (NPCs) direct the nucleocytoplasmic transport of macromolecules. Here, we provide a composite multiscale structure of the yeast NPC, based on improved 3D density maps from cryogenic electron microscopy and AlphaFold2 models. Key features of the inner and outer rings were integrated into a comprehensive model. We resolved flexible connectors that tie together the core scaffold, along with equatorial transmembrane complexes and a lumenal ring that anchor this channel within the pore membrane. The organization of the nuclear double outer ring reveals an architecture that may be shared with ancestral NPCs. Additional connections between the core scaffold and the central transporter suggest that under certain conditions, a degree of local organization is present at the periphery of the transport machinery. These connectors may couple conformational changes in the scaffold to the central transporter to modulate transport. Collectively, this analysis provides insights into assembly, transport, and NPC evolution. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41285.map.gz emd_41285.map.gz | 9.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41285-v30.xml emd-41285-v30.xml emd-41285.xml emd-41285.xml | 43 KB 43 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_41285_fsc.xml emd_41285_fsc.xml | 21.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_41285.png emd_41285.png | 160.3 KB | ||
| Filedesc metadata |  emd-41285.cif.gz emd-41285.cif.gz | 11.1 KB | ||
| その他 |  emd_41285_additional_1.map.gz emd_41285_additional_1.map.gz emd_41285_additional_2.map.gz emd_41285_additional_2.map.gz emd_41285_additional_3.map.gz emd_41285_additional_3.map.gz emd_41285_additional_4.map.gz emd_41285_additional_4.map.gz emd_41285_half_map_1.map.gz emd_41285_half_map_1.map.gz emd_41285_half_map_2.map.gz emd_41285_half_map_2.map.gz | 5.6 MB 1 MB 5.6 MB 96.2 MB 31.9 MB 31.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41285 http://ftp.pdbj.org/pub/emdb/structures/EMD-41285 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41285 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41285 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8tieMC  8t9lC  8tj5C C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41285.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41285.map.gz / 形式: CCP4 / 大きさ: 421.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | full c8 ring of double Y-complexes from multibody volume | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.66 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: multibody half map
| ファイル | emd_41285_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | multibody half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
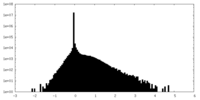
| 密度ヒストグラム |
-追加マップ: recombined 2 Y-complex protomer, bfactor -600 and local...
| ファイル | emd_41285_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | recombined 2 Y-complex protomer, bfactor -600 and local resolution box 120 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: multibody half map
| ファイル | emd_41285_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | multibody half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: post processed multibody volume
| ファイル | emd_41285_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | post processed multibody volume | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map of c8 ring
| ファイル | emd_41285_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map of c8 ring | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
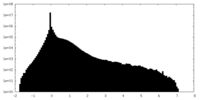
| 密度ヒストグラム |
-ハーフマップ: half map of c8 ring
| ファイル | emd_41285_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map of c8 ring | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Nuclear Pore Complex
| 全体 | 名称: Nuclear Pore Complex 核膜孔 核膜孔 |
|---|---|
| 要素 |
|
-超分子 #1: Nuclear Pore Complex
| 超分子 | 名称: Nuclear Pore Complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Protein A tagged Mlp1 pullout of NPC |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母)株: MATa ade2-1 ura3-1 his3-11,15 trp1-1 leu2-3,112 can1-100 MLP1-PPX-ProteinA::HIS5 Organelle: nucleus / 細胞中の位置: nuclear envelope |
-超分子 #2: double Nup84 Y-complex
| 超分子 | 名称: double Nup84 Y-complex / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: all / 詳細: Nup84 Y-complexes in nuclear double outer ring |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母)株: MATa ade2-1 ura3-1 his3-11,15 trp1-1 leu2-3,112 can1-100 MLP1-PPX-ProteinA::HIS5 Organelle: nucleus / 細胞中の位置: nuclear envelope |
-分子 #1: Nucleoporin NUP120
| 分子 | 名称: Nucleoporin NUP120 / タイプ: protein_or_peptide / ID: 1 / 詳細: long arm of Nup84 Y-complex / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) / 株: MATa ade2-1 ura3-1 his3-11 Saccharomyces cerevisiae (パン酵母) / 株: MATa ade2-1 ura3-1 his3-11 |
| 分子量 | 理論値: 120.560328 KDa |
| 配列 | 文字列: MACLSRIDAN LLQYYEKPEP NNTVDLYVSN NSNNNGLKEG DKSISTPVPQ PYGSEYSNCL LLSNSEYICY HFSSRSTLLT FYPLSDAYH GKTINIHLPN ASMNQRYTLT IQEVEQQLLV NVILKDGSFL TLQLPLSFLF SSANTLNGEW FHLQNPYDFT V RVPHFLFY ...文字列: MACLSRIDAN LLQYYEKPEP NNTVDLYVSN NSNNNGLKEG DKSISTPVPQ PYGSEYSNCL LLSNSEYICY HFSSRSTLLT FYPLSDAYH GKTINIHLPN ASMNQRYTLT IQEVEQQLLV NVILKDGSFL TLQLPLSFLF SSANTLNGEW FHLQNPYDFT V RVPHFLFY VSPQFSVVFL EDGGLLGLKK VDGVHYEPLL FNDNSYLKSL TRFFSRSSKS DYDSVISCKL FHERYLIVLT QN CHLKIWD LTSFTLIQDY DMVSQSDSDP SHFRKVEAVG EYLSLYNNTL VTLLPLENGL FQMGTLLVDS SGILTYTFQN NIP TNLSAS AIWSIVDLVL TRPLELNVEA SYLNLIVLWK SGTASKLQIL NVNDESFKNY EWIESVNKSL VDLQSEHDLD IVTK TGDVE RGFCNLKSRY GTQIFERAQQ ILSENKIIMA HNEDEEYLAN LETILRDVKT AFNEASSITL YGDEIILVNC FQPYN HSLY KLNTTVENWF YNMHSETDGS ELFKYLRTLN GFASTLSNDV LRSISKKFLD IITGELPDSM TTVEKFTDIF KNCLEN QFE ITNLKILFDE LNSFDIPVVL NDLINNQMKP GIFWKKDFIS AIKFDGFTSI ISLESLHQLL SIHYRITLQV LLTFVLF DL DTEIFGQHIS TLLDLHYKQF LLLNLYRQDK CLLAEVLLKD SSEFSFGVKF FNYGQLIAYI DSLNSNVYNA SITENSFF M TFFRSYIIEN TSHKNIRFFL ENVECPFYLR HNEVQEFMFA MTLFSCGNFD QSYEIFQLHD YPEAINDKLP TFLEDLKSE NYHGDSIWKD LLCTFTVPYR HSAFYYQLSL LFDRNNSQEF ALKCISKSAE YSLKEIQIEE LQDFKEKQHI HYLNLLIHFR MFEEVLDVL RLGHECLSDT VRTNFLQLLL QEDIYSRDFF STLLRLCNAH SDNGELYLRT VDIKIVDSIL SQNLRSGDWE C FKKLYCFR MLNKSERAAA EVLYQYILMQ ADLDVIRKRK CYLMVINVLS SFDSAYDQWI LNGSKVVTLT DLRDELRGL UniProtKB: Nucleoporin NUP120 |
-分子 #2: Nucleoporin NUP85
| 分子 | 名称: Nucleoporin NUP85 / タイプ: protein_or_peptide / ID: 2 / 詳細: short arm of Nup84 Y-complex / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 分子量 | 理論値: 84.972438 KDa |
| 配列 | 文字列: MTIDDSNRLL MDVDQFDFLD DGTAQLSNNK TDEEEQLYKR DPVSGAILVP MTVNDQPIEK NGDKMPLKFK LGPLSYQNMA FITAKDKYK LYPVRIPRLD TSKEFSAYVS GLFEIYRDLG DDRVFNVPTI GVVNSNFAKE HNATVNLAME AILNELEVFI G RVKDQDGR ...文字列: MTIDDSNRLL MDVDQFDFLD DGTAQLSNNK TDEEEQLYKR DPVSGAILVP MTVNDQPIEK NGDKMPLKFK LGPLSYQNMA FITAKDKYK LYPVRIPRLD TSKEFSAYVS GLFEIYRDLG DDRVFNVPTI GVVNSNFAKE HNATVNLAME AILNELEVFI G RVKDQDGR VNRFYELEES LTVLNCLRTM YFILDGQDVE ENRSEFIESL LNWINRSDGE PDEEYIEQVF SVKDSTAGKK VF ETQYFWK LLNQLVLRGL LSQAIGCIER SDLLPYLSDT CAVSFDAVSD SIELLKQYPK DSSSTFREWK NLVLKLSQAF GSS ATDISG ELRDYIEDFL LVIGGNQRKI LQYSRTWYES FCGFLLYYIP SLELSAEYLQ MSLEANVVDI TNDWEQPCVD IISG KIHSI LPVMESLDSC TAAFTAMICE AKGLIENIFE GEKNSDDYSN EDNEMLEDLF SYRNGMASYM LNSFAFELCS LGDKE LWPV AIGLIALSAT GTRSAKKMVI AELLPHYPFV TNDDIEWMLS ICVEWRLPEI AKEIYTTLGN QMLSAHNIIE SIANFS RAG KYELVKSYSW LLFEASCMEG QKLDDPVLNA IVSKNSPAED DVIIPQDILD CVVTNSMRQT LAPYAVLSQF YELRDRE DW GQALRLLLLL IEFPYLPKHY LVLLVAKFLY PIFLLDDKKL MDEDSVATVI EVIETKWDDA DEKSSNLYET IIEADKSL P SSMATLLKNL RKKLNFKLCQ AFM UniProtKB: Nucleoporin NUP85 |
-分子 #3: NUP145 isoform 1
| 分子 | 名称: NUP145 isoform 1 / タイプ: protein_or_peptide / ID: 3 / 詳細: stem junction of Nup84 Y-complex / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 分子量 | 理論値: 145.810578 KDa |
| 配列 | 文字列: MFNKSVNSGF TFGNQNTSTP TSTPAQPSSS LQFPQKSTGL FGNVNVNANT STPSPSGGLF NANSNANSIS QQPANNSLFG NKPAQPSGG LFGATNNTTS KSAGSLFGNN NATANSTGST GLFSGSNNIA SSTQNGGLFG NSNNNNITST TQNGGLFGKP T TTPAGAGG ...文字列: MFNKSVNSGF TFGNQNTSTP TSTPAQPSSS LQFPQKSTGL FGNVNVNANT STPSPSGGLF NANSNANSIS QQPANNSLFG NKPAQPSGG LFGATNNTTS KSAGSLFGNN NATANSTGST GLFSGSNNIA SSTQNGGLFG NSNNNNITST TQNGGLFGKP T TTPAGAGG LFGNSSSTNS TTGLFGSNNT QSSTGIFGQK PGASTTGGLF GNNGASFPRS GETTGTMSTN PYGINISNVP MA VADMPRS ITSSLSDVNG KSDAEPKPIE NRRTYSFSSS VSGNAPLPLA SQSSLVSRLS TRLKATQKST SPNEIFSPSY SKP WLNGAG SAPLVDDFFS SKMTSLAPNE NSIFPQNGFN FLSSQRADLT ELRKLKIDSN RSAAKKLKLL SGTPAITKKH MQDE QDSSE NEPIANADSV TNIDRKENRD NNLDNTYLNG KEQSNNLNKQ DGENTLQHEK SSSFGYWCSP SPEQLERLSL KQLAA VSNF VIGRRGYGCI TFQHDVDLTA FTKSFREELF GKIVIFRSSK TVEVYPDEAT KPMIGHGLNV PAIITLENVY PVDKKT KKP MKDTTKFAEF QVFDRKLRSM REMNYISYNP FGGTWTFKVN HFSIWGLVNE EDAEIDEDDL SKQEDGGEQP LRKVRTL AQ SKPSDKEVIL KTDGTFGTLS GKDDSIVEEK AYEPDLSDAD FEGIEASPKL DVSKDWVEQL ILAGSSLRSV FATSKEFD G PCQNEIDLLF SECNDEIDNA KLIMKERRFT ASYTFAKFST GSMLLTKDIV GKSGVSIKRL PTELQRKFLF DDVYLDKEI EKVTIEARKS NPYPQISESS LLFKDALDYM EKTSSDYNLW KLSSILFDPV SYPYKTDNDQ VKMALLKKER HCRLTSWIVS QIGPEIEEK IRNSSNEIEQ IFLYLLLNDV VRASKLAIES KNGHLSVLIS YLGSNDPRIR DLAELQLQKW STGGCSIDKN I SKIYKLLS GSPFEGLFSL KELESEFSWL CLLNLTLCYG QIDEYSLESL VQSHLDKFSL PYDDPIGVIF QLYAANENTE KL YKEVRQR TNALDVQFCW YLIQTLRFNG TRVFSKETSD EATFAFAAQL EFAQLHGHSL FVSCFLNDDK AAEDTIKRLV MRE ITLLRA STNDHILNRL KIPSQLIFNA QALKDRYEGN YLSEVQNLLL GSSYDLAEMA IVTSLGPRLL LSNNPVQNNE LKTL REILN EFPDSERDKW SVSINVFEVY LKLVLDNVET QETIDSLISG MKIFYDQYKH CREVAACCNV MSQEIVSKIL EKNNP SIGD SKAKLLELPL GQPEKAYLRG EFAQDLMKCT YKI UniProtKB: NUP145 isoform 1 |
-分子 #4: Protein transport protein SEC13
| 分子 | 名称: Protein transport protein SEC13 / タイプ: protein_or_peptide / ID: 4 / 詳細: cofactor of Nup145C on stem of Nup84 Y-complex / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 分子量 | 理論値: 33.082965 KDa |
| 配列 | 文字列: MVVIANAHNE LIHDAVLDYY GKRLATCSSD KTIKIFEVEG ETHKLIDTLT GHEGPVWRVD WAHPKFGTIL ASCSYDGKVL IWKEENGRW SQIAVHAVHS ASVNSVQWAP HEYGPLLLVA SSDGKVSVVE FKENGTTSPI IIDAHAIGVN SASWAPATIE E DGEHNGTK ...文字列: MVVIANAHNE LIHDAVLDYY GKRLATCSSD KTIKIFEVEG ETHKLIDTLT GHEGPVWRVD WAHPKFGTIL ASCSYDGKVL IWKEENGRW SQIAVHAVHS ASVNSVQWAP HEYGPLLLVA SSDGKVSVVE FKENGTTSPI IIDAHAIGVN SASWAPATIE E DGEHNGTK ESRKFVTGGA DNLVKIWKYN SDAQTYVLES TLEGHSDWVR DVAWSPTVLL RSYLASVSQD RTCIIWTQDN EQ GPWKKTL LKEEKFPDVL WRASWSLSGN VLALSGGDNK VTLWKENLEG KWEPAGEVHQ UniProtKB:  Protein transport protein SEC13 Protein transport protein SEC13 |
-分子 #5: Nucleoporin Seh1
| 分子 | 名称: Nucleoporin Seh1 / タイプ: protein_or_peptide / ID: 5 / 詳細: cofactor Nup85 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 分子量 | 理論値: 34.652078 KDa |
| 配列 | 文字列: MQPFDSGHDD LVHDVVYDFY GRHVATCSSD QHIKVFKLDK DTSNWELSDS WRAHDSSIVA IDWASPEYGR IIASASYDKT VKLWEEDPD QEECSGRRWN KLCTLNDSKG SLYSVKFAPA HLGLKLACLG NDGILRLYDA LEPSDLRSWT LTSEMKVLSI P PANHLQSD ...文字列: MQPFDSGHDD LVHDVVYDFY GRHVATCSSD QHIKVFKLDK DTSNWELSDS WRAHDSSIVA IDWASPEYGR IIASASYDKT VKLWEEDPD QEECSGRRWN KLCTLNDSKG SLYSVKFAPA HLGLKLACLG NDGILRLYDA LEPSDLRSWT LTSEMKVLSI P PANHLQSD FCLSWCPSRF SPEKLAVSAL EQAIIYQRGK DGKLHVAAKL PGHKSLIRSI SWAPSIGRWY QLIATGCKDG RI RIFKITE KNLQVELLSE HDDHNGEVWS VSWNLTGTIL SSAGDDGKVR LWKATYSNEF KCMSVITAQQ |
-分子 #6: Nucleoporin NUP84
| 分子 | 名称: Nucleoporin NUP84 / タイプ: protein_or_peptide / ID: 6 / 詳細: tail of Nup84 Y-complex / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 分子量 | 理論値: 83.718867 KDa |
| 配列 | 文字列: MELSPTYQTE RFTKFSDTLK EFKIEQNNEQ NPIDPFNIIR EFRSAAGQLA LDLANSGDES NVISSKDWEL EARFWHLVEL LLVFRNADL DLDEMELHPY NSRGLFEKKL MQDNKQLYQI WIVMVWLKEN TYVMERPKNV PTSKWLNSIT SGGLKSCDLD F PLRENTNV ...文字列: MELSPTYQTE RFTKFSDTLK EFKIEQNNEQ NPIDPFNIIR EFRSAAGQLA LDLANSGDES NVISSKDWEL EARFWHLVEL LLVFRNADL DLDEMELHPY NSRGLFEKKL MQDNKQLYQI WIVMVWLKEN TYVMERPKNV PTSKWLNSIT SGGLKSCDLD F PLRENTNV LDVKDKEEDH IFFKYIYELI LAGAIDEALE EAKLSDNISI CMILCGIQEY LNPVIDTQIA NEFNTQQGIK KH SLWRRTV YSLSQQAGLD PYERAIYSYL SGAIPNQEVL QYSDWESDLH IHLNQILQTE IENYLLENNQ VGTDELILPL PSH ALTVQE VLNRVASRHP SESEHPIRVL MASVILDSLP SVIHSSVEML LDVVKGTEAS NDIIDKPYLL RIVTHLAICL DIIN PGSVE EVDKSKLITT YISLLKLQGL YENIPIYATF LNESDCLEAC SFILSSLEDP QVRKKQIETI NFLRLPASNI LRRTT QRVF DETEQEYSPS NEISISFDVN NIDMHLIYGV EWLIEGKLYV DAVHSIIALS RRFLLNGRVK ALEQFMERNN IGEICK NYE LEKIADNISK DENEDQFLEE ITQYEHLIKG IREYEEWQKS VSLLSSESNI PTLIEKLQGF SKDTFELIKT FLVDLTS SN FADSADYEIL YEIRALYTPF LLMELHKKLV EAAKLLKIPK FISEALAFTS LVANENDKIY LLFQSSGKLK EYLDLVAR T ATLSN UniProtKB: Nucleoporin NUP84 |
-分子 #7: NUP133 isoform 1
| 分子 | 名称: NUP133 isoform 1 / タイプ: protein_or_peptide / ID: 7 / 詳細: tail Nup84 Y-complex / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 分子量 | 理論値: 133.452672 KDa |
| 配列 | 文字列: MSEKKVHLRL RKELSVPIAV VENESLAQLS YEEESQASLM DISMEQQQLR LHSHFDNSKV FTENNRYIVK TLQTDYSSGF SNDDELNGY IDMQIGYGLV NDHKKVYIWN IHSTQKDTPY ITVPFRSDDN DEIAVAPRCI LTFPATMDES PLALNPNDQD E TGGLIIIK ...文字列: MSEKKVHLRL RKELSVPIAV VENESLAQLS YEEESQASLM DISMEQQQLR LHSHFDNSKV FTENNRYIVK TLQTDYSSGF SNDDELNGY IDMQIGYGLV NDHKKVYIWN IHSTQKDTPY ITVPFRSDDN DEIAVAPRCI LTFPATMDES PLALNPNDQD E TGGLIIIK GSKAIYYEDI NSINNLNFKL SEKFSHELEL PINSSGGEKC DLMLNCEPAG IVLSTNMGRI FFITIRNSMG KP QLKLGKL LNKPFKLGIW SKIFNTNSSV VSLRNGPILG KGTRLVYITT NKGIFQTWQL SATNSHPTKL IDVNIYEAIL ESL QDLYPF AHGTLKIWDS HPLQDESSQL FLSSIYDSSC NETYYILSTI IFDSSSNSFT IFSTYRLNTF MESITDTKFK PKIF IPQME NANDTNEVTS ILVMFPNAVV ITQVNSKLDS SYSMRRKWED IVSLRNDIDI IGSGYDSKSL YVLTKQMGVL QFFVK ENEE TNSKPEVGFV KSHVDQAVYF SKINANPIDF NLPPEISLDQ ESIEHDLKLT SEEIFHSNGK YIPPMLNTLG QHLSVR KEF FQNFLTFVAK NFNYKISPEL KLDLIEKFEI LNCCIKFNSI IRQSDVLNDI WEKTLSNYNL TQNEHLTTKT VVINSPD VF PVIFKQFLNH VVFVLFPSQN QNFKLNVTNL INLCFYDGIL EEGEKTIRYE LLELDPMEVD TSKLPWFINF DYLNCINQ C FFDFTFACEE EGSLDSYKEG LLKIVKILYY QFNQFKIWIN TQPVKSVNAN DNFININNLY DDNHLDWNHV LCKVNLKEQ CIQIAEFYKD LSGLVQTLQT LDQNDSTTVS LYETFFNEFP KEFSFTLFEY LIKHKKLNDL IFRFPQQHDV LIQFFQESAP KYGHVAWIQ QILDGSYADA MNTLKNITVD DSKKGESLSE CELHLNVAKL SSLLVEKDNL DINTLRKIQY NLDTIDAEKN I SNKLKKGE VQICKRFKNG SIREVFNILV EELKSTTVVN LSDLVELYSM LDDEESLFIP LRLLSVDGNL LNFEVKKFLN AL VWRRIVL LNASNEGDKL LQHIVKRVFD EELPKNNDFP LPSVDLLCDK SLLTPEYISE TYGRFPIDQN AIREEIYEEI SQV ETLNSD NSLEIKLHST IGSVAKEKNY TINYETNTVE Y UniProtKB: NUP133 isoform 1 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 20mM HEPES,50mM Potassium acetate,20mM NaCl,2mM MgCl2,1mM DTT |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK III |
| 詳細 | One step affinity purified |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 倍率(補正後): 37651 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.5 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 詳細 | Preliminary grid screening done manually with individual images of low magnification montages of candidate meshes. |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 2-40 / 撮影したグリッド数: 1 / 実像数: 4015 / 平均電子線量: 40.0 e/Å2 / 詳細: 3218 images retained after triage |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: Other / Chain - Initial model type: experimental model / 詳細: none |
|---|---|
| 詳細 | Molecular dynamics flexible fitting after docking Alphafold2 CSMs for Nups into the 3D map of double Y complex ring with Chimera |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 当てはまり具合の基準: cross correlation |
| 得られたモデル |  PDB-8tie: |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X