+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Tropomyosin-receptor kinase fused gene protein (TRK-fused gene protein; TFG) Low Complexity Domain (residues 237-327) P285L mutant, amyloid fiber | |||||||||||||||||||||
 マップデータ マップデータ | ||||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||
 キーワード キーワード |  amyloid (アミロイド) / amyloid (アミロイド) /  mutation (突然変異) / mutation (突然変異) /  Charcot-Marie-Tooth disease (シャルコー・マリー・トゥース病) / Hereditary motor and sensory neuropathy with proximal dominant involvement / PROTEIN FIBRIL Charcot-Marie-Tooth disease (シャルコー・マリー・トゥース病) / Hereditary motor and sensory neuropathy with proximal dominant involvement / PROTEIN FIBRIL | |||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報COPII vesicle coating / COPII-mediated vesicle transport / Signaling by ALK fusions and activated point mutants / endoplasmic reticulum to Golgi vesicle-mediated transport / endoplasmic reticulum exit site /  生物発光 / generation of precursor metabolites and energy / positive regulation of canonical NF-kappaB signal transduction / intracellular membrane-bounded organelle / identical protein binding ...COPII vesicle coating / COPII-mediated vesicle transport / Signaling by ALK fusions and activated point mutants / endoplasmic reticulum to Golgi vesicle-mediated transport / endoplasmic reticulum exit site / 生物発光 / generation of precursor metabolites and energy / positive regulation of canonical NF-kappaB signal transduction / intracellular membrane-bounded organelle / identical protein binding ...COPII vesicle coating / COPII-mediated vesicle transport / Signaling by ALK fusions and activated point mutants / endoplasmic reticulum to Golgi vesicle-mediated transport / endoplasmic reticulum exit site /  生物発光 / generation of precursor metabolites and energy / positive regulation of canonical NF-kappaB signal transduction / intracellular membrane-bounded organelle / identical protein binding / 生物発光 / generation of precursor metabolites and energy / positive regulation of canonical NF-kappaB signal transduction / intracellular membrane-bounded organelle / identical protein binding /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||||||||
| 手法 | らせん対称体再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.59 Å クライオ電子顕微鏡法 / 解像度: 2.59 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Rosenberg GM / Sawaya MR / Boyer DR / Ge P / Abskharon R / Eisenberg DS | |||||||||||||||||||||
| 資金援助 |  米国, 6件 米国, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: PNAS Nexus / 年: 2023 ジャーナル: PNAS Nexus / 年: 2023タイトル: Fibril structures of TFG protein mutants validate the identification of TFG as a disease-related amyloid protein by the IMPAcT method. 著者: Gregory M Rosenberg / Romany Abskharon / David R Boyer / Peng Ge / Michael R Sawaya / David S Eisenberg /  要旨: We previously presented a bioinformatic method for identifying diseases that arise from a mutation in a protein's low-complexity domain that drives the protein into pathogenic amyloid fibrils. One ...We previously presented a bioinformatic method for identifying diseases that arise from a mutation in a protein's low-complexity domain that drives the protein into pathogenic amyloid fibrils. One protein so identified was the tropomyosin-receptor kinase-fused gene protein (TRK-fused gene protein or TFG). Mutations in TFG are associated with degenerative neurological conditions. Here, we present experimental evidence that confirms our prediction that these conditions are amyloid-related. We find that the low-complexity domain of TFG containing the disease-related mutations G269V or P285L forms amyloid fibrils, and we determine their structures using cryo-electron microscopy (cryo-EM). These structures are unmistakably amyloid in nature and confirm the propensity of the mutant TFG low-complexity domain to form amyloid fibrils. Also, despite resulting from a pathogenic mutation, the fibril structures bear some similarities to other amyloid structures that are thought to be nonpathogenic and even functional, but there are other factors that support these structures' relevance to disease, including an increased propensity to form amyloid compared with the wild-type sequence, structure-stabilizing influence from the mutant residues themselves, and double-protofilament amyloid cores. Our findings elucidate two potentially disease-relevant structures of a previously unknown amyloid and also show how the structural features of pathogenic amyloid fibrils may not conform to the features commonly associated with pathogenicity. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41198.map.gz emd_41198.map.gz | 198.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41198-v30.xml emd-41198-v30.xml emd-41198.xml emd-41198.xml | 16.7 KB 16.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_41198.png emd_41198.png | 149 KB | ||
| Filedesc metadata |  emd-41198.cif.gz emd-41198.cif.gz | 6 KB | ||
| その他 |  emd_41198_half_map_1.map.gz emd_41198_half_map_1.map.gz emd_41198_half_map_2.map.gz emd_41198_half_map_2.map.gz | 171.1 MB 171.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41198 http://ftp.pdbj.org/pub/emdb/structures/EMD-41198 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41198 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41198 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8terMC  8teqC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41198.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41198.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.539 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_41198_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
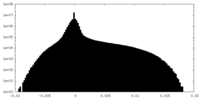
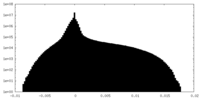
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_41198_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
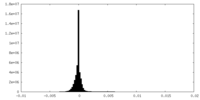
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : amyloid fibril of protein TFG P285L
| 全体 | 名称: amyloid fibril of protein TFG P285L |
|---|---|
| 要素 |
|
-超分子 #1: amyloid fibril of protein TFG P285L
| 超分子 | 名称: amyloid fibril of protein TFG P285L / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: TRK-fused gene protein Low Complexity Domain P285L mutant
| 分子 | 名称: TRK-fused gene protein Low Complexity Domain P285L mutant タイプ: protein_or_peptide / ID: 1 / コピー数: 20 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 40.464754 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: MSYYHHHHHH DYDIPTTENL YFQGAMVSKG EEDNMAIIKE FMRFKVHMEG SVNGHEFEIE GEGEGRPYEG TQTAKLKVTK GGPLPFAWD ILSPQFMYGS KAYVKHPADI PDYLKLSFPE GFKWERVMNF EDGGVVTVTQ DSSLQDGEFI YKVKLRGTNF P SDGPVMQK ...文字列: MSYYHHHHHH DYDIPTTENL YFQGAMVSKG EEDNMAIIKE FMRFKVHMEG SVNGHEFEIE GEGEGRPYEG TQTAKLKVTK GGPLPFAWD ILSPQFMYGS KAYVKHPADI PDYLKLSFPE GFKWERVMNF EDGGVVTVTQ DSSLQDGEFI YKVKLRGTNF P SDGPVMQK KTMGWEASSE RMYPEDGALK GEIKQRLKLK DGGHYDAEVK TTYKAKKPVQ LPGAYNVNIK LDITSHNEDY TI VEQYERA EGRHSTGGMD ELYKAGTMDP GQIEGQMYQQ YQQQAGYGAQ QPQAPPQQPQ QYGIQYSASY SQQTGPQQLQ QFQ GYGQQP TSQAPAPAFS GQPQQLPAQP PQQYQASNYP AQ UniProtKB: Uncharacterized protein, Protein TFG |
-分子 #2: water
| 分子 | 名称: water / タイプ: ligand / ID: 2 / コピー数: 240 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 10 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: PBS |
| グリッド | モデル: Quantifoil R1.2/1.3 |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV / 詳細: 8 seconds, blot force 0. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 1.8 µm / 倍率(公称値): 81000 Bright-field microscopy / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 1.8 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 実像数: 7920 / 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 最終 角度割当 | タイプ: NOT APPLICABLE |
| 最終 再構成 | 使用したクラス数: 1 想定した対称性 - らせんパラメータ - Δz: 2.39 Å 想定した対称性 - らせんパラメータ - ΔΦ: 179.02 ° 想定した対称性 - らせんパラメータ - 軸対称性: C1 (非対称) 解像度のタイプ: BY AUTHOR / 解像度: 2.59 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION / 使用した粒子像数: 193640 |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 温度因子: 14.16 / 当てはまり具合の基準: Correlation coefficient |
|---|---|
| 得られたモデル |  PDB-8ter: |
 ムービー
ムービー コントローラー
コントローラー










 Z
Z Y
Y X
X