+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 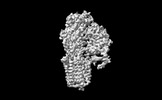 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of ATP13A2 in the E1P-ADP state | |||||||||
 マップデータ マップデータ | Cryo-EM structure of ATP13A2 in the E1P-ADP state | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Cryo-EM structure of ATP13A2 in the E1P-ADP state /  Membrane protein (膜タンパク質) / Membrane protein (膜タンパク質) /  PROTEIN TRANSPORT PROTEIN TRANSPORT | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報polyamine transmembrane transporter activity / ABC-type polyamine transporter activity / polyamine transmembrane transport / spermine transmembrane transport / peptidyl-aspartic acid autophosphorylation / regulation of ubiquitin-specific protease activity / regulation of autophagosome size / extracellular exosome biogenesis /  regulation of chaperone-mediated autophagy / P-type ion transporter activity ...polyamine transmembrane transporter activity / ABC-type polyamine transporter activity / polyamine transmembrane transport / spermine transmembrane transport / peptidyl-aspartic acid autophosphorylation / regulation of ubiquitin-specific protease activity / regulation of autophagosome size / extracellular exosome biogenesis / regulation of chaperone-mediated autophagy / P-type ion transporter activity ...polyamine transmembrane transporter activity / ABC-type polyamine transporter activity / polyamine transmembrane transport / spermine transmembrane transport / peptidyl-aspartic acid autophosphorylation / regulation of ubiquitin-specific protease activity / regulation of autophagosome size / extracellular exosome biogenesis /  regulation of chaperone-mediated autophagy / P-type ion transporter activity / negative regulation of lysosomal protein catabolic process / regulation of chaperone-mediated autophagy / P-type ion transporter activity / negative regulation of lysosomal protein catabolic process /  regulation of lysosomal protein catabolic process / autophagosome-lysosome fusion / intracellular monoatomic cation homeostasis / regulation of autophagy of mitochondrion / autophagosome organization / protein localization to lysosome / regulation of lysosomal protein catabolic process / autophagosome-lysosome fusion / intracellular monoatomic cation homeostasis / regulation of autophagy of mitochondrion / autophagosome organization / protein localization to lysosome /  phosphatidic acid binding / multivesicular body membrane / positive regulation of exosomal secretion / ATPase-coupled monoatomic cation transmembrane transporter activity / intracellular zinc ion homeostasis / トランスロカーゼ; 他の化合物の輸送を触媒; ヌクレオシド三リン酸の加水分解に伴う / regulation of protein localization to nucleus / cupric ion binding / phosphatidylinositol-3,5-bisphosphate binding / regulation of mitochondrion organization / phosphatidic acid binding / multivesicular body membrane / positive regulation of exosomal secretion / ATPase-coupled monoatomic cation transmembrane transporter activity / intracellular zinc ion homeostasis / トランスロカーゼ; 他の化合物の輸送を触媒; ヌクレオシド三リン酸の加水分解に伴う / regulation of protein localization to nucleus / cupric ion binding / phosphatidylinositol-3,5-bisphosphate binding / regulation of mitochondrion organization /  regulation of endopeptidase activity / lysosomal transport / cellular response to zinc ion / regulation of endopeptidase activity / lysosomal transport / cellular response to zinc ion /  regulation of intracellular protein transport / lipid homeostasis / autophagosome membrane / Ion transport by P-type ATPases / regulation of intracellular protein transport / lipid homeostasis / autophagosome membrane / Ion transport by P-type ATPases /  オートファゴソーム / オートファゴソーム /  regulation of macroautophagy / cellular response to manganese ion / regulation of neuron apoptotic process / regulation of macroautophagy / cellular response to manganese ion / regulation of neuron apoptotic process /  小胞 / 小胞 /  エンドソーム / lysosomal lumen / monoatomic ion transmembrane transport / positive regulation of protein secretion / transmembrane transport / intracellular calcium ion homeostasis / エンドソーム / lysosomal lumen / monoatomic ion transmembrane transport / positive regulation of protein secretion / transmembrane transport / intracellular calcium ion homeostasis /  オートファジー / late endosome / cellular response to oxidative stress / late endosome membrane / manganese ion binding / intracellular iron ion homeostasis / vesicle / protein autophosphorylation / オートファジー / late endosome / cellular response to oxidative stress / late endosome membrane / manganese ion binding / intracellular iron ion homeostasis / vesicle / protein autophosphorylation /  リソソーム / neuron projection / lysosomal membrane / neuronal cell body / positive regulation of gene expression / リソソーム / neuron projection / lysosomal membrane / neuronal cell body / positive regulation of gene expression /  ATP hydrolysis activity / zinc ion binding / ATP hydrolysis activity / zinc ion binding /  ATP binding / ATP binding /  生体膜 生体膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.73 Å クライオ電子顕微鏡法 / 解像度: 3.73 Å | |||||||||
 データ登録者 データ登録者 | Liu ZM / Mu JQ / Xue CY | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Conformational cycle of human polyamine transporter ATP13A2. 著者: Jianqiang Mu / Chenyang Xue / Lei Fu / Zongjun Yu / Minhan Nie / Mengqi Wu / Xinmeng Chen / Kun Liu / Ruiqian Bu / Ying Huang / Baisheng Yang / Jianming Han / Qianru Jiang / Kevin C Chan / ...著者: Jianqiang Mu / Chenyang Xue / Lei Fu / Zongjun Yu / Minhan Nie / Mengqi Wu / Xinmeng Chen / Kun Liu / Ruiqian Bu / Ying Huang / Baisheng Yang / Jianming Han / Qianru Jiang / Kevin C Chan / Ruhong Zhou / Huilin Li / Ancheng Huang / Yong Wang / Zhongmin Liu /  要旨: Dysregulation of polyamine homeostasis strongly associates with human diseases. ATP13A2, which is mutated in juvenile-onset Parkinson's disease and autosomal recessive spastic paraplegia 78, is a ...Dysregulation of polyamine homeostasis strongly associates with human diseases. ATP13A2, which is mutated in juvenile-onset Parkinson's disease and autosomal recessive spastic paraplegia 78, is a transporter with a critical role in balancing the polyamine concentration between the lysosome and the cytosol. Here, to better understand human ATP13A2-mediated polyamine transport, we use single-particle cryo-electron microscopy to solve high-resolution structures of human ATP13A2 in six intermediate states, including the putative E2 structure for the P5 subfamily of the P-type ATPases. These structures comprise a nearly complete conformational cycle spanning the polyamine transport process and capture multiple substrate binding sites distributed along the transmembrane regions, suggesting a potential polyamine transport pathway. Integration of high-resolution structures, biochemical assays, and molecular dynamics simulations allows us to obtain a better understanding of the structural basis of how hATP13A2 transports polyamines, providing a mechanistic framework for ATP13A2-related diseases. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_35392.map.gz emd_35392.map.gz | 97.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-35392-v30.xml emd-35392-v30.xml emd-35392.xml emd-35392.xml | 16 KB 16 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_35392.png emd_35392.png | 36.5 KB | ||
| Filedesc metadata |  emd-35392.cif.gz emd-35392.cif.gz | 6.1 KB | ||
| その他 |  emd_35392_half_map_1.map.gz emd_35392_half_map_1.map.gz emd_35392_half_map_2.map.gz emd_35392_half_map_2.map.gz | 95.6 MB 95.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-35392 http://ftp.pdbj.org/pub/emdb/structures/EMD-35392 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35392 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35392 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8iesMC  8iekC  8ielC  8iemC  8ienC  8ieoC  8ierC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_35392.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_35392.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of ATP13A2 in the E1P-ADP state | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.095 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: The half a map of ATP13A2 in the E1P-ADP state
| ファイル | emd_35392_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | The half_a map of ATP13A2 in the E1P-ADP state | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: The half b map of ATP13A2 in the E1P-ADP state
| ファイル | emd_35392_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | The half_b map of ATP13A2 in the E1P-ADP state | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM structure of ATP13A2 in the E1P-ADP state
| 全体 | 名称: Cryo-EM structure of ATP13A2 in the E1P-ADP state |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM structure of ATP13A2 in the E1P-ADP state
| 超分子 | 名称: Cryo-EM structure of ATP13A2 in the E1P-ADP state / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Polyamine-transporting ATPase 13A2
| 分子 | 名称: Polyamine-transporting ATPase 13A2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO EC番号: トランスロカーゼ; 他の化合物の輸送を触媒; ヌクレオシド三リン酸の加水分解に伴う |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 125.560297 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: RLSGYCGSPW RVIGYHVVVW MMAGIPLLLF RWKPLWGVRL RLRPCNLAHA ETLVIEIRDK EDSSWQLFTV QVQTEAIGEG SLEPSPQSQ AEDGRSQAAV GAVPEGAWKD TAQLHKSEEA VSVGQKRVLR YYLFQGQRYI WIETQQAFYQ VSLLDHGRSC D DVHRSRHG ...文字列: RLSGYCGSPW RVIGYHVVVW MMAGIPLLLF RWKPLWGVRL RLRPCNLAHA ETLVIEIRDK EDSSWQLFTV QVQTEAIGEG SLEPSPQSQ AEDGRSQAAV GAVPEGAWKD TAQLHKSEEA VSVGQKRVLR YYLFQGQRYI WIETQQAFYQ VSLLDHGRSC D DVHRSRHG LSLQDQMVRK AIYGPNVISI PVKSYPQLLV DEALNPYYGF QAFSIALWLA DHYYWYALCI FLISSISICL SL YKTRKQS QTLRDMVKLS MRVCVCRPGG EEEWVDSSEL VPGDCLVLPQ EGGLMPCDAA LVAGECMVNE SSLTGESIPV LKT ALPEGL GPYCAETHRR HTLFCGTLIL QARAYVGPHV LAVVTRTGFC TAKGGLVSSI LHPRPINFKF YKHSMKFVAA LSVL ALLGT IYSIFILYRN RVPLNEIVIR ALDLVTVVVP PALPAAMTVC TLYAQSRLRR QGIFCIHPLR INLGGKLQLV CFDKT GTLT EDGLDVMGVV PLKGQAFLPL VPEPRRLPVG PLLRALATCH ALSRLQDTPV GDPMDLKMVE STGWVLEEEP AADSAF GTQ VLAVMRPPLW EPQLQAMEEP PVPVSVLHRF PFSSALQRMS VVVAWPGATQ PEAYVKGSPE LVAGLCNPET VPTDFAQ ML QSYTAAGYRV VALASKPLPT VPSLEAAQQL TRDTVEGDLS LLGLLVMRNL LKPQTTPVIQ ALRRTRIRAV MVTGDNLQ T AVTVARGCGM VAPQEHLIIV HATHPERGQP ASLEFLPMES PTAVNGVKDP DQAASYTVEP DPRSRHLALS GPTFGIIVK HFPKLLPKVL VQGTVFARMA PEQKTELVCE LQKLQYCVGM CGDGANDCGA LKAADVGISL SQAEASVVSP FTSSMASIEC VPMVIREGR CSLDTSFSVF KYMALYSLTQ FISVLILYTI NTNLGDLQFL AIDLVITTTV AVLMSRTGPA LVLGRVRPPG A LLSVPVLS SLLLQMVLVT GVQLGGYFLT LAQPWFVPLN RTVAAPDNLP NYENTVVFSL SSFQYLILAA AVSKGAPFRR PL YTNVPFL VALALLSSVL VGLVLVPGLL QGPLALRNIT DTGFKLLLLG LVTLNFVGAF MLESVLDQCL PACLRRLRPK RAS KKRFKQ LERELAEQPW PPLPAGPLR UniProtKB: Polyamine-transporting ATPase 13A2 |
-分子 #2: TETRAFLUOROALUMINATE ION
| 分子 | 名称: TETRAFLUOROALUMINATE ION / タイプ: ligand / ID: 2 / コピー数: 1 / 式: ALF |
|---|---|
| 分子量 | 理論値: 102.975 Da |
| Chemical component information |  ChemComp-ALF: |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #4: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 1 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 1.2 µm Bright-field microscopy / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.73 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 151804 |
 ムービー
ムービー コントローラー
コントローラー



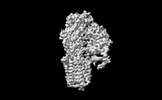

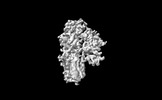



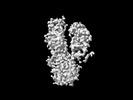





 Z
Z Y
Y X
X