+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | GluK1-1a extracellular domain captured in SYM2081 bound desensitized state | |||||||||
 マップデータ マップデータ | GluK1-1a extracellular domain captured in desensitized state | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  Kainate receptor (カイニン酸型グルタミン酸受容体) / GluK1-1a splice variant / Kainate receptor (カイニン酸型グルタミン酸受容体) / GluK1-1a splice variant /  MEMBRANE PROTEIN (膜タンパク質) MEMBRANE PROTEIN (膜タンパク質) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ligand-gated monoatomic ion channel activity /  signaling receptor activity / signaling receptor activity /  postsynaptic membrane postsynaptic membrane類似検索 - 分子機能 | |||||||||
| 生物種 |   Rattus norvegicus (ドブネズミ) Rattus norvegicus (ドブネズミ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 8.01 Å クライオ電子顕微鏡法 / 解像度: 8.01 Å | |||||||||
 データ登録者 データ登録者 | Dhingra S / Kumar J | |||||||||
| 資金援助 |  インド, 1件 インド, 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2023 ジャーナル: Elife / 年: 2023タイトル: Functional Implications of the Exon 9 Splice Insert in GluK1 Kainate Receptors 著者: Dhingra S / Chopade PM / Vinnakota R / Kumar J | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
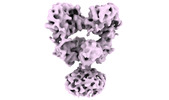
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34083.map.gz emd_34083.map.gz | 32.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34083-v30.xml emd-34083-v30.xml emd-34083.xml emd-34083.xml | 17.6 KB 17.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_34083.png emd_34083.png | 51.3 KB | ||
| マスクデータ |  emd_34083_msk_1.map emd_34083_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-34083.cif.gz emd-34083.cif.gz | 6 KB | ||
| その他 |  emd_34083_additional_1.map.gz emd_34083_additional_1.map.gz emd_34083_half_map_1.map.gz emd_34083_half_map_1.map.gz emd_34083_half_map_2.map.gz emd_34083_half_map_2.map.gz | 57.6 MB 59.5 MB 59.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34083 http://ftp.pdbj.org/pub/emdb/structures/EMD-34083 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34083 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34083 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34083.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34083.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | GluK1-1a extracellular domain captured in desensitized state | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.38 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_34083_msk_1.map emd_34083_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
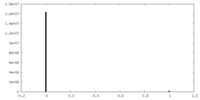
| 投影像・断面図 |
| ||||||||||||
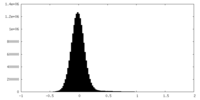
| 密度ヒストグラム |
-追加マップ: GluK1-1a extracellular domain captured in desensitized state additional EM...
| ファイル | emd_34083_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | GluK1-1a extracellular domain captured in desensitized state_additional EM map | ||||||||||||
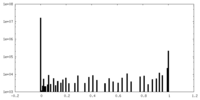
| 投影像・断面図 |
| ||||||||||||
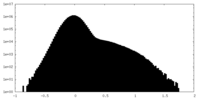
| 密度ヒストグラム |
-ハーフマップ: GluK1-1a extracellular domain captured in desensitized state halfmap A...
| ファイル | emd_34083_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | GluK1-1a extracellular domain captured in desensitized state halfmap_A | ||||||||||||
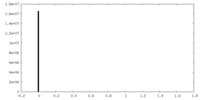
| 投影像・断面図 |
| ||||||||||||
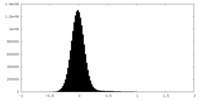
| 密度ヒストグラム |
-ハーフマップ: GluK1-1a extracellular domain captured in desensitized state halfmap B...
| ファイル | emd_34083_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | GluK1-1a extracellular domain captured in desensitized state halfmap_B | ||||||||||||
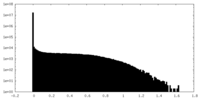
| 投影像・断面図 |
| ||||||||||||
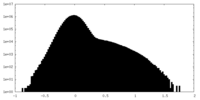
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of GluK1-1a extracellular domain with SYM2081
| 全体 | 名称: Complex of GluK1-1a extracellular domain with SYM2081 |
|---|---|
| 要素 |
|
-超分子 #1: Complex of GluK1-1a extracellular domain with SYM2081
| 超分子 | 名称: Complex of GluK1-1a extracellular domain with SYM2081 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Rattus norvegicus (ドブネズミ) Rattus norvegicus (ドブネズミ) |
| 分子量 | 理論値: 390 KDa |
-分子 #1: Glutamate receptor
| 分子 | 名称: Glutamate receptor / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Rattus norvegicus (ドブネズミ) Rattus norvegicus (ドブネズミ) |
| 分子量 | 理論値: 95.606672 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVLRIGGIFE TVENEPVNVE ELAFKFAVTS INRNRTLMPN TTLTYDIQRI NLFDSFEASR RACDQLALGV AALFGPSHSS SVSAVQSIC NALEVPHIQT RWKHPSVDSR DLFYINLYPD YAAISRAVLD LVLYYNWKTV TVVYEDSTGL IRLQELIKAP S RYNIKIKI ...文字列: QVLRIGGIFE TVENEPVNVE ELAFKFAVTS INRNRTLMPN TTLTYDIQRI NLFDSFEASR RACDQLALGV AALFGPSHSS SVSAVQSIC NALEVPHIQT RWKHPSVDSR DLFYINLYPD YAAISRAVLD LVLYYNWKTV TVVYEDSTGL IRLQELIKAP S RYNIKIKI RQLPPANKDA KPLLKEMKKS KEFYVIFDCS HETAAEILKQ ILFMGMMTEY YHYFFTTLDL FALDLELYRY SG VNMTGFR LLNIDNPHVS SIIEKWSMER LQAPPRPETG LLDGMMTTEA ALMYDAVYMV AIASHRASQL TVSSLQCHRH KPW RLGPRF MNLIKEARWD GLTGRITFNK TDGLRKDFDL DIISLKEEGT EKASGEVSKH LYKVWKKIGI WNSNSGLNMT DGNR DRSNN ITDSLANRTL IVTTILEEPY VMYRKSDKPL YGNDRFEGYC LDLLKELSNI LGFLYDVKLV PDGKYGAQND KGEWN GMVK ELIDHRADLA VAPLTITYVR EKVIDFSKPF MTLGISILYR KPNGTNPGVF SFLNPLSPDI WMYVLLAYLG VSVVLF VIA RFTPYEWYNP HPCNPDSDVV ENNFTLLNSF WFGVGALMQQ GSELMPKALS TRIVGGIWWF FTLIIISSYT ANLAAFL TV ERMESPIDSA DDLAKQTKIE YGAVRDGSTM TFFKKSKIST YEKMWAFMSS RQQSALVKNS DEGIQRVLTT DYALLMES T SIEYVTQRNC NLTQIGGLID SKGYGVGTPI GSPYRDKITI AILQLQEEGK LHMMKEKWWR GNGCPEEDSK EASALGVEN IGGIFIVLAA GLVLSVFVAI GEFLYKSRKN NDVEQHYLVP R UniProtKB:  グルタミン酸受容体 グルタミン酸受容体 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.57 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Quantifoil R0.6/1 / 材質: GOLD / メッシュ: 300 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 0.5 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 1100 / 平均露光時間: 60.0 sec. / 平均電子線量: 19.5 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: PDB ENTRY PDBモデル - PDB ID: 詳細: Unpublished coordinates for the amino-terminal domain model 3C32 used for ligand binding domain model |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 8.01 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 5372 |
 ムービー
ムービー コントローラー
コントローラー










 Z
Z Y
Y X
X