+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3373 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | electron density map of murine leukaemia virus envelope glycoprotein as reconstructed by subtomogram averaging applying a mask on 1 protomer on murine leukemia virus particles and virus like particles and applying 3fold symmetry to the single protomer afterwards | |||||||||
 マップデータ マップデータ | murine leukemia virus Env reconstructed by subtomogram averaging of 1 protomer, thereafter application of 3-fold symmetry | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  murine leukemia virus (マウス白血病ウイルス) / murine leukemia virus (マウス白血病ウイルス) /  retrovirus (レトロウイルス科) / envelope glycoprotein / retrovirus (レトロウイルス科) / envelope glycoprotein /  cryo electron tomography / subtomogram averaging cryo electron tomography / subtomogram averaging | |||||||||
| 生物種 |   Murine leukemia virus (ネズミ白血病ウイルス) Murine leukemia virus (ネズミ白血病ウイルス) | |||||||||
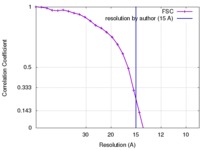
| 手法 | サブトモグラム平均法 /  クライオ電子顕微鏡法 / 解像度: 15.0 Å クライオ電子顕微鏡法 / 解像度: 15.0 Å | |||||||||
 データ登録者 データ登録者 | Riedel C / Vasishtan D / Siebert CA / Whittle C / Lehmann MJ / Mothes W / Grunewald K | |||||||||
 引用 引用 |  ジャーナル: J Struct Biol / 年: 2017 ジャーナル: J Struct Biol / 年: 2017タイトル: Native structure of a retroviral envelope protein and its conformational change upon interaction with the target cell. 著者: Christiane Riedel / Daven Vasishtan / C Alistair Siebert / Cathy Whittle / Maik J Lehmann / Walther Mothes / Kay Grünewald /     要旨: Enveloped viruses enter their host cells by membrane fusion. The process of attachment and fusion in retroviruses is mediated by a single viral envelope glycoprotein (Env). Conformational changes of ...Enveloped viruses enter their host cells by membrane fusion. The process of attachment and fusion in retroviruses is mediated by a single viral envelope glycoprotein (Env). Conformational changes of Env in the course of fusion are a focus of intense studies. Here we provide further insight into the changes occurring in retroviral Env during its initial interaction with the cell, employing murine leukemia virus (MLV) as model system. We first determined the structure of both natively membrane anchored MLV Env and MLV Env tagged with YFP in the proline rich region (PRR) by electron cryo tomography (cET) and sub-volume averaging. At a resolution of ∼20Å, native MLV Env presents as a hollow trimer (height ∼85Å, diameter ∼120Å) composed of step-shaped protomers. The major difference to the YFP-tagged protein was in regions outside of the central trimer. Next, we focused on elucidating the changes in MLV Env upon interaction with a host cell. Virus interaction with the plasma membrane occurred over a large surface and Env clustering on the binding site was observed. Sub-volume averaging did yield a low-resolution structure of Env interacting with the cell, which had lost its threefold symmetry and was elongated by ∼35Å in comparison to the unbound protein. This indicates a major rearrangement of Env upon host cell binding. At the site of virus interaction, the otherwise clearly defined bilayer structure of the host cell plasma membrane was much less evident, indicative of integral membrane protein accumulation and/or a change in membrane lipid composition. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3373.map.gz emd_3373.map.gz | 37.3 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3373-v30.xml emd-3373-v30.xml emd-3373.xml emd-3373.xml | 15.7 KB 15.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3373_fsc.xml emd_3373_fsc.xml | 2.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3373.tif emd_3373.tif | 732.6 KB | ||
| マスクデータ |  emd_3373_msk_1.map emd_3373_msk_1.map | 381.2 KB |  マスクマップ マスクマップ | |
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3373 http://ftp.pdbj.org/pub/emdb/structures/EMD-3373 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3373 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3373 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3373.map.gz / 形式: CCP4 / 大きさ: 373 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3373.map.gz / 形式: CCP4 / 大きさ: 373 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | murine leukemia virus Env reconstructed by subtomogram averaging of 1 protomer, thereafter application of 3-fold symmetry | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.6 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-セグメンテーションマップ: This mask represents 1 protomer.
| 注釈 | This mask represents 1 protomer. | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ファイル |  emd_3373_msk_1.map emd_3373_msk_1.map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : murine leukemia virus Env protein on murine leukemia virus and vi...
| 全体 | 名称: murine leukemia virus Env protein on murine leukemia virus and virus like particles, reconstruction of 1 protomer and afterwards application of C3. |
|---|---|
| 要素 |
|
-超分子 #1000: murine leukemia virus Env protein on murine leukemia virus and vi...
| 超分子 | 名称: murine leukemia virus Env protein on murine leukemia virus and virus like particles, reconstruction of 1 protomer and afterwards application of C3. タイプ: sample / ID: 1000 / 集合状態: trimer / Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 226 KDa |
-分子 #1: murine leukemia virus Env protein
| 分子 | 名称: murine leukemia virus Env protein / タイプ: protein_or_peptide / ID: 1 / 詳細: imaged on intact virus and virus like particles / コピー数: 3 / 集合状態: trimer / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:   Murine leukemia virus (ネズミ白血病ウイルス) Murine leukemia virus (ネズミ白血病ウイルス)株: Friend's murine leukemia virus |
| 分子量 | 理論値: 226 KDa |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 / 詳細: DMEM + 10% FSC |
|---|---|
| グリッド | 詳細: C-flat copper grids (Protochips, CF-2/1-2C) |
| 凍結 | 凍結剤: ETHANE-PROPANE MIXTURE / 装置: OTHER / 手法: manually blotted for 3sec |
- 電子顕微鏡法 #1
電子顕微鏡法 #1
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 95000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 1.5 µm Bright-field microscopy / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 特殊光学系 | エネルギーフィルター - 名称: Gatan QUANTUM 964 postcolumn energy filter エネルギーフィルター - エネルギー下限: 0.0 eV エネルギーフィルター - エネルギー上限: 20.0 eV |
| 試料ステージ | 試料ホルダーモデル: OTHER / Tilt series - Axis1 - Min angle: -45 ° / Tilt series - Axis1 - Max angle: 45 ° |
| Microscopy ID | 1 |
| 日付 | 2014年8月6日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 60 e/Å2 |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 電子顕微鏡法 #2
電子顕微鏡法 #2
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 95000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 1.5 µm Bright-field microscopy / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 特殊光学系 | エネルギーフィルター - 名称: Gatan QUANTUM 964 postcolumn energy filter エネルギーフィルター - エネルギー下限: 0.0 eV エネルギーフィルター - エネルギー上限: 20.0 eV |
| 試料ステージ | 試料ホルダーモデル: OTHER / Tilt series - Axis1 - Min angle: -45 ° / Tilt series - Axis1 - Max angle: 45 ° |
| Microscopy ID | 2 |
| 日付 | 2014年8月13日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 60 e/Å2 |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 電子顕微鏡法 #3
電子顕微鏡法 #3
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 95000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 1.5 µm Bright-field microscopy / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 特殊光学系 | エネルギーフィルター - 名称: Gatan QUANTUM 964 postcolumn energy filter エネルギーフィルター - エネルギー下限: 0.0 eV エネルギーフィルター - エネルギー上限: 20.0 eV |
| 試料ステージ | 試料ホルダーモデル: OTHER / Tilt series - Axis1 - Min angle: -45 ° / Tilt series - Axis1 - Max angle: 45 ° |
| Microscopy ID | 3 |
| 日付 | 2014年10月8日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 60 e/Å2 |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー















 Z
Z Y
Y X
X