| 登録情報 | データベース: EMDB / ID: EMD-31716
|
|---|
| タイトル | Cryo-EM structure of Alphavirus M1 |
|---|
 マップデータ マップデータ | Cryo-EM structure of alphavirus M1 |
|---|
 試料 試料 | - ウイルス:
  Alphavirus M1 (ウイルス) Alphavirus M1 (ウイルス)- タンパク質・ペプチド: E1 glycoprotein
- タンパク質・ペプチド: E2 glycoprotein
- タンパク質・ペプチド: Capsid protein
 カプシド カプシド
- リガンド: 2-acetamido-2-deoxy-beta-D-glucopyranose
|
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
T=4 icosahedral viral capsid / host cell cytoplasm / membrane => GO:0016020 / symbiont entry into host cell / serine-type endopeptidase activity / fusion of virus membrane with host endosome membrane / virion attachment to host cell / host cell plasma membrane / virion membrane / structural molecule activity ...T=4 icosahedral viral capsid / host cell cytoplasm / membrane => GO:0016020 / symbiont entry into host cell / serine-type endopeptidase activity / fusion of virus membrane with host endosome membrane / virion attachment to host cell / host cell plasma membrane / virion membrane / structural molecule activity /  タンパク質分解 / タンパク質分解 /  細胞膜 / 細胞膜 /  細胞質類似検索 - 分子機能 細胞質類似検索 - 分子機能 Alphavirus E2 glycoprotein, domain B / Peptidase S3, togavirin / Alphavirus E2 glycoprotein / Alphavirus E3 spike glycoprotein / Alphavirus E1 glycoprotein / Alphavirus E2 glycoprotein, domain A / Alphavirus E2 glycoprotein, domain C / Alphavirus E2 glycoprotein / Alphavirus core protein / Alphavirus E3 glycoprotein ...Alphavirus E2 glycoprotein, domain B / Peptidase S3, togavirin / Alphavirus E2 glycoprotein / Alphavirus E3 spike glycoprotein / Alphavirus E1 glycoprotein / Alphavirus E2 glycoprotein, domain A / Alphavirus E2 glycoprotein, domain C / Alphavirus E2 glycoprotein / Alphavirus core protein / Alphavirus E3 glycoprotein / Alphavirus E1 glycoprotein / Alphavirus core protein (CP) domain profile. / Flavivirus/Alphavirus glycoprotein, immunoglobulin-like domain superfamily / Flavivirus glycoprotein, central and dimerisation domain superfamily / Flaviviral glycoprotein E, dimerisation domain / Immunoglobulin E-set / Peptidase S1, PA clan, chymotrypsin-like fold / Peptidase S1, PA clan類似検索 - ドメイン・相同性 |
|---|
| 生物種 |   Alphavirus M1 (ウイルス) Alphavirus M1 (ウイルス) |
|---|
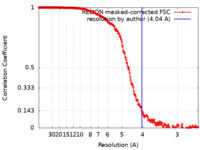
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.04 Å クライオ電子顕微鏡法 / 解像度: 4.04 Å |
|---|
 データ登録者 データ登録者 | Gao Y / Jia X / Zhang Q |
|---|
| 資金援助 |  中国, 5件 中国, 5件 | Organization | Grant number | 国 |
|---|
| National Natural Science Foundation of China (NSFC) | 81973342 |  中国 中国 | | National Natural Science Foundation of China (NSFC) | 81773751 |  中国 中国 | | National Natural Science Foundation of China (NSFC) | 81772675 |  中国 中国 | | National Natural Science Foundation of China (NSFC) | 81903652 |  中国 中国 | | National Natural Science Foundation of China (NSFC) | 81802536 |  中国 中国 |
|
|---|
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Published
タイトル: Cryo-EM evidence of viral N-glycosylation reveal receptor binding mechanisms of alphavirus M1
著者: Song D / Jia X |
|---|
| 履歴 | | 登録 | 2021年8月15日 | - |
|---|
| ヘッダ(付随情報) 公開 | 2022年8月17日 | - |
|---|
| マップ公開 | 2022年8月17日 | - |
|---|
| 更新 | 2022年8月17日 | - |
|---|
| 現状 | 2022年8月17日 | 処理サイト: PDBj / 状態: 公開 |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報
 マップデータ
マップデータ 試料
試料 機能・相同性情報
機能・相同性情報 タンパク質分解 /
タンパク質分解 /  細胞膜 /
細胞膜 /  細胞質
細胞質
 Alphavirus M1 (ウイルス)
Alphavirus M1 (ウイルス) 単粒子再構成法 /
単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.04 Å
クライオ電子顕微鏡法 / 解像度: 4.04 Å  データ登録者
データ登録者 中国, 5件
中国, 5件  引用
引用 ジャーナル: To Be Published
ジャーナル: To Be Published 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_31716.map.gz
emd_31716.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-31716-v30.xml
emd-31716-v30.xml emd-31716.xml
emd-31716.xml EMDBヘッダ
EMDBヘッダ emd_31716_fsc.xml
emd_31716_fsc.xml FSCデータファイル
FSCデータファイル emd_31716.png
emd_31716.png http://ftp.pdbj.org/pub/emdb/structures/EMD-31716
http://ftp.pdbj.org/pub/emdb/structures/EMD-31716 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31716
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31716
 F&H 検索
F&H 検索 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_31716.map.gz / 形式: CCP4 / 大きさ: 600.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_31716.map.gz / 形式: CCP4 / 大きさ: 600.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素
 Alphavirus M1 (ウイルス)
Alphavirus M1 (ウイルス)
 Alphavirus M1 (ウイルス)
Alphavirus M1 (ウイルス)
 Alphavirus M1 (ウイルス)
Alphavirus M1 (ウイルス)
 Alphavirus M1 (ウイルス)
Alphavirus M1 (ウイルス)
 クライオ電子顕微鏡法
クライオ電子顕微鏡法 解析
解析 単粒子再構成法
単粒子再構成法 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN Bright-field microscopy
Bright-field microscopy
 ムービー
ムービー コントローラー
コントローラー