+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-24208 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of PPPA bound human ACAT2 | |||||||||
 マップデータ マップデータ | Structure of PPPA bound human ACAT2 | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 sterol O-acyltransferase / sterol O-acyltransferase /  sterol O-acyltransferase activity / cholesterol O-acyltransferase activity / sterol O-acyltransferase activity / cholesterol O-acyltransferase activity /  O-acyltransferase activity / very-low-density lipoprotein particle assembly / low-density lipoprotein particle clearance / fatty-acyl-CoA binding / intestinal cholesterol absorption / LDL clearance / cholesterol efflux ... O-acyltransferase activity / very-low-density lipoprotein particle assembly / low-density lipoprotein particle clearance / fatty-acyl-CoA binding / intestinal cholesterol absorption / LDL clearance / cholesterol efflux ... sterol O-acyltransferase / sterol O-acyltransferase /  sterol O-acyltransferase activity / cholesterol O-acyltransferase activity / sterol O-acyltransferase activity / cholesterol O-acyltransferase activity /  O-acyltransferase activity / very-low-density lipoprotein particle assembly / low-density lipoprotein particle clearance / fatty-acyl-CoA binding / intestinal cholesterol absorption / LDL clearance / cholesterol efflux / O-acyltransferase activity / very-low-density lipoprotein particle assembly / low-density lipoprotein particle clearance / fatty-acyl-CoA binding / intestinal cholesterol absorption / LDL clearance / cholesterol efflux /  cholesterol binding / macrophage derived foam cell differentiation / cholesterol binding / macrophage derived foam cell differentiation /  acyltransferase activity / acyltransferase activity /  刷子縁 / cholesterol metabolic process / cholesterol homeostasis / endoplasmic reticulum membrane / 刷子縁 / cholesterol metabolic process / cholesterol homeostasis / endoplasmic reticulum membrane /  小胞体 小胞体類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.87 Å クライオ電子顕微鏡法 / 解像度: 3.87 Å | |||||||||
 データ登録者 データ登録者 | Li X / Long T | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2021 ジャーナル: Structure / 年: 2021タイトル: Molecular structures of human ACAT2 disclose mechanism for selective inhibition. 著者: Tao Long / Yang Liu / Xiaochun Li /  要旨: Endoplasmic reticulum-localized acyl-CoA:cholesterol acyltransferases (ACAT), including ACAT1 and ACAT2, convert cholesterol to cholesteryl esters that become incorporated into lipoproteins or stored ...Endoplasmic reticulum-localized acyl-CoA:cholesterol acyltransferases (ACAT), including ACAT1 and ACAT2, convert cholesterol to cholesteryl esters that become incorporated into lipoproteins or stored in cytosolic lipid droplets. Selective inhibition of ACAT2 has been shown to considerably attenuate hypercholesterolemia and atherosclerosis in mice. Here, we report cryogenic electron microscopy structures of human ACAT2 bound to its specific inhibitor pyripyropene A or the general ACAT inhibitor nevanimibe. Structural analysis reveals that ACAT2 has a topology in membranes similar to that of ACAT1. A catalytic core with an entry site occupied by a cholesterol molecule and another site for allosteric activation of ACAT2 is observed in these structures. Enzymatic assays show that mutations within sites of cholesterol entry or allosteric activation attenuate ACAT2 activity in vitro. Together, these results reveal mechanisms for ACAT2-mediated esterification of cholesterol, providing a blueprint to design new ACAT2 inhibitors for use in the prevention of cardiovascular disease. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24208.map.gz emd_24208.map.gz | 26.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24208-v30.xml emd-24208-v30.xml emd-24208.xml emd-24208.xml | 9.7 KB 9.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_24208.png emd_24208.png | 35.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24208 http://ftp.pdbj.org/pub/emdb/structures/EMD-24208 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24208 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24208 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24208.map.gz / 形式: CCP4 / 大きさ: 137.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24208.map.gz / 形式: CCP4 / 大きさ: 137.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Structure of PPPA bound human ACAT2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.833 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : human ACAT2 with PPPA
| 全体 | 名称: human ACAT2 with PPPA |
|---|---|
| 要素 |
|
-超分子 #1: human ACAT2 with PPPA
| 超分子 | 名称: human ACAT2 with PPPA / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Sterol O-acyltransferase 2
| 分子 | 名称: Sterol O-acyltransferase 2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO / EC番号:  sterol O-acyltransferase sterol O-acyltransferase |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 60.893 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDYKDDDDKE PGGARLRLQR TGGLGGERER QPCGDGNTET HRAPDLVQWT RHMEAVKAQL LEQAQGQLRE LLDRAMREAI QSYPSQDKP LPPPPPGSLS RTQEPSLGKQ KVFIIRKSLL DELMEVQHFR TIYHMFIAGL CVFIISTLAI DFIDEGRLLL E FDLLIFSF ...文字列: MDYKDDDDKE PGGARLRLQR TGGLGGERER QPCGDGNTET HRAPDLVQWT RHMEAVKAQL LEQAQGQLRE LLDRAMREAI QSYPSQDKP LPPPPPGSLS RTQEPSLGKQ KVFIIRKSLL DELMEVQHFR TIYHMFIAGL CVFIISTLAI DFIDEGRLLL E FDLLIFSF GQLPLALVTW VPMFLSTLLA PYQALRLWAR GTWTQATGLG CALLAAHAVV LCALPVHVAV EHQLPPASRC VL VFEQVRF LMKSYSFLRE AVPGILRARR GEGIQAPSFS SYLYFLFCPT LIYRETYPRT PYVRWNYVAK NFAQALGCVL YAC FILGRL CVPVFANMSR EPFSTRALVL SILHATLPGI FMLLLIFFAF LHCWLNAFAE MLRFGDRMFY RDWWNSTSFS NYYR TWNVV VHDWLYSYVY QDGLRLLGAR ARGVAMLGVF LVSAVAHEYI FCFVLGFFYP VMLILFLVIG GMLNFMMHDQ RTGPA WNVL MWTMLFLGQG IQVSLYCQEW YARRHCPLPQ ATFWGLVTPR SWSCHT |
-分子 #2: (3S,4R,4aR,6S,6aS,12R,12aS,12bS)-4-[(acetyloxy)methyl]-12-hydroxy...
| 分子 | 名称: (3S,4R,4aR,6S,6aS,12R,12aS,12bS)-4-[(acetyloxy)methyl]-12-hydroxy-4,6a,12b-trimethyl-11-oxo-9-(pyridin-3-yl)-1,3,4,4a,5,6,6a,12,12a,12b-decahydro-2H,11H-naphtho[2,1-b]pyrano[3,4-e]pyran-3,6-diyl diacetate タイプ: ligand / ID: 2 / コピー数: 4 / 式: 7T8 |
|---|---|
| 分子量 | 理論値: 583.626 Da |
| Chemical component information | 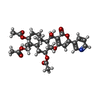 ChemComp-7T8: |
-分子 #3: CHOLESTEROL
| 分子 | 名称: CHOLESTEROL / タイプ: ligand / ID: 3 / コピー数: 8 / 式: CLR |
|---|---|
| 分子量 | 理論値: 386.654 Da |
| Chemical component information |  ChemComp-CLR: |
-分子 #4: OLEIC ACID
| 分子 | 名称: OLEIC ACID / タイプ: ligand / ID: 4 / コピー数: 4 / 式: OLA |
|---|---|
| 分子量 | 理論値: 282.461 Da |
| Chemical component information |  ChemComp-OLA: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: DARK FIELD |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 60.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION |
|---|---|
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.87 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 153208 |
 ムービー
ムービー コントローラー
コントローラー
















