+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20060 | |||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
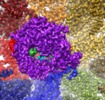
| タイトル | In situ structure of Rotavirus RNA-dependent RNA polymerase at transcript-elongated state | |||||||||||||||||||||||||||
 マップデータ マップデータ | em-volume_P1 | |||||||||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||||||||
 キーワード キーワード |  RNA-dependent RNA polymerase (RNA依存性RNAポリメラーゼ) / capsid shell protein / RNA-dependent RNA polymerase (RNA依存性RNAポリメラーゼ) / capsid shell protein /  transcription (転写 (生物学)) / in situ structure / transcription (転写 (生物学)) / in situ structure /  rotavirus (ロタウイルス) / transcriptional factors / rotavirus (ロタウイルス) / transcriptional factors /  reovirus / reovirus /  VIRUS (ウイルス) / VIRUS (ウイルス) /  viral protein-rna-transferase complex (ウイルス性) viral protein-rna-transferase complex (ウイルス性) | |||||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報T=2 icosahedral viral capsid / viral inner capsid / viral genome replication /  virion component / viral nucleocapsid / virion component / viral nucleocapsid /  RNA依存性RNAポリメラーゼ / RNA依存性RNAポリメラーゼ /  RNA-dependent RNA polymerase activity / RNA-dependent RNA polymerase activity /  nucleotide binding / DNA-templated transcription / nucleotide binding / DNA-templated transcription /  RNA binding RNA binding類似検索 - 分子機能 | |||||||||||||||||||||||||||
| 生物種 |   Rotavirus A (ロタウイルス A) Rotavirus A (ロタウイルス A) | |||||||||||||||||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.6 Å クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||||||||||||||||||||
 データ登録者 データ登録者 | Ding K / Chang T | |||||||||||||||||||||||||||
| 資金援助 |  米国, 8件 米国, 8件
| |||||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2019 ジャーナル: Nat Commun / 年: 2019タイトル: In situ structures of rotavirus polymerase in action and mechanism of mRNA transcription and release. 著者: Ke Ding / Cristina C Celma / Xing Zhang / Thomas Chang / Wesley Shen / Ivo Atanasov / Polly Roy / Z Hong Zhou /   要旨: Transcribing and replicating a double-stranded genome require protein modules to unwind, transcribe/replicate nucleic acid substrates, and release products. Here we present in situ cryo-electron ...Transcribing and replicating a double-stranded genome require protein modules to unwind, transcribe/replicate nucleic acid substrates, and release products. Here we present in situ cryo-electron microscopy structures of rotavirus dsRNA-dependent RNA polymerase (RdRp) in two states pertaining to transcription. In addition to the previously discovered universal "hand-shaped" polymerase core domain shared by DNA polymerases and telomerases, our results show the function of N- and C-terminal domains of RdRp: the former opens the genome duplex to isolate the template strand; the latter splits the emerging template-transcript hybrid, guides genome reannealing to form a transcription bubble, and opens a capsid shell protein (CSP) to release the transcript. These two "helicase" domains also extensively interact with CSP, which has a switchable N-terminal helix that, like cellular transcriptional factors, either inhibits or promotes RdRp activity. The in situ structures of RdRp, CSP, and RNA in action inform mechanisms of not only transcription, but also replication. | |||||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20060.map.gz emd_20060.map.gz | 65.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20060-v30.xml emd-20060-v30.xml emd-20060.xml emd-20060.xml | 17.6 KB 17.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_20060.png emd_20060.png | 350.1 KB | ||
| Filedesc metadata |  emd-20060.cif.gz emd-20060.cif.gz | 7.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20060 http://ftp.pdbj.org/pub/emdb/structures/EMD-20060 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20060 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20060 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20060.map.gz / 形式: CCP4 / 大きさ: 70.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20060.map.gz / 形式: CCP4 / 大きさ: 70.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | em-volume_P1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.34 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Rotavirus A
| 全体 | 名称:   Rotavirus A (ロタウイルス A) Rotavirus A (ロタウイルス A) |
|---|---|
| 要素 |
|
-超分子 #1: Rotavirus A
| 超分子 | 名称: Rotavirus A / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 / NCBI-ID: 28875 / 生物種: Rotavirus A / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|
-分子 #1: RNA (5'-R(P*UP*AP*UP*AP*UP*AP*UP*AP*UP*AP*UP*AP*UP*AP*UP*AP*UP*A)-3')
| 分子 | 名称: RNA (5'-R(P*UP*AP*UP*AP*UP*AP*UP*AP*UP*AP*UP*AP*UP*AP*UP*AP*UP*A)-3') タイプ: rna / ID: 1 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:   Rotavirus A (ロタウイルス A) Rotavirus A (ロタウイルス A) |
| 分子量 | 理論値: 5.673388 KDa |
| 配列 | 文字列: UAUAUAUAUA UAUAUAUA |
-分子 #2: RNA (5'-R(P*AP*UP*AP*UP*AP*UP*AP*UP*AP*UP*AP*UP*AP*UP*AP*UP*A)-3')
| 分子 | 名称: RNA (5'-R(P*AP*UP*AP*UP*AP*UP*AP*UP*AP*UP*AP*UP*AP*UP*AP*UP*A)-3') タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:   Rotavirus A (ロタウイルス A) Rotavirus A (ロタウイルス A) |
| 分子量 | 理論値: 5.367222 KDa |
| 配列 | 文字列: AUAUAUAUAU AUAUAUA |
-分子 #3: RNA-dependent RNA polymerase of rotavirus A
| 分子 | 名称: RNA-dependent RNA polymerase of rotavirus A / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO / EC番号:  RNA依存性RNAポリメラーゼ RNA依存性RNAポリメラーゼ |
|---|---|
| 由来(天然) | 生物種:   Rotavirus A (ロタウイルス A) Rotavirus A (ロタウイルス A) |
| 分子量 | 理論値: 125.276305 KDa |
| 配列 | 文字列: MGKYNLILSE YLSFIYNSQS AVQIPIYYSS NSELENRCIE FHSKCLENSK NGLSLKKLFV EYSDVIENAT LLSILSYSYD KYNAVERKL VKYAKGKPLE ADLTVNELDY ENNKITSELF PTAEEYTDLL MDPAILTSLS SNLNAVMFWL EKHENDVAEK L KIYKRRLD ...文字列: MGKYNLILSE YLSFIYNSQS AVQIPIYYSS NSELENRCIE FHSKCLENSK NGLSLKKLFV EYSDVIENAT LLSILSYSYD KYNAVERKL VKYAKGKPLE ADLTVNELDY ENNKITSELF PTAEEYTDLL MDPAILTSLS SNLNAVMFWL EKHENDVAEK L KIYKRRLD LFTIVASTVN KYGVPRHNAK YRYEYEVMKD KPYYLVTWAN SSIEMLMSVF SHEDYLIARE LIVLSYSNRS TL AKLVSSP MSILVALVDI NGTFITNEEL ELEFSNKYVR AIVPDQTFDE LKQMLDNMRK AGLTDIPKMI QDWLVDCSIE KFP LMAKIY SWSFHVGFRK QKMLDAALDQ LKTEYTEDVD DEMYREYTML IRDEVVKMLE EPVKHDDHLL QDSELAGLLS MSSA SNGES RQLKFGRKTI FSTKKNMHVM DDMANGRYTP GIIPPVNVDK PIPLGRRDVP GRRTRIIFIL PYEYFIAQHA VVEKM LIYA KHTREYAEFY SQSNQLLSYG DVTRFLSNNS MVLYTDVSQW DSSQHNTQPF RKGIIMGLDM LANMTNDARV IQTLNL YKQ TQINLMDSYV QIPDGNVIKK IQYGAVASGE KQTKAANSIA NLALIKTVLS RISNKYSFAT KIIRVDGDDN YAVLQFN TE VTKQMVQDVS NDVRETYARM NTKVKALVST VGIEIAKRYI AGGKIFFRAG INLLNNEKKG QSTQWDQAAV LYSNYIVN R LRGFETDREF ILTKIMQMTS VAITGSLRLF PSERVLTTNS TFKVFDSEDF IIEYGTTDDE VYIQRAFMSL SSQKSGIAD EIAASSTFKN YVSRLSEQLL FSKNNIVSRG IALTEKAKLN SYAPISLEKR RAQISALLTM LQKPVTFKSS KITINDILRD IKPFFTVNE AHLPIQYQKF MPTLPDNVQY IIQCIGSRTY QIEDDGSKSA ISRLISKYSV YKPSIEELYK VISLHENEIQ L YLISLGIP KIDADTYVGS KIYSQDKYRI LESYVYNLLS INYGCYQLFD FNSPDLEKLI RIPFKGKIPA VTFILHLYAK LE VINHAIK NGSWISLFCN YPKSEMIKLW KKMWNITSLR SPYTNANFFQ D UniProtKB:  RNA依存性RNAポリメラーゼ RNA依存性RNAポリメラーゼ |
-分子 #4: Inner capsid protein VP2
| 分子 | 名称: Inner capsid protein VP2 / タイプ: protein_or_peptide / ID: 4 / コピー数: 10 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Rotavirus A (ロタウイルス A) Rotavirus A (ロタウイルス A) |
| 分子量 | 理論値: 103.425992 KDa |
| 配列 | 文字列: MAYRKRGARR ETNLKQDDRM QEKEENKNVN TNSENKNATK PQLSEKVLSQ KEEVITDNQE EIKIADEVKK SNKEESKQLL EVLKTKEEH QKEVQYEILQ KTIPTFEPKE SILKKLEDIK PEQVKKQTKL FRIFEPRQLP VYRANGEKEL RNRWYWKLKR D TLPDGDYD ...文字列: MAYRKRGARR ETNLKQDDRM QEKEENKNVN TNSENKNATK PQLSEKVLSQ KEEVITDNQE EIKIADEVKK SNKEESKQLL EVLKTKEEH QKEVQYEILQ KTIPTFEPKE SILKKLEDIK PEQVKKQTKL FRIFEPRQLP VYRANGEKEL RNRWYWKLKR D TLPDGDYD VREYFLNLYD QVLTEMPDYL LLKDMAVENK NSRDAGKVVD SETAAICDAI FQDEETEGVV RRFIAEMRQR VQ ADRNVVN YPSILHPIDH AFNEYFLQHQ LVEPLNNDII FNYIPERIRN DVNYILNMDR NLPSTARYIR PNLLQDRLNL HDN FESLWD TITTSNYILA RSVVPDLKEL VSTEAQIQKM SQDLQLEALT IQSETQFLTG INSQAANDCF KTLIAAMLSQ RTMS LDFVT TNYMSLISGM WLLTVVPNDM FIRESLVACQ LAIINTIIYP AFGMQRMHYR NGDPQTPFQI AEQQIQNFQV ANWLH FVNN NQFRQVVIDG VLNQVLNDNI RNGHVVNQLM EALMQLSRQQ FPTMPVDYKR SIQRGILLLS NRLGQLVDLT RLLAYN YET LMACITMNMQ HVQTLTTEKL QLTSVTSLCM LIGNATVIPS PQTLFHYYNV NVNFHSNYNE RINDAVAIIT AANRLNL YQ KKMKSIVEDF LKRLQIFDIS RVPDDQMYRL RDRLRLLPVE IRRLDIFNLI LMNMEQIERA SDKIAQGVII AYRDMQLE R DEMYGYVNIA RNLDGFQQIN LEELMRTGDY AQITNMLLNN QPVALVGALP FITDSSVISL VAKLDATVFA QIVKLRKVD TLKPILYKIN SDSNDFYLVA NYDWVPTSTT KVYKQIPQQF DFRASMHMLT SNLTFTVYSD LLAFVSADTV EPINAVAFDN MRIMNEL UniProtKB: Inner capsid protein VP2 |
-分子 #5: URIDINE 5'-TRIPHOSPHATE
| 分子 | 名称: URIDINE 5'-TRIPHOSPHATE / タイプ: ligand / ID: 5 / コピー数: 1 / 式: UTP |
|---|---|
| 分子量 | 理論値: 484.141 Da |
| Chemical component information |  ChemComp-UTP: |
-分子 #6: GUANOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: GUANOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 6 / コピー数: 1 / 式: GTP |
|---|---|
| 分子量 | 理論値: 523.18 Da |
| Chemical component information |  ChemComp-GTP: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm Bright-field microscopy / Cs: 2.7 mm |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / 平均電子線量: 18.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER / 詳細: Gaussian Ball |
|---|---|
| 初期 角度割当 | タイプ: PROJECTION MATCHING / ソフトウェア - 名称: RELION (ver. 2.0) |
| 最終 3次元分類 | ソフトウェア - 名称: RELION (ver. 2.0) |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION (ver. 2.0) |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 3.6 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 2.0) / 使用した粒子像数: 411438 |
 ムービー
ムービー コントローラー
コントローラー