+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Upright KimA dimer with bound c-di-AMP from B. subtilis | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | potassium importer /  second messenger (セカンドメッセンジャー) / second messenger (セカンドメッセンジャー) /  c-di-AMP / c-di-AMP /  MEMBRANE PROTEIN (膜タンパク質) MEMBRANE PROTEIN (膜タンパク質) | |||||||||
| 機能・相同性 | Amino acid/polyamine transporter I /  Amino acid permease / Amino acid permease /  symporter activity / potassium ion transport / symporter activity / potassium ion transport /  metal ion binding / metal ion binding /  細胞膜 / Potassium transporter KimA 細胞膜 / Potassium transporter KimA 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Bacillus subtilis (枯草菌) Bacillus subtilis (枯草菌) | |||||||||
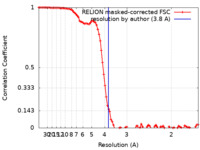
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.8 Å クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Vonck J / Wieferig JP | |||||||||
| 資金援助 |  ドイツ, 2件 ドイツ, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Cyclic di-AMP traps proton-coupled K transporters of the KUP family in an inward-occluded conformation. 著者: Michael F Fuss / Jan-Philip Wieferig / Robin A Corey / Yvonne Hellmich / Igor Tascón / Joana S Sousa / Phillip J Stansfeld / Janet Vonck / Inga Hänelt /    要旨: Cyclic di-AMP is the only known essential second messenger in bacteria and archaea, regulating different proteins indispensable for numerous physiological processes. In particular, it controls ...Cyclic di-AMP is the only known essential second messenger in bacteria and archaea, regulating different proteins indispensable for numerous physiological processes. In particular, it controls various potassium and osmolyte transporters involved in osmoregulation. In Bacillus subtilis, the K/H symporter KimA of the KUP family is inactivated by c-di-AMP. KimA sustains survival at potassium limitation at low external pH by mediating potassium ion uptake. However, at elevated intracellular K concentrations, further K accumulation would be toxic. In this study, we reveal the molecular basis of how c-di-AMP binding inhibits KimA. We report cryo-EM structures of KimA with bound c-di-AMP in detergent solution and reconstituted in amphipols. By combining structural data with functional assays and molecular dynamics simulations we reveal how c-di-AMP modulates transport. We show that an intracellular loop in the transmembrane domain interacts with c-di-AMP bound to the adjacent cytosolic domain. This reduces the mobility of transmembrane helices at the cytosolic side of the K binding site and therefore traps KimA in an inward-occluded conformation. #1:  ジャーナル: Biorxiv / 年: 2023 ジャーナル: Biorxiv / 年: 2023タイトル: Cyclic di-AMP traps proton-coupled K+ transporters of the KUP family in an inward-occluded conformation 著者: Fuss MF / Wieferig JP / Corey R / Hellmich Y / Tascon I / Sousa JS / Stansfeld P / Vonck J / Haenelt I | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15895.map.gz emd_15895.map.gz | 65.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15895-v30.xml emd-15895-v30.xml emd-15895.xml emd-15895.xml | 25.7 KB 25.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15895_fsc.xml emd_15895_fsc.xml | 9.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15895.png emd_15895.png | 74.8 KB | ||
| その他 |  emd_15895_additional_1.map.gz emd_15895_additional_1.map.gz emd_15895_additional_2.map.gz emd_15895_additional_2.map.gz emd_15895_additional_3.map.gz emd_15895_additional_3.map.gz emd_15895_additional_4.map.gz emd_15895_additional_4.map.gz emd_15895_half_map_1.map.gz emd_15895_half_map_1.map.gz emd_15895_half_map_2.map.gz emd_15895_half_map_2.map.gz | 52.9 MB 54.7 MB 54.7 MB 65.6 MB 53.4 MB 53.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15895 http://ftp.pdbj.org/pub/emdb/structures/EMD-15895 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15895 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15895 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8b71MC  8b70C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15895.map.gz / 形式: CCP4 / 大きさ: 70.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15895.map.gz / 形式: CCP4 / 大きさ: 70.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.831 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: unsharpened map
| ファイル | emd_15895_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
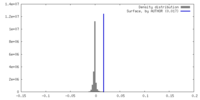
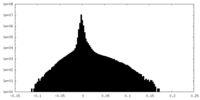
| 密度ヒストグラム |
-追加マップ: Halfmap2 focused refinement half-dimer
| ファイル | emd_15895_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Halfmap2 focused refinement half-dimer | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
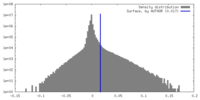
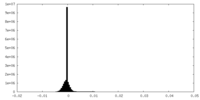
| 密度ヒストグラム |
-追加マップ: Halfmap1 focused refinement half-dimer
| ファイル | emd_15895_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Halfmap1 focused refinement half-dimer | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
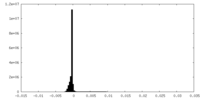
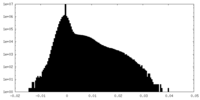
| 密度ヒストグラム |
-追加マップ: Focused refinement half-dimer
| ファイル | emd_15895_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Focused refinement half-dimer | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
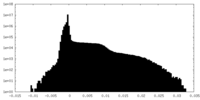
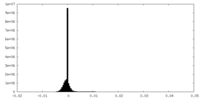
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_15895_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_15895_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Upright dimer KimA with c-di-AMP bound
| 全体 | 名称: Upright dimer KimA with c-di-AMP bound |
|---|---|
| 要素 |
|
-超分子 #1: Upright dimer KimA with c-di-AMP bound
| 超分子 | 名称: Upright dimer KimA with c-di-AMP bound / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Bacillus subtilis (枯草菌) Bacillus subtilis (枯草菌) |
| 分子量 | 理論値: 134 KDa |
-分子 #1: Potassium transporter KimA
| 分子 | 名称: Potassium transporter KimA / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Bacillus subtilis (枯草菌) Bacillus subtilis (枯草菌) |
| 分子量 | 理論値: 66.838977 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MYHSIKRFLI GKPLKSQAAG EQKLTKLKAL AMLSSDALSS VAYGTEQILI ILATISAAAF WYSIPIAVGV LILLLALILS YRQIIYAYP QGGGAYIVSK ENLGEKPGLI AGGSLLVDYI LTVAVSISAG TDAITSAFPA LHDYHVPIAI FLVLVIMILN L RGLSESAS ...文字列: MYHSIKRFLI GKPLKSQAAG EQKLTKLKAL AMLSSDALSS VAYGTEQILI ILATISAAAF WYSIPIAVGV LILLLALILS YRQIIYAYP QGGGAYIVSK ENLGEKPGLI AGGSLLVDYI LTVAVSISAG TDAITSAFPA LHDYHVPIAI FLVLVIMILN L RGLSESAS ILAYPVYLFV VALLVLIAVG LFKLMTGQID QPAHHTSLGT PVAGITLFLL LKAFSSGCSA LTGVEAISNA IP AFKNPPA RNAARTLAMM GILLAILFSG ITVLAYGYGT APKPDETVVS QIASETFGRN VFYYVIQGVT SLILVLAANT GFS AFPQLA FNLARDQYMP RMFTVRGDRL GFSNGIIFLG FASIVLIILF GGQTEHLIPL YAVGVFIPFT LSQTGMCMKW IKQK PKGWI GKMLINSCGA LISFMVLSIL FVTKFNVVWP VLIFMPIVVL LFFAIKNHYT AVGEQLRIVD KEPEEIKGTV VIVPV AGVT TVVQKSIHYA KSLSDQVIAV HVSFDREQEK KFEKRWEELN NGVRLVTLHS SYRSLVHPFD KFLETVEAKA KKEQFS VMV LFPQFITKKR WHTILHNQSA FLLRVRLFWK KDIMVATLPY HFKK |
-分子 #2: (2R,3R,3aS,5R,7aR,9R,10R,10aS,12R,14aR)-2,9-bis(6-amino-9H-purin-...
| 分子 | 名称: (2R,3R,3aS,5R,7aR,9R,10R,10aS,12R,14aR)-2,9-bis(6-amino-9H-purin-9-yl)octahydro-2H,7H-difuro[3,2-d:3',2'-j][1,3,7,9,2,8 ]tetraoxadiphosphacyclododecine-3,5,10,12-tetrol 5,12-dioxide タイプ: ligand / ID: 2 / コピー数: 2 / 式: 2BA |
|---|---|
| 分子量 | 理論値: 658.412 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.25 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE |
| 詳細 | Reconstituted in amphipols PMAL C8 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.1 µm Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.1 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 55.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 得られたモデル |  PDB-8b71: |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)