+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Enterococcus faecalis EfrCD in complex with a nanobody | ||||||||||||
 マップデータ マップデータ | EfrCD volume | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード |  ABC transporter / ABC transporter /  Nanobody (ナノボディ) / Nanobody (ナノボディ) /  Enterococcus faecalis (エンテロコッカス・ファエカリス) / Enterococcus faecalis (エンテロコッカス・ファエカリス) /  MEMBRANE PROTEIN (膜タンパク質) MEMBRANE PROTEIN (膜タンパク質) | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |   Enterococcus faecalis (乳酸球菌) / Enterococcus faecalis (乳酸球菌) /   Vicugna pacos (アルパカ) Vicugna pacos (アルパカ) | ||||||||||||
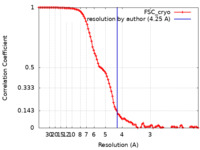
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.25 Å クライオ電子顕微鏡法 / 解像度: 4.25 Å | ||||||||||||
 データ登録者 データ登録者 | Ehrenbolger K / Hutter CAJ / Meier G / Seeger MA / Barandun J | ||||||||||||
| 資金援助 |  スウェーデン, スウェーデン,  スイス, 3件 スイス, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Chem Biol / 年: 2023 ジャーナル: Nat Chem Biol / 年: 2023タイトル: Deep mutational scan of a drug efflux pump reveals its structure-function landscape. 著者: Gianmarco Meier / Sujani Thavarasah / Kai Ehrenbolger / Cedric A J Hutter / Lea M Hürlimann / Jonas Barandun / Markus A Seeger /   要旨: Drug efflux is a common resistance mechanism found in bacteria and cancer cells, but studies providing comprehensive functional insights are scarce. In this study, we performed deep mutational ...Drug efflux is a common resistance mechanism found in bacteria and cancer cells, but studies providing comprehensive functional insights are scarce. In this study, we performed deep mutational scanning (DMS) on the bacterial ABC transporter EfrCD to determine the drug efflux activity profile of more than 1,430 single variants. These systematic measurements revealed that the introduction of negative charges at different locations within the large substrate binding pocket results in strongly increased efflux activity toward positively charged ethidium, whereas additional aromatic residues did not display the same effect. Data analysis in the context of an inward-facing cryogenic electron microscopy structure of EfrCD uncovered a high-affinity binding site, which releases bound drugs through a peristaltic transport mechanism as the transporter transits to its outward-facing conformation. Finally, we identified substitutions resulting in rapid Hoechst influx without affecting the efflux activity for ethidium and daunorubicin. Hence, single mutations can convert EfrCD into a drug-specific ABC importer. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12816.map.gz emd_12816.map.gz | 93.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12816-v30.xml emd-12816-v30.xml emd-12816.xml emd-12816.xml | 21.1 KB 21.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12816_fsc.xml emd_12816_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12816.png emd_12816.png | 98.7 KB | ||
| その他 |  emd_12816_half_map_1.map.gz emd_12816_half_map_1.map.gz emd_12816_half_map_2.map.gz emd_12816_half_map_2.map.gz | 91.9 MB 91.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12816 http://ftp.pdbj.org/pub/emdb/structures/EMD-12816 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12816 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12816 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7ocyMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12816.map.gz / 形式: CCP4 / 大きさ: 98.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12816.map.gz / 形式: CCP4 / 大きさ: 98.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EfrCD volume | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.04 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: EfrCD half map2
| ファイル | emd_12816_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EfrCD half map2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: EfrCD half map1
| ファイル | emd_12816_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EfrCD half map1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : EfrCD in complex with a nanobody
| 全体 | 名称: EfrCD in complex with a nanobody |
|---|---|
| 要素 |
|
-超分子 #1: EfrCD in complex with a nanobody
| 超分子 | 名称: EfrCD in complex with a nanobody / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|
-超分子 #2: Enterococcus faecalis
| 超分子 | 名称: Enterococcus faecalis / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Enterococcus faecalis (乳酸球菌) Enterococcus faecalis (乳酸球菌) |
-超分子 #3: nanobody
| 超分子 | 名称: nanobody / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #3 |
|---|---|
| 由来(天然) | 生物種:   Vicugna pacos (アルパカ) Vicugna pacos (アルパカ) |
-分子 #1: ABC transporter ATP-binding protein
| 分子 | 名称: ABC transporter ATP-binding protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Enterococcus faecalis (乳酸球菌) Enterococcus faecalis (乳酸球菌) |
| 分子量 | 理論値: 62.905289 KDa |
| 組換発現 | 生物種:   Lactococcus lactis (乳酸菌) Lactococcus lactis (乳酸菌) |
| 配列 | 文字列: MDLIIQHAKK YKGSVVIALL AVIVMVVSAL WQPKLLQQVL EAIMNDDSDK MKNLGIQLIA IAGLGLVAGV INTIFSAKVA QGVSADIRE ATFRKIQTFS FGNIEKFSAG NLVVRLTNDV TQIQNVIMIA LQTLFRIPFL FIGSFILAML TLPQLWWVIV A LVIAVILI ...文字列: MDLIIQHAKK YKGSVVIALL AVIVMVVSAL WQPKLLQQVL EAIMNDDSDK MKNLGIQLIA IAGLGLVAGV INTIFSAKVA QGVSADIRE ATFRKIQTFS FGNIEKFSAG NLVVRLTNDV TQIQNVIMIA LQTLFRIPFL FIGSFILAML TLPQLWWVIV A LVIAVILI SMLSFSQMGK HFMIIQNLID KINGIAKENL LGIRVVKSFV QEKNQLSRFT KVSEELTTHN LIVGSLFAVM IP AFMLVAN LAVVGSIFFV SNLVKDDPTL IGGVASFMNY LMQIMMAIII GGMMMMMTSR AAVSIKRIKE VMETEPDVTY KKV PEQELI GSVEFDHVSF RYPGDEEDTL KDISFSIQPG EMIGIVGATG AGKSTLAQLI PRLFDPTEGK IEVGGVDLRE VNEH SLRKT VSFVLQKAIL FSGTIAQNLR HGKRDASEAD MERASGIAQA KEFIEKLAEG YDAPVEERSN NFSGGQKQRL SITRG VIGE PKILILDDST SALDARSERL VREALDKELK ETTTIVIAQK ISSVVHADRI LVLDNGRLVG EGTHEELAAT NPVYQE IYE TQKGKEEA |
-分子 #2: ABC transporter ATP-binding protein
| 分子 | 名称: ABC transporter ATP-binding protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Enterococcus faecalis (乳酸球菌) Enterococcus faecalis (乳酸球菌) |
| 分子量 | 理論値: 66.213734 KDa |
| 組換発現 | 生物種:   Lactococcus lactis (乳酸菌) Lactococcus lactis (乳酸菌) |
| 配列 | 文字列: MTDLIKASKF FYHYLKRYKV SFLFIFLAIF AATYLQVKAP QFVGEAIQEL AKYAVNVMQG KDDKSAFVSV IWKLLIFYVL TSAASFIYS ILFTQVVGKS TNRMRIGLFN KLEKLTIRFF DSHQDGEILS RFTSDLDNIQ NSLNQALLQV LTNIALLVGV L IMMFRQNV ...文字列: MTDLIKASKF FYHYLKRYKV SFLFIFLAIF AATYLQVKAP QFVGEAIQEL AKYAVNVMQG KDDKSAFVSV IWKLLIFYVL TSAASFIYS ILFTQVVGKS TNRMRIGLFN KLEKLTIRFF DSHQDGEILS RFTSDLDNIQ NSLNQALLQV LTNIALLVGV L IMMFRQNV ELAWATIAST PIAILIAVFV ISKARKYVDL QQDEVGKLNG YMDEKISGQR VIITNGLQEE TIDGFLEQNE KV RAATYKG QVYSGLLFPM MQGMSLVNTA IVIFFGGWLA INGSVDRAAA LGLVVMFVQY SQQYYQPLMQ ISSGYSMIQL AVT GARRLN EMFDEPDEIR PENGEKLEEI NKAVALNHVV FGYNPETPVL KDVSIHVDKG EMVALVGPTG SGKTTIMNLM NRFY DVNEG AVTFDGVDIR EMDLDSLRSH VGIVLQESVL FSGTIRENIA FGKPEATDEE IVQAAKQANI HEFIVNLEQG YDTEI TEEN NLFSTGQKQL VSIARTIITN PELLILDEAT SNVDTVTEAK IQKAMDEAIK GRTSFVIAHR LKTILNADRI IVLRDG EVI EEGNHHELVE QDGFYAELYK NQFVFE |
-分子 #3: Nanobody
| 分子 | 名称: Nanobody / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Vicugna pacos (アルパカ) Vicugna pacos (アルパカ) |
| 分子量 | 理論値: 12.718138 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21 (大腸菌) Escherichia coli BL21 (大腸菌) |
| 配列 | 文字列: GPSQLQLVES GGGLVQAGDT LRLSCEASRS FNRMGWYRQA PGKQRDMVAH IFSDGRTRYA DSVQGRFTIS RDNAKNTVYL QMNNLKPED TAVYYCNGFF IQDFWGQGTP VTVSA |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 8 mg/mL | ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. | ||||||||
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK IV 詳細: blot force of -5, waiting time of 1 second, blot time of 4.5 seconds at 4C. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm Bright-field microscopy / Cs: 2.7 mm最大 デフォーカス(公称値): 3.3000000000000003 µm 最小 デフォーカス(公称値): 1.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 実像数: 3135 / 平均電子線量: 52.1 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X