+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of a CRISPR effector in complex with regulator | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Desulfonema magnum (バクテリア) Desulfonema magnum (バクテリア) | |||||||||
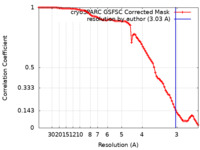
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.03 Å クライオ電子顕微鏡法 / 解像度: 3.03 Å | |||||||||
 データ登録者 データ登録者 | Babatunde EE / Davide T / Bertrand B / Sergey N / Alexander M / Henning S / Dong CN | |||||||||
| 資金援助 |  スイス, 2件 スイス, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2023 ジャーナル: Nat Struct Mol Biol / 年: 2023タイトル: Structural insights into the regulation of Cas7-11 by TPR-CHAT. 著者: Babatunde Ekundayo / Davide Torre / Bertrand Beckert / Sergey Nazarov / Alexander Myasnikov / Henning Stahlberg / Dongchun Ni /  要旨: The CRISPR-guided caspase (Craspase) complex is an assembly of the target-specific RNA nuclease known as Cas7-11 bound to CRISPR RNA (crRNA) and an ancillary protein known as TPR-CHAT ...The CRISPR-guided caspase (Craspase) complex is an assembly of the target-specific RNA nuclease known as Cas7-11 bound to CRISPR RNA (crRNA) and an ancillary protein known as TPR-CHAT (tetratricopeptide repeats (TPR) fused with a CHAT domain). The Craspase complex holds promise as a tool for gene therapy and biomedical research, but its regulation is poorly understood. TPR-CHAT regulates Cas7-11 nuclease activity via an unknown mechanism. In the present study, we use cryoelectron microscopy to determine structures of the Desulfonema magnum (Dm) Craspase complex to gain mechanistic insights into its regulation. We show that DmTPR-CHAT stabilizes crRNA-bound DmCas7-11 in a closed conformation via a network of interactions mediated by the DmTPR-CHAT N-terminal domain, the DmCas7-11 insertion finger and Cas11-like domain, resulting in reduced target RNA accessibility and cleavage. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
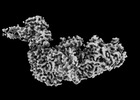
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14847.map.gz emd_14847.map.gz | 63.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14847-v30.xml emd-14847-v30.xml emd-14847.xml emd-14847.xml | 25.8 KB 25.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14847_fsc.xml emd_14847_fsc.xml | 8.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14847.png emd_14847.png | 63.6 KB | ||
| その他 |  emd_14847_additional_1.map.gz emd_14847_additional_1.map.gz emd_14847_additional_2.map.gz emd_14847_additional_2.map.gz emd_14847_additional_3.map.gz emd_14847_additional_3.map.gz emd_14847_half_map_1.map.gz emd_14847_half_map_1.map.gz emd_14847_half_map_2.map.gz emd_14847_half_map_2.map.gz | 34 MB 63.4 MB 63.3 MB 62.3 MB 62.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14847 http://ftp.pdbj.org/pub/emdb/structures/EMD-14847 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14847 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14847 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14847.map.gz / 形式: CCP4 / 大きさ: 67 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14847.map.gz / 形式: CCP4 / 大きさ: 67 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.2769 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #3
| ファイル | emd_14847_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
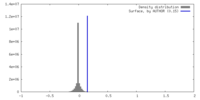
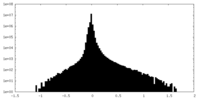
| 密度ヒストグラム |
-追加マップ: #2
| ファイル | emd_14847_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
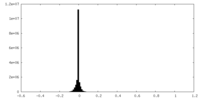
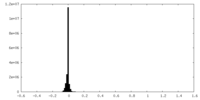
| 密度ヒストグラム |
-追加マップ: #1
| ファイル | emd_14847_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
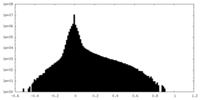
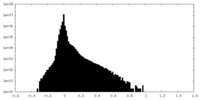
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_14847_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
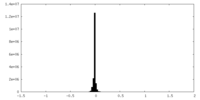
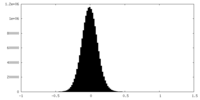
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_14847_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cas7-11 with TRP-CHAT-NTD
| 全体 | 名称: Cas7-11 with TRP-CHAT-NTD |
|---|---|
| 要素 |
|
-超分子 #1: Cas7-11 with TRP-CHAT-NTD
| 超分子 | 名称: Cas7-11 with TRP-CHAT-NTD / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  Desulfonema magnum (バクテリア) Desulfonema magnum (バクテリア) |
| 分子量 | 理論値: 220 KDa |
-分子 #1: Cas7-11
| 分子 | 名称: Cas7-11 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Desulfonema magnum (バクテリア) Desulfonema magnum (バクテリア) |
| 分子量 | 理論値: 207.402859 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MGSSHHHHHH SQDPMQHIIP LTITFLESFR VIEWHKSSDR NSLRFLRGYA YARWHRSLKN NKGRPYITGT LVRSAIIRAA EELLWLNDG NYKGAICCPG EFNGSNAKVF REGKARRLRR RQTLTWPTKC ACHEKEPCPF CLLLGRYEKS SKTSKSPVLN N VNFSNFNV ...文字列: MGSSHHHHHH SQDPMQHIIP LTITFLESFR VIEWHKSSDR NSLRFLRGYA YARWHRSLKN NKGRPYITGT LVRSAIIRAA EELLWLNDG NYKGAICCPG EFNGSNAKVF REGKARRLRR RQTLTWPTKC ACHEKEPCPF CLLLGRYEKS SKTSKSPVLN N VNFSNFNV LGKEKEFLNI EDVADMRVVN RVDQQSGKAE DFFNIWEITD GAWKIFRGEI QVSDKGWSDE ENFSKFLTLL KG ASVLVDK ISGGLCYLTL DKPELAELPA VKTEKDVLEP GDDASVLEIS RPPYWNNMLN LAGTISEAFE REDKLVHLRL FAD TVRELR RSDIETLDLP KGHADRLGKP SDHFIWDIEI NKKVKLRNWL FWIFNEFRDT YAYFDWRTFC EALGQALYLE AKKQ VPNQF SSERPVGATP AMEVKTPEHD PGRAAQGPRY EWLIKGELVS QTPFFFGWST EADNREHTNL KLLAARDGRL RLPLS VLRG ILRRDIKVVL DNKCRAELAM KQPCSCPVCN LMKKITIRDS FSSNYAEPPK IRHKIRLDPK SGTVAKGALF DSEVGP RGV VFPFELRLRS ADDTLPQALK TVFSWWQQGT VSFSGDAGTG KGIFCLRNLK SIRWDLKTEM DKYAATLGGR KTPVGKW DK CHIPDDKTYP WVKETVEISV CSPFITKDPV NSLIDSAGYD AICYTTVDLE KSENINSLPE SEISLLFEMF DLQYPMSY L FPNKDIYLLK GESFRGMLRT AVGRGENLLL REHEDCSCTL CRIFGNEHNA GKIRVEDFII QGEPRTKLVD RVAIDRFTA GAKDKFKFDA APIVGTPTNK LKFRGNIWIH RDLDGLACES LKLALEDIEN GLYPFGGLGN AGFGWVNYNP LSHPAQEENK ADFSLTKKM ELNWSMKELS TDKIYWPHYF LPFGKKVLRE KTPPSHACID ENEDSELYSG KIVCTLETRT PLIIPDSEFQ G EEHKSYDF FNLNGELCIP GSEIRGMISS VFEALTNSCM RIFDEKKRLS WRMNPNKKDK KNNNRRELDD FIPGRVTNDR KM EEMKEYR YPFYDQAITA NDKQNKYFDQ WEATIELTDE SLEKLKAEKI LQSVLDALRP LTKKKEKYKN TETFVFDLKK FIG KIDDNQ QMISEALERG KVRLTGNSLK QISKTKKIPR QILNQLKGLK DNTYENREQF ISVLKTTVRG INDEQISLIL DNID EDVRL TDLSLTKIRK AKVPQTLVDM LADLKKDEPY KNEKEFLSEF KKKMGEIIGL ILKHAAKTGG DVPRYNHPTP TDKML LSLA AYNRNHKHEN GKAEYRIVKP KHNLKVDFMF AVTPFENPFK GYNPAAVVEE PVGGYLKVSG PNKIEKVKKV NPNSVS VRD DKNQEIIHNG VYLRKITVAN AKSKNKLRER LVPEFAWYDK DSEAAYAMTK RCERVFVEIG AKPIPIQPSA REKFKIL TQ EYQKNAKQQK TPEAFQTILP KDGELRPGDL VYFREDKKTN TVTDIIPVRI SRTVDDEVLA RKIPDDVGDV RPCVREIL D KEKQKEIADA GVKEVFQHHP DGLCPACSLF GTTFYKGRIA FGFAFHKDKD PELANNGKHI TLPLLERPRP TWSMPKKES RVPGRKFYVH HQGWERVIKH SNLDESDPNA TKQTVNNRSV QAIKEEQKFQ FEVRFENLRE WELGLLVYVL QLEPQFAHKL GMGKALGFG SVRIRVGEIH SGPKELDESR LVSSAMKKME EIWDRDKNVL EKLFRLLYFN ESKDIKVRYP KLQKEKEEEE E ESGYMELA KEEYQPEQRR NKLTNPWEGW GNILKKPAIL I |
-分子 #3: TPR-CHAT
| 分子 | 名称: TPR-CHAT / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Desulfonema magnum (バクテリア) Desulfonema magnum (バクテリア) |
| 分子量 | 理論値: 17.361045 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MSSAFSGLKI PELSVDPAEV FKSDNPQLVS VLLDEFELQE QRPFFSGLIP EKQINIALKK SPQLKKLACH LLEAYEINGR RWKHADRRR VLEKAIRLLE KVSNELKGDI QKLENNVKES GKDSEELNKT REKHGEILAD MGRAYLHRAK II |
-分子 #2: gRNA
| 分子 | 名称: gRNA / タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Desulfonema magnum (バクテリア) Desulfonema magnum (バクテリア) |
| 分子量 | 理論値: 12.344251 KDa |
| 配列 | 文字列: UAUGUGAUGG AACCUCUCCG GAUAAUUCUA UCUCUUCUG |
-分子 #4: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 4 / コピー数: 4 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.9 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 96000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 96000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 60.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 温度因子: 43 |
|---|---|
| 得られたモデル |  PDB-7zol: |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)