+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of ubiquitinated FANCI in complex with FANCD2 and double-stranded DNA | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of CD40 signaling pathway / regulation of regulatory T cell differentiation / homologous chromosome pairing at meiosis / double-strand break repair involved in meiotic recombination / gamete generation / neuronal stem cell population maintenance / brain morphogenesis /  DNA repair complex / mitotic intra-S DNA damage checkpoint signaling / Peptide chain elongation ...regulation of CD40 signaling pathway / regulation of regulatory T cell differentiation / homologous chromosome pairing at meiosis / double-strand break repair involved in meiotic recombination / gamete generation / neuronal stem cell population maintenance / brain morphogenesis / DNA repair complex / mitotic intra-S DNA damage checkpoint signaling / Peptide chain elongation ...regulation of CD40 signaling pathway / regulation of regulatory T cell differentiation / homologous chromosome pairing at meiosis / double-strand break repair involved in meiotic recombination / gamete generation / neuronal stem cell population maintenance / brain morphogenesis /  DNA repair complex / mitotic intra-S DNA damage checkpoint signaling / Peptide chain elongation / Selenocysteine synthesis / Formation of a pool of free 40S subunits / Eukaryotic Translation Termination / Response of EIF2AK4 (GCN2) to amino acid deficiency / SRP-dependent cotranslational protein targeting to membrane / Viral mRNA Translation / Nonsense Mediated Decay (NMD) independent of the Exon Junction Complex (EJC) / GTP hydrolysis and joining of the 60S ribosomal subunit / L13a-mediated translational silencing of Ceruloplasmin expression / Major pathway of rRNA processing in the nucleolus and cytosol / Nonsense Mediated Decay (NMD) enhanced by the Exon Junction Complex (EJC) / interstrand cross-link repair / DNA repair complex / mitotic intra-S DNA damage checkpoint signaling / Peptide chain elongation / Selenocysteine synthesis / Formation of a pool of free 40S subunits / Eukaryotic Translation Termination / Response of EIF2AK4 (GCN2) to amino acid deficiency / SRP-dependent cotranslational protein targeting to membrane / Viral mRNA Translation / Nonsense Mediated Decay (NMD) independent of the Exon Junction Complex (EJC) / GTP hydrolysis and joining of the 60S ribosomal subunit / L13a-mediated translational silencing of Ceruloplasmin expression / Major pathway of rRNA processing in the nucleolus and cytosol / Nonsense Mediated Decay (NMD) enhanced by the Exon Junction Complex (EJC) / interstrand cross-link repair /  DNA polymerase binding / condensed chromosome / Maturation of protein E / Maturation of protein E / ER Quality Control Compartment (ERQC) / Myoclonic epilepsy of Lafora / FLT3 signaling by CBL mutants / Prevention of phagosomal-lysosomal fusion / IRAK2 mediated activation of TAK1 complex / Alpha-protein kinase 1 signaling pathway / DNA polymerase binding / condensed chromosome / Maturation of protein E / Maturation of protein E / ER Quality Control Compartment (ERQC) / Myoclonic epilepsy of Lafora / FLT3 signaling by CBL mutants / Prevention of phagosomal-lysosomal fusion / IRAK2 mediated activation of TAK1 complex / Alpha-protein kinase 1 signaling pathway /  グリコーゲン合成 / IRAK1 recruits IKK complex / IRAK1 recruits IKK complex upon TLR7/8 or 9 stimulation / Membrane binding and targetting of GAG proteins / Constitutive Signaling by NOTCH1 HD Domain Mutants / Endosomal Sorting Complex Required For Transport (ESCRT) / NOTCH2 Activation and Transmission of Signal to the Nucleus / IRAK2 mediated activation of TAK1 complex upon TLR7/8 or 9 stimulation / PTK6 Regulates RTKs and Their Effectors AKT1 and DOK1 / Negative regulation of FLT3 / Regulation of FZD by ubiquitination / TICAM1,TRAF6-dependent induction of TAK1 complex / TICAM1-dependent activation of IRF3/IRF7 / APC/C:Cdc20 mediated degradation of Cyclin B / Downregulation of ERBB4 signaling / p75NTR recruits signalling complexes / TRAF6 mediated IRF7 activation in TLR7/8 or 9 signaling / APC-Cdc20 mediated degradation of Nek2A / PINK1-PRKN Mediated Mitophagy / TRAF6-mediated induction of TAK1 complex within TLR4 complex / InlA-mediated entry of Listeria monocytogenes into host cells / Pexophagy / Regulation of innate immune responses to cytosolic DNA / VLDLR internalisation and degradation / Downregulation of ERBB2:ERBB3 signaling / NRIF signals cell death from the nucleus / Activated NOTCH1 Transmits Signal to the Nucleus / Translesion synthesis by REV1 / NF-kB is activated and signals survival / Regulation of PTEN localization / Translesion synthesis by POLK / グリコーゲン合成 / IRAK1 recruits IKK complex / IRAK1 recruits IKK complex upon TLR7/8 or 9 stimulation / Membrane binding and targetting of GAG proteins / Constitutive Signaling by NOTCH1 HD Domain Mutants / Endosomal Sorting Complex Required For Transport (ESCRT) / NOTCH2 Activation and Transmission of Signal to the Nucleus / IRAK2 mediated activation of TAK1 complex upon TLR7/8 or 9 stimulation / PTK6 Regulates RTKs and Their Effectors AKT1 and DOK1 / Negative regulation of FLT3 / Regulation of FZD by ubiquitination / TICAM1,TRAF6-dependent induction of TAK1 complex / TICAM1-dependent activation of IRF3/IRF7 / APC/C:Cdc20 mediated degradation of Cyclin B / Downregulation of ERBB4 signaling / p75NTR recruits signalling complexes / TRAF6 mediated IRF7 activation in TLR7/8 or 9 signaling / APC-Cdc20 mediated degradation of Nek2A / PINK1-PRKN Mediated Mitophagy / TRAF6-mediated induction of TAK1 complex within TLR4 complex / InlA-mediated entry of Listeria monocytogenes into host cells / Pexophagy / Regulation of innate immune responses to cytosolic DNA / VLDLR internalisation and degradation / Downregulation of ERBB2:ERBB3 signaling / NRIF signals cell death from the nucleus / Activated NOTCH1 Transmits Signal to the Nucleus / Translesion synthesis by REV1 / NF-kB is activated and signals survival / Regulation of PTEN localization / Translesion synthesis by POLK /  Regulation of BACH1 activity / Synthesis of active ubiquitin: roles of E1 and E2 enzymes / Translesion synthesis by POLI / Gap-filling DNA repair synthesis and ligation in GG-NER / MAP3K8 (TPL2)-dependent MAPK1/3 activation / TICAM1, RIP1-mediated IKK complex recruitment / cytosolic ribosome / Downregulation of TGF-beta receptor signaling / Josephin domain DUBs / Activation of IRF3, IRF7 mediated by TBK1, IKKε (IKBKE) / Regulation of activated PAK-2p34 by proteasome mediated degradation / InlB-mediated entry of Listeria monocytogenes into host cell / IKK complex recruitment mediated by RIP1 / JNK (c-Jun kinases) phosphorylation and activation mediated by activated human TAK1 / TGF-beta receptor signaling in EMT (epithelial to mesenchymal transition) / N-glycan trimming in the ER and Calnexin/Calreticulin cycle / Autodegradation of Cdh1 by Cdh1:APC/C / TNFR1-induced NF-kappa-B signaling pathway / APC/C:Cdc20 mediated degradation of Securin / positive regulation of protein ubiquitination / Asymmetric localization of PCP proteins / TCF dependent signaling in response to WNT / SCF-beta-TrCP mediated degradation of Emi1 / Regulation of NF-kappa B signaling / NIK-->noncanonical NF-kB signaling / Ubiquitin-dependent degradation of Cyclin D / AUF1 (hnRNP D0) binds and destabilizes mRNA / Negative regulators of DDX58/IFIH1 signaling / TNFR2 non-canonical NF-kB pathway / NOTCH3 Activation and Transmission of Signal to the Nucleus / activated TAK1 mediates p38 MAPK activation / Assembly of the pre-replicative complex / Vpu mediated degradation of CD4 / Deactivation of the beta-catenin transactivating complex / Degradation of DVL / Ubiquitin Mediated Degradation of Phosphorylated Cdc25A / Recognition of DNA damage by PCNA-containing replication complex Regulation of BACH1 activity / Synthesis of active ubiquitin: roles of E1 and E2 enzymes / Translesion synthesis by POLI / Gap-filling DNA repair synthesis and ligation in GG-NER / MAP3K8 (TPL2)-dependent MAPK1/3 activation / TICAM1, RIP1-mediated IKK complex recruitment / cytosolic ribosome / Downregulation of TGF-beta receptor signaling / Josephin domain DUBs / Activation of IRF3, IRF7 mediated by TBK1, IKKε (IKBKE) / Regulation of activated PAK-2p34 by proteasome mediated degradation / InlB-mediated entry of Listeria monocytogenes into host cell / IKK complex recruitment mediated by RIP1 / JNK (c-Jun kinases) phosphorylation and activation mediated by activated human TAK1 / TGF-beta receptor signaling in EMT (epithelial to mesenchymal transition) / N-glycan trimming in the ER and Calnexin/Calreticulin cycle / Autodegradation of Cdh1 by Cdh1:APC/C / TNFR1-induced NF-kappa-B signaling pathway / APC/C:Cdc20 mediated degradation of Securin / positive regulation of protein ubiquitination / Asymmetric localization of PCP proteins / TCF dependent signaling in response to WNT / SCF-beta-TrCP mediated degradation of Emi1 / Regulation of NF-kappa B signaling / NIK-->noncanonical NF-kB signaling / Ubiquitin-dependent degradation of Cyclin D / AUF1 (hnRNP D0) binds and destabilizes mRNA / Negative regulators of DDX58/IFIH1 signaling / TNFR2 non-canonical NF-kB pathway / NOTCH3 Activation and Transmission of Signal to the Nucleus / activated TAK1 mediates p38 MAPK activation / Assembly of the pre-replicative complex / Vpu mediated degradation of CD4 / Deactivation of the beta-catenin transactivating complex / Degradation of DVL / Ubiquitin Mediated Degradation of Phosphorylated Cdc25A / Recognition of DNA damage by PCNA-containing replication complex類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) / synthetic construct (人工物) Homo sapiens (ヒト) / synthetic construct (人工物) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.14 Å クライオ電子顕微鏡法 / 解像度: 4.14 Å | |||||||||
 データ登録者 データ登録者 | Lemonidis K / Rennie ML / Arkinson C / Streetley J / Clarke M / Chaugule VK / Walden H | |||||||||
| 資金援助 | European Union, 1件
| |||||||||
 引用 引用 |  ジャーナル: EMBO J / 年: 2023 ジャーナル: EMBO J / 年: 2023タイトル: Structural and biochemical basis of interdependent FANCI-FANCD2 ubiquitination. 著者: Kimon Lemonidis / Martin L Rennie / Connor Arkinson / Viduth K Chaugule / Mairi Clarke / James Streetley / Helen Walden /  要旨: Di-monoubiquitination of the FANCI-FANCD2 (ID2) complex is a central and crucial step for the repair of DNA interstrand crosslinks via the Fanconi anaemia pathway. While FANCD2 ubiquitination ...Di-monoubiquitination of the FANCI-FANCD2 (ID2) complex is a central and crucial step for the repair of DNA interstrand crosslinks via the Fanconi anaemia pathway. While FANCD2 ubiquitination precedes FANCI ubiquitination, FANCD2 is also deubiquitinated at a faster rate than FANCI, which can result in a FANCI-ubiquitinated ID2 complex (I D2). Here, we present a 4.1 Å cryo-EM structure of I D2 complex bound to double-stranded DNA. We show that this complex, like ID2 and I D2 , is also in the closed ID2 conformation and clamps on DNA. The target lysine of FANCD2 (K561) becomes fully exposed in the I D2-DNA structure and is thus primed for ubiquitination. Similarly, FANCI's target lysine (K523) is also primed for ubiquitination in the ID2 -DNA complex. The I D2-DNA complex exhibits deubiquitination resistance, conferred by the presence of DNA and FANCD2. ID2 -DNA, on the other hand, can be efficiently deubiquitinated by USP1-UAF1, unless further ubiquitination on FANCI occurs. Therefore, FANCI ubiquitination effectively maintains FANCD2 ubiquitination in two ways: it prevents excessive FANCD2 deubiquitination within an I D2 -DNA complex, and it enables re-ubiquitination of FANCD2 within a transient, closed-on-DNA, I D2 complex. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14694.map.gz emd_14694.map.gz | 4.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14694-v30.xml emd-14694-v30.xml emd-14694.xml emd-14694.xml | 23.3 KB 23.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14694_fsc.xml emd_14694_fsc.xml | 12 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14694.png emd_14694.png | 151 KB | ||
| その他 |  emd_14694_additional_1.map.gz emd_14694_additional_1.map.gz emd_14694_half_map_1.map.gz emd_14694_half_map_1.map.gz emd_14694_half_map_2.map.gz emd_14694_half_map_2.map.gz | 115.9 MB 116.1 MB 116.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14694 http://ftp.pdbj.org/pub/emdb/structures/EMD-14694 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14694 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14694 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7zf1MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14694.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14694.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.023 Å | ||||||||||||||||||||||||||||||||||||
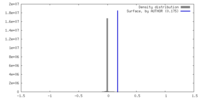
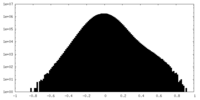
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_14694_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
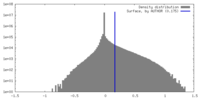
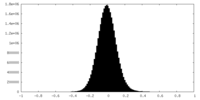
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_14694_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
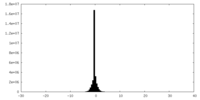
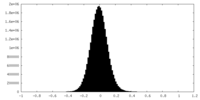
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_14694_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
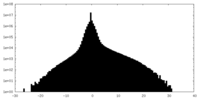
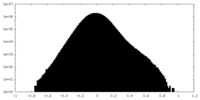
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of ubiquitinated FANCI with FANCD2 and double-stranded DNA.
| 全体 | 名称: Complex of ubiquitinated FANCI with FANCD2 and double-stranded DNA. |
|---|---|
| 要素 |
|
-超分子 #1: Complex of ubiquitinated FANCI with FANCD2 and double-stranded DNA.
| 超分子 | 名称: Complex of ubiquitinated FANCI with FANCD2 and double-stranded DNA. タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Fanconi anemia group I protein
| 分子 | 名称: Fanconi anemia group I protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 152.658594 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: MHHHHHHENL YFQGKPIPNP LLGLDSTMDQ KILSLAAEKT ADKLQEFLQT LREGDLTNLL QNQAVKGKVA GALLRAIFKG SPCSEEAGT LRRRKIYTCC IQLVESGDLQ KEIASEIIGL LMLEAHHFPG PLLVELANEF ISAVREGSLV NGKSLELLPI I LTALATKK ...文字列: MHHHHHHENL YFQGKPIPNP LLGLDSTMDQ KILSLAAEKT ADKLQEFLQT LREGDLTNLL QNQAVKGKVA GALLRAIFKG SPCSEEAGT LRRRKIYTCC IQLVESGDLQ KEIASEIIGL LMLEAHHFPG PLLVELANEF ISAVREGSLV NGKSLELLPI I LTALATKK ENLAYGKGVL SGEECKKQLI NTLCSGRWDQ QYVIQLTSMF KDVPLTAEEV EFVVEKALSM FSKMNLQEIP PL VYQLLVL SSKGSRKSVL EGIIAFFSAL DKQHNEEQSG DELLDVVTVP SGELRHVEGT IILHIVFAIK LDYELGRELV KHL KVGQQG DSNNNLSPFS IALLLSVTRI QRFQDQVLDL LKTSVVKSFK DLQLLQGSKF LQNLVPHRSY VSTMILEVVK NSVH SWDHV TQGLVELGFI LMDSYGPKKV LDGKTIETSP SLSRMPNQHA CKLGANILLE TFKIHEMIRQ EILEQVLNRV VTRAS SPIS HFLDLLSNIV MYAPLVLQSC SSKVTEAFDY LSFLPLQTVQ RLLKAVQPLL KVSMSMRDCL ILVLRKAMFA NQLDAR KSA VAGFLLLLKN FKVLGSLSSS QCSQSLSVSQ VHVDVHSHYN SVANETFCLE IMDSLRRCLS QQADVRLMLY EGFYDVL RR NSQLANSVMQ TLLSQLKQFY EPKPDLLPPL KLEACILTQG DKISLQEPLD YLLCCIQHCL AWYKNTVIPL QQGEEEEE E EEAFYEDLDD ILESITNRMI KSELEDFELD KSADFSQSTS IGIKNNICAF LVMGVCEVLI EYNFSISSFS KNRFEDILS LFMCYKKLSD ILNEKAGKAK TKMANKTSDS LLSMKFVSSL LTALFRDSIQ SHQESLSVLR SSNEFMRYAV NVALQKVQQL KETGHVSGP DGQNPEKIFQ NLCDITRVLL WRYTSIPTSV EESGKKEKGK SISLLCLEGL QKIFSAVQQF YQPKIQQFLR A LDVTDKEG EEREDADVSV TQRTAFQIRQ FQRSLLNLLS SQEEDFNSKE ALLLVTVLTS LSKLLEPSSP QFVQMLSWTS KI CKENSRE DALFCKSLMN LLFSLHVSYK SPVILLRDLS QDIHGHLGDI DQDVEVEKTN HFAIVNLRTA APTVCLLVLS QAE KVLEEV DWLITKLKGQ VSQETLSEEA SSQATLPNQP VEKAIIMQLG TLLTFFHELV QTALPSGSCV DTLLKDLCKM YTTL TALVR YYLQVCQSSG GIPKNMEKLV KLSGSHLTPL CYSFISYVQN KSKSLNYTGE KKEKPAAVAT AMARVLRETK PIPNL IFAI EQYEKFLIHL SKKSKVNLMQ HMKLSTSRDF KIKGNILDMV LREDGEDENE EGTASEHGGQ NKEPAKKKRK K |
-分子 #2: Fanconi anemia group D2 protein
| 分子 | 名称: Fanconi anemia group D2 protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 166.313719 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: MHHHHHHLEV LFQGPGSMVS KRRLSKSEDK ESLTEDASKT RKQPLSKKTK KSHIANEVEE NDSIFVKLLK ISGIILKTGE SQNQLAVDQ IAFQKKLFQT LRRHPSYPKI IEEFVSGLES YIEDEDSFRN CLLSCERLQD EEASMGASYS KSLIKLLLGI D ILQPAIIK ...文字列: MHHHHHHLEV LFQGPGSMVS KRRLSKSEDK ESLTEDASKT RKQPLSKKTK KSHIANEVEE NDSIFVKLLK ISGIILKTGE SQNQLAVDQ IAFQKKLFQT LRRHPSYPKI IEEFVSGLES YIEDEDSFRN CLLSCERLQD EEASMGASYS KSLIKLLLGI D ILQPAIIK TLFEKLPEYF FENKNSDEIN IPRLIVSQLK WLDRVVDGKD LTTKIMQLIS IAPENLQHDI ITSLPEILGD SQ HADVGKE LSDLLIENTS LTVPILDVLS SLRLDPNFLL KVRQLVMDKL SSIRLEDLPV IIKFILHSVT AMDTLEVISE LRE KLDLQH CVLPSRLQAS QVKLKSKGRA SSSGNQESSG QSCIILLFDV IKSAIRYEKT ISEAWIKAIE NTASVSEHKV FDLV MLFII YSTNTQTKKY IDRVLRNKIR SGCIQEQLLQ STFSVHYLVL KDMCSSILSL AQSLLHSLDQ SIISFGSLLY KYAFK FFDT YCQQEVVGAL VTHICSGNEA EVDTALDVLL ELVVLNPSAM MMNAVFVKGI LDYLDNISPQ QIRKLFYVLS TLAFSK QNE ASSHIQDDMH LVIRKQLSST VFKYKLIGII GAVTMAGIMA ADRSESPSLT QERANLSDEQ CTQVTSLLQL VHSCSEQ SP QASALYYDEF ANLIQHEKLD PKALEWVGHT ICNDFQDAFV VDSCVVPEGD FPFPVKALYG LEEYDTQDGI AINLLPLL F SQDFAKDGGP VTSQESGQKL VSPLCLAPYF RLLRLCVERQ HNGNLEEIDG LLDCPIFLTD LEPGEKLESM SAKERSFMC SLIFLTLNWF REIVNAFCQE TSPEMKGKVL TRLKHIVELQ IILEKYLAVT PDYVPPLGNF DVETLDITPH TVTAISAKIR KKGKIERKQ KTDGSKTSSS DTLSEEKNSE CDPTPSHRGQ LNKEFTGKEE KTSLLLHNSH AFFRELDIEV FSILHCGLVT K FILDTEMH TEATEVVQLG PPELLFLLED LSQKLESMLT PPIARRVPFL KNKGSRNIGF SHLQQRSAQE IVHCVFQLLT PM CNHLENI HNYFQCLAAE NHGVVDGPGV KVQEYHIMSS CYQRLLQIFH GLFAWSGFSQ PENQNLLYSA LHVLSSRLKQ GEH SQPLEE LLSQSVHYLQ NFHQSIPSFQ CALYLIRLLM VILEKSTASA QNKEKIASLA RQFLCRVWPS GDKEKSNISN DQLH ALLCI YLEHTESILK AIEEIAGVGV PELINSPKDA SSSTFPTLTR HTFVVFFRVM MAELEKTVKK IEPGTAADSQ QIHEE KLLY WNMAVRDFSI LINLIKVFDS HPVLHVCLKY GRLFVEAFLK QCMPLLDFSF RKHREDVLSL LETFQLDTRL LHHLCG HSK IHQDTRLTQH VPLLKKTLEL LVCRVKAMLT LNNCREAFWL GNLKNRDLQG EEIKSQNSQE STADESEDDM SSQASKS KA TEDGEEDEVS AGEKEQDSDE SYDDSD |
-分子 #3: Ubiquitin-60S ribosomal protein L40
| 分子 | 名称: Ubiquitin-60S ribosomal protein L40 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 8.875125 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: GPGSMQIFVK TLTGKTITLE VEPSDTIENV KAKIQDKEGI PPDQQRLIFA GKQLEDGRTL SDYNIQKEST LHLVLRLRGG |
-分子 #4: DNA (61-MER)
| 分子 | 名称: DNA (61-MER) / タイプ: dna / ID: 4 詳細: 61-MER DNA modelled using chain S of PDB entry 6VAE as initial model for refinement. Complementary to chain S. コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 8.951746 KDa |
| 配列 | 文字列: (DG)(DG)(DC)(DA)(DC)(DA)(DG)(DG)(DT)(DT) (DC)(DA)(DG)(DA)(DG)(DC)(DA)(DG)(DG)(DC) (DG)(DT)(DT)(DC)(DC)(DG)(DT)(DT)(DC) |
-分子 #5: DNA (61-MER)
| 分子 | 名称: DNA (61-MER) / タイプ: dna / ID: 5 詳細: 61-MER DNA modelled using chain T of PDB entry 6VAE as initial model for refinement. Complementary to chain S. コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 8.880711 KDa |
| 配列 | 文字列: (DG)(DA)(DA)(DC)(DG)(DG)(DA)(DA)(DC)(DG) (DC)(DC)(DT)(DG)(DC)(DT)(DC)(DT)(DG)(DA) (DA)(DC)(DC)(DT)(DG)(DT)(DG)(DC)(DC) |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL CRYO ARM 300 |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy最大 デフォーカス(公称値): 3.8000000000000003 µm 最小 デフォーカス(公称値): 0.5 µm |
| 撮影 | フィルム・検出器のモデル: DIRECT ELECTRON DE-64 (8k x 8k) 検出モード: COUNTING / 平均電子線量: 46.0 e/Å2 |
- 画像解析
画像解析
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
|---|---|
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 4.14 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 139601 |
FSC曲線 (解像度の算出) | 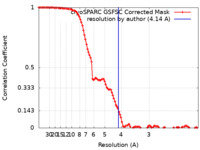 |
 ムービー
ムービー コントローラー
コントローラー
































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)