+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9801 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
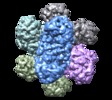
| タイトル | Cryo-EM Structure of Sulfolobus solfataricus ketol-acid reductoisomerase (Sso-KARI) in complex with Mg2+, NADH, and CPD at pH7.5 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Bi-specific /  Thermostable (耐熱性) / Reductoisomerase / Magnesium-dependent / Thermostable (耐熱性) / Reductoisomerase / Magnesium-dependent /  Dodecamer / Dodecamer /  Knotted protein / Knotted protein /  ISOMERASE (異性化酵素) ISOMERASE (異性化酵素) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ketol-acid reductoisomerase (NADP+) /  ketol-acid reductoisomerase activity / valine biosynthetic process / isoleucine biosynthetic process ketol-acid reductoisomerase activity / valine biosynthetic process / isoleucine biosynthetic process類似検索 - 分子機能 | |||||||||
| 生物種 |   Sulfolobus solfataricus P2 (古細菌) / Sulfolobus solfataricus P2 (古細菌) /   Saccharolobus solfataricus (strain ATCC 35092 / DSM 1617 / JCM 11322 / P2) (古細菌) Saccharolobus solfataricus (strain ATCC 35092 / DSM 1617 / JCM 11322 / P2) (古細菌) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.38 Å クライオ電子顕微鏡法 / 解像度: 3.38 Å | |||||||||
 データ登録者 データ登録者 | Chen CY / Chang YC / Lin KF / Huang CH / Lin BL | |||||||||
| 資金援助 |  台湾, 2件 台湾, 2件
| |||||||||
 引用 引用 |  ジャーナル: J Am Chem Soc / 年: 2019 ジャーナル: J Am Chem Soc / 年: 2019タイトル: Use of Cryo-EM To Uncover Structural Bases of pH Effect and Cofactor Bispecificity of Ketol-Acid Reductoisomerase. 著者: Chin-Yu Chen / Yuan-Chih Chang / Bo-Lin Lin / Kuan-Fu Lin / Chun-Hsiang Huang / Dong-Lin Hsieh / Tzu-Ping Ko / Ming-Daw Tsai /  要旨: While cryo-EM is revolutionizing structural biology, its impact on enzymology is yet to be fully demonstrated. The ketol-acid reductoisomerase (KARI) catalyzes conversion of (2 S)-acetolactate or (2 ...While cryo-EM is revolutionizing structural biology, its impact on enzymology is yet to be fully demonstrated. The ketol-acid reductoisomerase (KARI) catalyzes conversion of (2 S)-acetolactate or (2 S)-aceto-2-hydroxybutyrate to 2,3-dihydroxy-3-alkylbutyrate. We found that KARI from archaea Sulfolobus solfataricus (Sso-KARI) is unusual in being a dodecamer, bispecific to NADH and NADPH, and losing activity above pH 7.8. While crystals were obtainable only at pH 8.5, cryo-EM structures were solved at pH 7.5 and 8.5 for Sso-KARI:2Mg. The results showed that the distances of the two catalytic Mg ions are lengthened in both structures at pH 8.5. We next solved cryo-EM structures of two Sso-KARI complexes, with NADH+inhibitor and NADPH+inhibitor at pH 7.5, which indicate that the bispecificity can be attributed to a unique asparagine at the cofactor binding loop. Unexpectedly, Sso-KARI also differs from other KARI enzymes in lacking "induced-fit", reflecting structural rigidity. Thus, cryo-EM is powerful for structural and mechanistic enzymology. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9801.map.gz emd_9801.map.gz | 120 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9801-v30.xml emd-9801-v30.xml emd-9801.xml emd-9801.xml | 14.5 KB 14.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_9801.png emd_9801.png | 195.6 KB | ||
| Filedesc metadata |  emd-9801.cif.gz emd-9801.cif.gz | 6.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9801 http://ftp.pdbj.org/pub/emdb/structures/EMD-9801 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9801 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9801 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9801.map.gz / 形式: CCP4 / 大きさ: 129.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9801.map.gz / 形式: CCP4 / 大きさ: 129.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.87 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM Structure of Sulfolobus solfataricus ketol-acid reductois...
| 全体 | 名称: Cryo-EM Structure of Sulfolobus solfataricus ketol-acid reductoisomerase (Sso-KARI) dodecamer in complex with Mg2+, NADH, and CPD at pH7.5 |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM Structure of Sulfolobus solfataricus ketol-acid reductois...
| 超分子 | 名称: Cryo-EM Structure of Sulfolobus solfataricus ketol-acid reductoisomerase (Sso-KARI) dodecamer in complex with Mg2+, NADH, and CPD at pH7.5 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Sulfolobus solfataricus P2 (古細菌) Sulfolobus solfataricus P2 (古細菌) |
| 分子量 | 理論値: 446 KDa |
-分子 #1: Putative ketol-acid reductoisomerase 2
| 分子 | 名称: Putative ketol-acid reductoisomerase 2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 12 / 光学異性体: LEVO / EC番号: ketol-acid reductoisomerase (NADP+) |
|---|---|
| 由来(天然) | 生物種:   Saccharolobus solfataricus (strain ATCC 35092 / DSM 1617 / JCM 11322 / P2) (古細菌) Saccharolobus solfataricus (strain ATCC 35092 / DSM 1617 / JCM 11322 / P2) (古細菌)株: ATCC 35092 / DSM 1617 / JCM 11322 / P2 |
| 分子量 | 理論値: 37.229855 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: MDKTVLDANL DPLKGKTIGV IGYGNQGRVQ ATIMRENGLN VIVGNVKDKY YELAKKEGFE VYEIDEAVRR SDVALLLIPD EVMKEVYEK KIAPVLQGKK EFVLDFASGY NVAFGLIRPP KSVDTIMVAP RMVGEGIMDL HKQGKGYPVL LGVKQDASGK A WDYAKAIA ...文字列: MDKTVLDANL DPLKGKTIGV IGYGNQGRVQ ATIMRENGLN VIVGNVKDKY YELAKKEGFE VYEIDEAVRR SDVALLLIPD EVMKEVYEK KIAPVLQGKK EFVLDFASGY NVAFGLIRPP KSVDTIMVAP RMVGEGIMDL HKQGKGYPVL LGVKQDASGK A WDYAKAIA KGIGAIPGGI AVISSFEEEA LLDLMSEHTW VPILFGAIKA CYDIAVKEYG VSPEAALLEF YASGELAEIA RL IAEEGIF NQMVHHSTTS QYGTLTRMFK YYDVVRRIVE NEAKYIWDGS FAKEWSLEQQ AGYPVFYRLW ELATQSEMAK AEK ELYKLL GRKVKND UniProtKB:  Putative ketol-acid reductoisomerase 2 Putative ketol-acid reductoisomerase 2 |
-分子 #2: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 2 / コピー数: 36 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #3: 1,4-DIHYDRONICOTINAMIDE ADENINE DINUCLEOTIDE
| 分子 | 名称: 1,4-DIHYDRONICOTINAMIDE ADENINE DINUCLEOTIDE / タイプ: ligand / ID: 3 / コピー数: 12 / 式: NAI |
|---|---|
| 分子量 | 理論値: 665.441 Da |
| Chemical component information |  ChemComp-NAI: |
-分子 #4: cyclopropane-1,1-dicarboxylic acid
| 分子 | 名称: cyclopropane-1,1-dicarboxylic acid / タイプ: ligand / ID: 4 / コピー数: 12 / 式: 9TY |
|---|---|
| 分子量 | 理論値: 130.099 Da |
| Chemical component information | 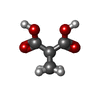 ChemComp-9TY: |
-分子 #5: water
| 分子 | 名称: water / タイプ: ligand / ID: 5 / コピー数: 192 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.15 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / 平均電子線量: 1.31 e/Å2 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 初期 角度割当 | タイプ: COMMON LINE / ソフトウェア - 名称: cisTEM (ver. 1.0.0-beta) |
| 最終 3次元分類 | ソフトウェア - 名称: cisTEM (ver. 1.0.0-beta) |
| 最終 角度割当 | タイプ: PROJECTION MATCHING / ソフトウェア - 名称: cisTEM (ver. 1.0.0-beta) |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: BACK PROJECTION / 解像度のタイプ: BY AUTHOR / 解像度: 3.38 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cisTEM (ver. 1.0.0-beta) / 使用した粒子像数: 18522 |
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A / Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 温度因子: 117.95 |
| 得られたモデル |  PDB-6jd1: |
 ムービー
ムービー コントローラー
コントローラー