+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of insect gustatory receptor BmGr9 | ||||||||||||
 マップデータ マップデータ | Map of unbound BmGr9 | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | gustatory receptor /  homotetramer / homotetramer /  ligand-gated ion channel (リガンド依存性イオンチャネル) / seven transmembrane / ligand-gated ion channel (リガンド依存性イオンチャネル) / seven transmembrane /  MEMBRANE PROTEIN (膜タンパク質) MEMBRANE PROTEIN (膜タンパク質) | ||||||||||||
| 機能・相同性 | 7TM chemoreceptor / 7tm Chemosensory receptor / sensory perception of taste /  シグナル伝達 / シグナル伝達 /  細胞膜 / Gustatory receptor 細胞膜 / Gustatory receptor 機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |   Bombyx mori (カイコ) Bombyx mori (カイコ) | ||||||||||||
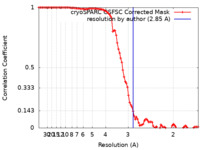
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.85 Å クライオ電子顕微鏡法 / 解像度: 2.85 Å | ||||||||||||
 データ登録者 データ登録者 | Frank HM / Walsh Jr RM / Garrity PA / Gaudet R | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: bioRxiv / 年: 2023 ジャーナル: bioRxiv / 年: 2023タイトル: Structure of an insect gustatory receptor. 著者: Heather M Frank / Sanket Walujkar / Richard M Walsh / Willem J Laursen / Douglas L Theobald / Paul A Garrity / Rachelle Gaudet /  要旨: Gustatory Receptors (GRs) are critical for insect chemosensation and are potential targets for controlling pests and disease vectors. However, GR structures have not been experimentally determined. ...Gustatory Receptors (GRs) are critical for insect chemosensation and are potential targets for controlling pests and disease vectors. However, GR structures have not been experimentally determined. We present structures of Gr9 (BmGr9), a fructose-gated cation channel, in agonist-free and fructose-bound states. BmGr9 forms a tetramer similar to distantly related insect Olfactory Receptors (ORs). Upon fructose binding, BmGr9's ion channel gate opens through helix S7b movements. In contrast to ORs, BmGR9's ligand-binding pocket, shaped by a kinked helix S4 and a shorter extracellular S3-S4 loop, is larger and solvent accessible in both agonist-free and fructose-bound states. Also unlike ORs, fructose binding by BmGr9 involves helix S5 and a binding pocket lined with aromatic and polar residues. Structure-based sequence alignments reveal distinct patterns of ligand-binding pocket residue conservation in GR subfamilies associated with distinct ligand classes. These data provide insight into the molecular basis of GR ligand specificity and function. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_43129.map.gz emd_43129.map.gz | 87.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-43129-v30.xml emd-43129-v30.xml emd-43129.xml emd-43129.xml | 18.2 KB 18.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_43129_fsc.xml emd_43129_fsc.xml | 9.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_43129.png emd_43129.png | 141.6 KB | ||
| Filedesc metadata |  emd-43129.cif.gz emd-43129.cif.gz | 6.7 KB | ||
| その他 |  emd_43129_half_map_1.map.gz emd_43129_half_map_1.map.gz emd_43129_half_map_2.map.gz emd_43129_half_map_2.map.gz | 86 MB 86 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-43129 http://ftp.pdbj.org/pub/emdb/structures/EMD-43129 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-43129 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-43129 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8vc1MC  8vc2C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_43129.map.gz / 形式: CCP4 / 大きさ: 93 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_43129.map.gz / 形式: CCP4 / 大きさ: 93 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map of unbound BmGr9 | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half map A of unbound BmGr9
| ファイル | emd_43129_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A of unbound BmGr9 | ||||||||||||
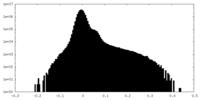
| 投影像・断面図 |
| ||||||||||||
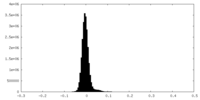
| 密度ヒストグラム |
-ハーフマップ: Half map B of unbound BmGr9
| ファイル | emd_43129_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B of unbound BmGr9 | ||||||||||||
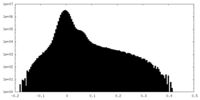
| 投影像・断面図 |
| ||||||||||||
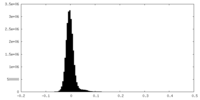
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Agonist-free BmGr9 homotetramer
| 全体 | 名称: Agonist-free BmGr9 homotetramer |
|---|---|
| 要素 |
|
-超分子 #1: Agonist-free BmGr9 homotetramer
| 超分子 | 名称: Agonist-free BmGr9 homotetramer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Bombyx mori (カイコ) Bombyx mori (カイコ) |
| 分子量 | 理論値: 218.05 KDa |
-分子 #1: Gustatory receptor
| 分子 | 名称: Gustatory receptor / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Bombyx mori (カイコ) Bombyx mori (カイコ) |
| 分子量 | 理論値: 54.57232 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGWSHPQFEK GGGSGGGSGG SAWSHPQFEK AALEVLFQGP GTMPPSPDLR ADEPKTPCLV GGAHAFILKI SSFCGLAPLR FEPRSQEYA VTISKGKCFY SYILVTFLVI CTIYGLVAEI GVGVEKSVRM SSRMSQVVSA CDILVVAVTA GVGVYGAPAR M RTMLSYME ...文字列: MGWSHPQFEK GGGSGGGSGG SAWSHPQFEK AALEVLFQGP GTMPPSPDLR ADEPKTPCLV GGAHAFILKI SSFCGLAPLR FEPRSQEYA VTISKGKCFY SYILVTFLVI CTIYGLVAEI GVGVEKSVRM SSRMSQVVSA CDILVVAVTA GVGVYGAPAR M RTMLSYME NIVAVDRELG RHHSAATERK LCALLLLILL SFTILLVDDF CFYAMQAGKT GRQWEIVTNY AGFYFLWYIV MV LELQFAF TALSLRARLK LFNEALNVTA SQVCKPVKKP KNSQLSVYAT SVRPVSCKRE NVIVETIRVR DKDDAFVMMK TAD GVPCLQ VPPCEAVGRL SRMRCTLCEV TRHIADGYGL PLVIILMSTL LHLIVTPYFL IMEIIVSTHR LHFLVLQFLW CTTH LIRML VVVEPCHYTM REGKRTEDIV CRLMTLAPHG GVLSSKLEVL SRLLMLQNIS YSPLGMCTLD RPLIVTVLGA VTTYL VILI QFQRYDS UniProtKB: Gustatory receptor |
-分子 #2: (7R,17E,20E)-4-HYDROXY-N,N,N-TRIMETHYL-9-OXO-7-[(PALMITOYLOXY)MET...
| 分子 | 名称: (7R,17E,20E)-4-HYDROXY-N,N,N-TRIMETHYL-9-OXO-7-[(PALMITOYLOXY)METHYL]-3,5,8-TRIOXA-4-PHOSPHAHEXACOSA-17,20-DIEN-1-AMINIUM 4-OXIDE タイプ: ligand / ID: 2 / コピー数: 8 / 式: PSC |
|---|---|
| 分子量 | 理論値: 759.068 Da |
| Chemical component information |  ChemComp-PSC: |
-分子 #3: (7S)-4-HYDROXY-N,N,N-TRIMETHYL-9-OXO-7-[(PALMITOYLOXY)METHYL]-3,5...
| 分子 | 名称: (7S)-4-HYDROXY-N,N,N-TRIMETHYL-9-OXO-7-[(PALMITOYLOXY)METHYL]-3,5,8-TRIOXA-4-PHOSPHAHEXACOSAN-1-AMINIUM 4-OXIDE タイプ: ligand / ID: 3 / コピー数: 20 / 式: PC7 |
|---|---|
| 分子量 | 理論値: 763.1 Da |
| Chemical component information |  ChemComp-PC7: |
-分子 #4: (3beta,14beta,17beta,25R)-3-[4-methoxy-3-(methoxymethyl)butoxy]sp...
| 分子 | 名称: (3beta,14beta,17beta,25R)-3-[4-methoxy-3-(methoxymethyl)butoxy]spirost-5-en タイプ: ligand / ID: 4 / コピー数: 4 / 式: 9Z9 |
|---|---|
| 分子量 | 理論値: 544.805 Da |
| Chemical component information |  ChemComp-9Z9: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8.25 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 倍率(補正後): 60240 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 105000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 105000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 11474 / 平均露光時間: 3.803 sec. / 平均電子線量: 74.492 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: Other / Chain - Initial model type: in silico model / 詳細: ColabFold generated |
|---|---|
| 詳細 | Initial fitting was done using DockInMap in PHENIX and refined through cycles of manual rebuilding in Coot, real-space refinement in PHENIX, and remodeling by simulations run in the ISOLDE plugin of ChimeraX. |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 温度因子: 58.19 当てはまり具合の基準: cross-correlation coefficient |
| 得られたモデル |  PDB-8vc1: |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X