+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | E. coli 70S ribosome with an improved MS2 tag inserted in H98 (50S focused map) | |||||||||
 マップデータ マップデータ | E. coli 70S ribosome with an improved MS2 tag inserted in H98 (50S focused map) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | MS2-tag / H98 /  ribosome (リボソーム) ribosome (リボソーム) | |||||||||
| 生物種 |   Escherichia coli (大腸菌) Escherichia coli (大腸菌) | |||||||||
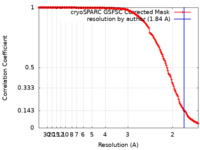
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 1.84 Å クライオ電子顕微鏡法 / 解像度: 1.84 Å | |||||||||
 データ登録者 データ登録者 | Nissley AJ / Cate JHD | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: RNA / 年: 2023 ジャーナル: RNA / 年: 2023タイトル: Interactions between terminal ribosomal RNA helices stabilize the large ribosomal subunit. 著者: Amos J Nissley / Tammam S Kamal / Jamie H D Cate /  要旨: The ribosome is a large ribonucleoprotein assembly that uses diverse and complex molecular interactions to maintain proper folding. In vivo assembled ribosomes have been isolated using MS2 tags ...The ribosome is a large ribonucleoprotein assembly that uses diverse and complex molecular interactions to maintain proper folding. In vivo assembled ribosomes have been isolated using MS2 tags installed in either the 16S or 23S ribosomal RNAs (rRNAs), to enable studies of ribosome structure and function in vitro. RNA tags in the 50S subunit have commonly been inserted into an extended helix H98 in 23S rRNA, as this addition does not affect cellular growth or in vitro ribosome activity. Here, we find that 50S subunits with MS2 tags inserted in H98 are destabilized compared to wild-type (WT) 50S subunits. We identify the loss of RNA-RNA tertiary contacts that bridge helices H1, H94, and H98 as the cause of destabilization. Using cryogenic electron microscopy (cryo-EM), we show that this interaction is disrupted by the addition of the MS2 tag and can be restored through the insertion of a single adenosine in the extended H98 helix. This work establishes ways to improve MS2 tags in the 50S subunit that maintain ribosome stability and investigates a complex RNA tertiary structure that may be important for stability in various bacterial ribosomes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_29484.map.gz emd_29484.map.gz | 324.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-29484-v30.xml emd-29484-v30.xml emd-29484.xml emd-29484.xml | 15.6 KB 15.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_29484_fsc.xml emd_29484_fsc.xml | 14.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_29484.png emd_29484.png | 122.9 KB | ||
| Filedesc metadata |  emd-29484.cif.gz emd-29484.cif.gz | 4.1 KB | ||
| その他 |  emd_29484_half_map_1.map.gz emd_29484_half_map_1.map.gz emd_29484_half_map_2.map.gz emd_29484_half_map_2.map.gz | 318.1 MB 318.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29484 http://ftp.pdbj.org/pub/emdb/structures/EMD-29484 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29484 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29484 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_29484.map.gz / 形式: CCP4 / 大きさ: 343 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_29484.map.gz / 形式: CCP4 / 大きさ: 343 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | E. coli 70S ribosome with an improved MS2 tag inserted in H98 (50S focused map) | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8248 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: E. coli 70S ribosome with an improved MS2...
| ファイル | emd_29484_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | E. coli 70S ribosome with an improved MS2 tag inserted in H98 (50S focused half map) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
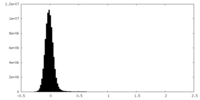
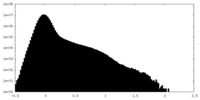
| 密度ヒストグラム |
-ハーフマップ: E. coli 70S ribosome with an improved MS2...
| ファイル | emd_29484_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | E. coli 70S ribosome with an improved MS2 tag inserted in H98 (50S focused half map) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
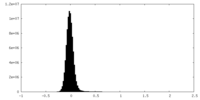
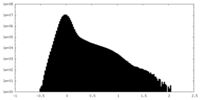
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : E. coli 70S ribosome
| 全体 | 名称: E. coli 70S ribosome |
|---|---|
| 要素 |
|
-超分子 #1: E. coli 70S ribosome
| 超分子 | 名称: E. coli 70S ribosome / タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-超分子 #2: 50S Subunit
| 超分子 | 名称: 50S Subunit / タイプ: complex / ID: 2 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-超分子 #3: 30S Subunit
| 超分子 | 名称: 30S Subunit / タイプ: complex / ID: 3 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.27 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm Bright-field microscopy / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 11736 / 平均電子線量: 40.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT |
|---|
 ムービー
ムービー コントローラー
コントローラー








 Z
Z Y
Y X
X