+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | YcaO-mediated ATP-dependent peptidase activity in ribosomal peptide biosynthesis | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 | Bacteriocin biosynthesis, cyclodehydratase domain / Thiazole/oxazole-forming peptide maturase, SagD family component / YcaO-like domain / YcaO cyclodehydratase, ATP-ad Mg2+-binding / YcaO domain profile. / ubiquitin-like modifier activating enzyme activity /  ユビキチン活性化酵素 / MusD ユビキチン活性化酵素 / MusD 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Desmonostoc sp. PCC 7906 (バクテリア) Desmonostoc sp. PCC 7906 (バクテリア) | |||||||||
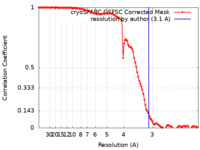
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.1 Å クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Zheng Y / Nair SK | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Chem Biol / 年: 2023 ジャーナル: Nat Chem Biol / 年: 2023タイトル: YcaO-mediated ATP-dependent peptidase activity in ribosomal peptide biosynthesis. 著者: Yiwu Zheng / Satish K Nair /  要旨: YcaO enzymes catalyze ATP-dependent post-translation modifications on peptides, including the installation of (ox/thi)azoline, thioamide and/or amidine moieties. Here we demonstrate that, in the ...YcaO enzymes catalyze ATP-dependent post-translation modifications on peptides, including the installation of (ox/thi)azoline, thioamide and/or amidine moieties. Here we demonstrate that, in the biosynthesis of the bis-methyloxazolic alkaloid muscoride A, the YcaO enzyme MusD carries out both ATP-dependent cyclodehydration and peptide bond cleavage, which is a mechanism unprecedented for such a reaction. YcaO-catalyzed modifications are proposed to occur through a backbone O-phosphorylated intermediate, but this mechanism remains speculative. We report, to our knowedge, the first characterization of an acyl-phosphate species consistent with the proposed mechanism for backbone amide activation. The 3.1-Å-resolution cryogenic electron microscopy structure of MusD along with biochemical analysis allow identification of residues that enable peptide cleavage reaction. Bioinformatics analysis identifies other cyanobactin pathways that may deploy bifunctional YcaO enzymes. Our structural, mutational and mechanistic studies expand the scope of modifications catalyzed by YcaO proteins to include peptide hydrolysis and provide evidence for a unifying mechanism for the catalytically diverse outcomes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26344.map.gz emd_26344.map.gz | 62.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26344-v30.xml emd-26344-v30.xml emd-26344.xml emd-26344.xml | 14.4 KB 14.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_26344_fsc.xml emd_26344_fsc.xml | 10.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_26344.png emd_26344.png | 104.4 KB | ||
| マスクデータ |  emd_26344_msk_1.map emd_26344_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| その他 |  emd_26344_half_map_1.map.gz emd_26344_half_map_1.map.gz emd_26344_half_map_2.map.gz emd_26344_half_map_2.map.gz | 115.7 MB 115.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26344 http://ftp.pdbj.org/pub/emdb/structures/EMD-26344 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26344 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26344 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7u58MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26344.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26344.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.054 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_26344_msk_1.map emd_26344_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_26344_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_26344_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : MusD
| 全体 | 名称: MusD |
|---|---|
| 要素 |
|
-超分子 #1: MusD
| 超分子 | 名称: MusD / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Desmonostoc sp. PCC 7906 (バクテリア) Desmonostoc sp. PCC 7906 (バクテリア) |
| 分子量 | 理論値: 88.76 kDa/nm |
-分子 #1: MusD
| 分子 | 名称: MusD / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Desmonostoc sp. PCC 7906 (バクテリア) / 株: PCC 7906 Desmonostoc sp. PCC 7906 (バクテリア) / 株: PCC 7906 |
| 分子量 | 理論値: 88.844805 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: SNAMKILQIK PHFRAEIIEP KHVYLLSESS THALTGELYC QLIPLLNGNY TVDEIINKLQ VDPSHIDYAL ERLQARGYIT EAIPQLTPE AVAFWGLLKV EPQVAYQCLQ QTQVYVSSVV NLPTQPLITA LEEVGIKAIN WDGELQEFPP HSLLVVLTDD Y LQPQLNKI ...文字列: SNAMKILQIK PHFRAEIIEP KHVYLLSESS THALTGELYC QLIPLLNGNY TVDEIINKLQ VDPSHIDYAL ERLQARGYIT EAIPQLTPE AVAFWGLLKV EPQVAYQCLQ QTQVYVSSVV NLPTQPLITA LEEVGIKAIN WDGELQEFPP HSLLVVLTDD Y LQPQLNKI NQIALKANQP WLLIKPVGTI LWLGPIFQPQ ITGCWECLAQ RLRVNREVEA SVLRQKNSSL QLSPSQELNS SV LQSNGNG VKSEVIECLP PPAAVIPSTL QTALHLATTE IAKWIVKQGV EDTTPFPTLE GKVITFDQRN LDLQTHILSL RPQ CPSCGN PNLLTERAFQ PLVLSSRKKQ FTSDGGHRAF SPDQTVNRYQ HLISPITGVV TSLVRASDPN DSLNHTYNAV HSFV IASNI GRMRRYLKHK SSGKGKTDSQ SKASGFCEAI ERYSGVYQGD EPRISATLAE LGEKAIHPAR CSLFSSEQYE YREEF NRRG GVFDWIPQPF DETKVIEWTP VWSLTEQTHK YIPTAYCYYG YPLPEDHEFC RANSNGDATG NTLEEAIIQG FFEIVE RDS VAIWWYNRLK RPAVDLASFN EPYLLEVQDL YRSNNRDLWV IDITADLDIP TFVAVSYLKD NKHQTILLGF GTHFDPK IA ILRAVTEVNQ IAFTCDGVEV TKEFVEMREW FKKATIENQP YLVPDSTVPA KVYQDYQQRW SDDIYEDVMT CVEISKNA G LETLVLDKTR PDIGLNVAKV IVPEMPHYWL RMGAKRIYDV PVKMGWLSTP LTEEQMNPIS VPI |
-分子 #2: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 2 / コピー数: 2 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 4 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | 2D array |
- 試料調製
試料調製
| 濃度 | 6 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: HEPES pH 7.5. 25 mM KCl. |
| グリッド | 材質: COPPER |
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: DIFFRACTION 回折 / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.6 µm 回折 / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.6 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 1.33 e/Å2 |
 ムービー
ムービー コントローラー
コントローラー






 Z
Z Y
Y X
X