+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Bestrhodopsin (rhodopsin-rhodopsin-bestrophin) complex | |||||||||
 マップデータ マップデータ | Sharpened cryoEM map | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Phaeocystis antarctica (真核生物) / Phaeocystis antarctica (真核生物) /   Phaeocystis (真核生物) Phaeocystis (真核生物) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.21 Å クライオ電子顕微鏡法 / 解像度: 3.21 Å | |||||||||
 データ登録者 データ登録者 | Matzov D / Kaczmarczyk I / Shalev-Benami M | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2022 ジャーナル: Nat Struct Mol Biol / 年: 2022タイトル: Rhodopsin-bestrophin fusion proteins from unicellular algae form gigantic pentameric ion channels. 著者: Andrey Rozenberg / Igor Kaczmarczyk / Donna Matzov / Johannes Vierock / Takashi Nagata / Masahiro Sugiura / Kota Katayama / Yuma Kawasaki / Masae Konno / Yujiro Nagasaka / Mako Aoyama / ...著者: Andrey Rozenberg / Igor Kaczmarczyk / Donna Matzov / Johannes Vierock / Takashi Nagata / Masahiro Sugiura / Kota Katayama / Yuma Kawasaki / Masae Konno / Yujiro Nagasaka / Mako Aoyama / Ishita Das / Efrat Pahima / Jonathan Church / Suliman Adam / Veniamin A Borin / Ariel Chazan / Sandra Augustin / Jonas Wietek / Julien Dine / Yoav Peleg / Akira Kawanabe / Yuichiro Fujiwara / Ofer Yizhar / Mordechai Sheves / Igor Schapiro / Yuji Furutani / Hideki Kandori / Keiichi Inoue / Peter Hegemann / Oded Béjà / Moran Shalev-Benami /    要旨: Many organisms sense light using rhodopsins, photoreceptive proteins containing a retinal chromophore. Here we report the discovery, structure and biophysical characterization of bestrhodopsins, a ...Many organisms sense light using rhodopsins, photoreceptive proteins containing a retinal chromophore. Here we report the discovery, structure and biophysical characterization of bestrhodopsins, a microbial rhodopsin subfamily from marine unicellular algae, in which one rhodopsin domain of eight transmembrane helices or, more often, two such domains in tandem, are C-terminally fused to a bestrophin channel. Cryo-EM analysis of a rhodopsin-rhodopsin-bestrophin fusion revealed that it forms a pentameric megacomplex (~700 kDa) with five rhodopsin pseudodimers surrounding the channel in the center. Bestrhodopsins are metastable and undergo photoconversion between red- and green-absorbing or green- and UVA-absorbing forms in the different variants. The retinal chromophore, in a unique binding pocket, photoisomerizes from all-trans to 11-cis form. Heterologously expressed bestrhodopsin behaves as a light-modulated anion channel. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13485.map.gz emd_13485.map.gz | 223.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13485-v30.xml emd-13485-v30.xml emd-13485.xml emd-13485.xml | 19.2 KB 19.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_13485.png emd_13485.png | 143.5 KB | ||
| その他 |  emd_13485_additional_1.map.gz emd_13485_additional_1.map.gz emd_13485_half_map_1.map.gz emd_13485_half_map_1.map.gz emd_13485_half_map_2.map.gz emd_13485_half_map_2.map.gz | 191.1 MB 194.4 MB 194.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13485 http://ftp.pdbj.org/pub/emdb/structures/EMD-13485 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13485 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13485 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13485.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13485.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened cryoEM map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.86 Å | ||||||||||||||||||||
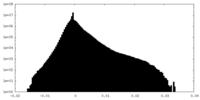
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Unsharpened cryoEM map
| ファイル | emd_13485_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened cryoEM map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
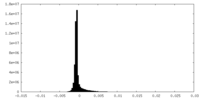
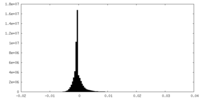
| 密度ヒストグラム |
-ハーフマップ: half map 1
| ファイル | emd_13485_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
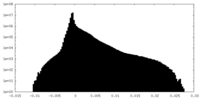
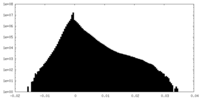
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_13485_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
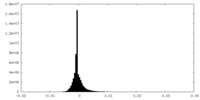
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Rhodopsin
| 全体 | 名称: Rhodopsin ロドプシン ロドプシン |
|---|---|
| 要素 |
|
-超分子 #1: Rhodopsin
| 超分子 | 名称: Rhodopsin / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Phaeocystis antarctica (真核生物) Phaeocystis antarctica (真核生物) |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 分子量 | 理論値: 670 KDa |
-分子 #1: Rhodopsin
| 分子 | 名称: Rhodopsin / タイプ: protein_or_peptide / ID: 1 / コピー数: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Phaeocystis (真核生物) Phaeocystis (真核生物) |
| 分子量 | 理論値: 134.01025 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: MASSTAAPPA AVPAIETAAP PSPEPRDSVS VNEGGDASSA QAGAAVGSTI IVGAPELEKE YSDLNFAQVA RDEGRRCLLM CVAFAIAIA HLYIYPALFG VRIVDQAEVP AEERTYFHHG WTAMLVIFFI EGVTVFLKVC STRKTRWLEK AVLQKLDGNI G VLIGEYIV ...文字列: MASSTAAPPA AVPAIETAAP PSPEPRDSVS VNEGGDASSA QAGAAVGSTI IVGAPELEKE YSDLNFAQVA RDEGRRCLLM CVAFAIAIA HLYIYPALFG VRIVDQAEVP AEERTYFHHG WTAMLVIFFI EGVTVFLKVC STRKTRWLEK AVLQKLDGNI G VLIGEYIV VAATYIIMGA NLIPVFEEPR SGRRVYAVRY MEWTIDACGL VYLDCRILFG MPFSKFRMLL VYSVLYMLFG LW AALASTW MWYAIFLSAS WFFFGLVCYY YWTFHRQNPS PLQQFGRAPI KQAILVFVIV WWVLYGVLFM LCFQAPDVVP QWL EQLLWT GMDVVMKLSH TVVLMAWRET QWEIDAVVDR QKVEAGRAIA QLDHQRAIHE RDLVRLRSRV YYGEHIKSEE EIKS EEVIS RSLRARKSRQ GQDGTEPSSA LSGASSTKEP PALEENSGSA APSWTAVLAK GKATSSPFAR VNKIFMREAG LCLVL CLAF VVALLHLPVY SEWFGVEVLD AEAVPHDELG FFHHGWTTML VVFLIESITV LLKVWSTWHD PRLAENVAQQ LSGNLG VLI AEYLVVGATY VILGYNLMPV FVVHRPGVAS RRVYAVRYME WAVDATGLIW LDCHCLFSRN FNEFRMAIVW TVAYMLF GL WSALASTWAW YWAFLLASWA AFLIVCLILV RFLRQDPYPH QPFGKTSVKP CILAFIIGWW VLYGILFMVC FQAPDAVP Q WLEQFLWTGM DVVMKLSHTV VLMAWRTTEW NVCELHGRNQ AEKSKKSLLN EGSHARQEKQ QLEAIMLEGG GVEAAGRVG SVSFAPSSSR RRKESDSTNW TATPGLRVDL SSMVRLEGQL AQGLVTDVHR KGMMRSEDLA ELKRLEESGF LQAQQHRNWE SQTREMTFL AHGINHIAYD PRSWMKTLTA VRGRAPTSFL LWVVLIESSI VLALSKFFGE SFDLGVSSGI HSLFGVLVSF L VVFRTQAA FKKWWSGRSA VSSLVQMSRT FAQQVCAYVK DEAYVNRMVR YSIATVVATR CHLRNTRIDP AMLLGVLKEE EI EELNRQK NLPFYTAWVI RSTLAEAVAE GACLPLHMAI ENAIKAIEQS IADAERLLTP MPFTYVVHVR TFLFIYLMGL PFI LVEDLG WLMLVAVSFL GYLMIGLENT AVQLENPFGT DCNHHPLDLY CLEVSQDLLH LLDLRASAKA Q |
-分子 #2: RETINAL
| 分子 | 名称: RETINAL / タイプ: ligand / ID: 2 / コピー数: 5 / 式: RET |
|---|---|
| 分子量 | 理論値: 284.436 Da |
| Chemical component information |  ChemComp-RET: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 0.86 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
|---|---|
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.21 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 27749 |
 ムービー
ムービー コントローラー
コントローラー






 Z
Z Y
Y X
X