+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: SASBDB / ID: SASDAZ7 |
|---|---|
 試料 試料 | IL-6R AIR-3A 2:4 complex
|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ciliary neurotrophic factor binding / hepatic immune response / interleukin-6 receptor activity / interleukin-6 binding / ciliary neurotrophic factor-mediated signaling pathway / interleukin-11 receptor activity / interleukin-11 binding / ciliary neurotrophic factor receptor complex / interleukin-6 receptor complex / T-helper 17 cell lineage commitment ...ciliary neurotrophic factor binding / hepatic immune response / interleukin-6 receptor activity / interleukin-6 binding / ciliary neurotrophic factor-mediated signaling pathway / interleukin-11 receptor activity / interleukin-11 binding / ciliary neurotrophic factor receptor complex / interleukin-6 receptor complex / T-helper 17 cell lineage commitment / endocrine pancreas development / negative regulation of collagen biosynthetic process / vascular endothelial growth factor production / negative regulation of interleukin-8 production / positive regulation of glomerular mesangial cell proliferation / positive regulation of leukocyte chemotaxis / neutrophil mediated immunity / cytokine receptor activity / cell surface receptor signaling pathway via STAT / Interleukin-6 signaling / interleukin-6-mediated signaling pathway / MAPK3 (ERK1) activation / MAPK1 (ERK2) activation / monocyte chemotaxis / positive regulation of osteoblast differentiation / positive regulation of chemokine production / extrinsic apoptotic signaling pathway / response to cytokine / positive regulation of smooth muscle cell proliferation / acute-phase response / positive regulation of interleukin-6 production / Transcriptional regulation of granulopoiesis / cytokine-mediated signaling pathway / Interleukin-4 and Interleukin-13 signaling / defense response to Gram-negative bacterium / Potential therapeutics for SARS / receptor complex / positive regulation of MAPK cascade / defense response to Gram-positive bacterium / apical plasma membrane / external side of plasma membrane / positive regulation of cell population proliferation / enzyme binding / protein homodimerization activity / extracellular space / extracellular region / plasma membrane 類似検索 - 分子機能 |
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
 引用 引用 |  ジャーナル: RNA Biol / 年: 2016 ジャーナル: RNA Biol / 年: 2016タイトル: Structure and target interaction of a G-quadruplex RNA-aptamer. 著者: Kristina Szameit / Katharina Berg / Sven Kruspe / Erica Valentini / Eileen Magbanua / Marcel Kwiatkowski / Isaure Chauvot de Beauchêne / Boris Krichel / Kira Schamoni / Charlotte Uetrecht / ...著者: Kristina Szameit / Katharina Berg / Sven Kruspe / Erica Valentini / Eileen Magbanua / Marcel Kwiatkowski / Isaure Chauvot de Beauchêne / Boris Krichel / Kira Schamoni / Charlotte Uetrecht / Dmitri I Svergun / Hartmut Schlüter / Martin Zacharias / Ulrich Hahn /  要旨: G-quadruplexes have recently moved into focus of research in nucleic acids, thereby evolving in scientific significance from exceptional secondary structure motifs to complex modulators of gene ...G-quadruplexes have recently moved into focus of research in nucleic acids, thereby evolving in scientific significance from exceptional secondary structure motifs to complex modulators of gene regulation. Aptamers (nucleic acid based ligands with recognition properties for a specific target) that form Gquadruplexes may have particular potential for therapeutic applications as they combine the characteristics of specific targeting and Gquadruplex mediated stability and regulation. We have investigated the structure and target interaction properties of one such aptamer: AIR-3 and its truncated form AIR-3A. These RNA aptamers are specific for human interleukin-6 receptor (hIL-6R), a key player in inflammatory diseases and cancer, and have recently been exploited for in vitro drug delivery studies. With the aim to resolve the RNA structure, global shape, RNA:protein interaction site and binding stoichiometry, we now investigated AIR-3 and AIR-3A by different methods including RNA structure probing, Small Angle X-ray scattering and microscale thermophoresis. Our findings suggest a broader spectrum of folding species than assumed so far and remarkable tolerance toward different modifications. Mass spectrometry based binding site analysis, supported by molecular modeling and docking studies propose a general Gquadruplex affinity for the target molecule hIL-6R. |
 登録者 登録者 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-モデル
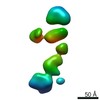
| モデル #305 |  タイプ: dummy / ダミー原子の半径: 5.50 A / カイ2乗値: 1.036  Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
|---|---|
| モデル #306 |  タイプ: mix / 対称性: P1 / カイ2乗値: 1.43  Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
- 試料
試料
 試料 試料 | 名称: IL-6R AIR-3A 2:4 complex / 試料濃度: 0.2 mg/ml / Entity id: 173 / 174 |
|---|---|
| バッファ | 名称: water / pH: 7.5 |
| 要素 #173 | タイプ: RNA / 記述: AIR-3A / 分子量: 6.388 / 分子数: 4 配列: GGGGAGGCUG UGGUGAGGG |
| 要素 #174 | 名称: IL-6R / タイプ: protein / 記述: Interleukin-6 receptor subunit alpha / 分子量: 40.964 / 分子数: 2 / 由来: Homo sapiens / 参照: UniProt: P08887 配列: MRAWIFFLLC LAGRALAAPL VHHHHHHALA PRRCPAQEVA RGVLTSLPGD SVTLTCPGVE PEDNATVHWV LRKPAAGSHP SRWAGMGRRL LLRSVQLHDS GNYSCYRAGR PAGTVHLLVD VPPEEPQLSC FRKSPLSNVV CEWGPRSTPS LTTKAVLLVR KFQNSPAEDF ...配列: MRAWIFFLLC LAGRALAAPL VHHHHHHALA PRRCPAQEVA RGVLTSLPGD SVTLTCPGVE PEDNATVHWV LRKPAAGSHP SRWAGMGRRL LLRSVQLHDS GNYSCYRAGR PAGTVHLLVD VPPEEPQLSC FRKSPLSNVV CEWGPRSTPS LTTKAVLLVR KFQNSPAEDF QEPCQYSQES QKFSCQLAVP EGDSSFYIVS MCVASSVGSK FSKTQTFQGC GILQPDPPAN ITVTAVARNP RWLSVTWQDP HSWNSSFYRL RFELRYRAER SKTFTTWMVK DLQHHCVIHD AWSGLRHVVQ LRAQEEFGQG EWSEWSPEAM GTPWTESRSP PAENEVSTPM QALTTNKDDD NILFRDSANA TSLPVQ |
-実験情報
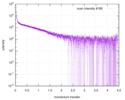
| ビーム | 設備名称: Diamond Light Source B21 / 地域: Oxfordshire / 国: UK  / 形状: 1 x 5 mm / 線源: X-ray synchrotron / スペクトロメータ・検出器間距離: 3.9 mm / 形状: 1 x 5 mm / 線源: X-ray synchrotron / スペクトロメータ・検出器間距離: 3.9 mm | |||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 検出器 | 名称: Pilatus 2M | |||||||||||||||||||||||||||
| スキャン |
| |||||||||||||||||||||||||||
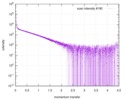
| 距離分布関数 P(R) |
| |||||||||||||||||||||||||||
| 結果 | コメント: The CORAL model fits the experimental curve with a Chi-squared-value of 1.43. The model appears slightly smaller probably because of the presence of free RNA dimers in solution.
|
 ムービー
ムービー コントローラー
コントローラー


 SASDAZ7
SASDAZ7