+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 9nk8 | ||||||
|---|---|---|---|---|---|---|---|
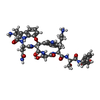
| タイトル | Thermothelomyces thermophilus SAM complex bound to darobactin A | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | MEMBRANE PROTEIN/ANTIBIOTIC / beta-barrel / sorting and assembly machinery / MEMBRANE PROTEIN-ANTIBIOTIC complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報SAM complex / protein insertion into mitochondrial outer membrane / mitochondrion organization / protein transport / mitochondrial outer membrane 類似検索 - 分子機能 | ||||||
| 生物種 |  Thermothelomyces thermophilus (菌類) Thermothelomyces thermophilus (菌類) Photorhabdus khanii (バクテリア) Photorhabdus khanii (バクテリア) | ||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3 Å | ||||||
 データ登録者 データ登録者 | Diederichs, K. / Botos, I. / Buchanan, S.K. | ||||||
| 資金援助 |  米国, 1件 米国, 1件
| ||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2025 ジャーナル: Nat Commun / 年: 2025タイトル: The dynamic lateral gate of the mitochondrial β-barrel biogenesis machinery is blocked by darobactin A. 著者: Kathryn A Diederichs / Istvan Botos / Scout Hayashi / Gvantsa Gutishvili / Vadim Kotov / Katie Kuo / Akira Iinishi / Gwendolyn Cooper / Benjamin Schwarz / Herve Celia / Thomas C Marlovits / ...著者: Kathryn A Diederichs / Istvan Botos / Scout Hayashi / Gvantsa Gutishvili / Vadim Kotov / Katie Kuo / Akira Iinishi / Gwendolyn Cooper / Benjamin Schwarz / Herve Celia / Thomas C Marlovits / Kim Lewis / James C Gumbart / Joseph A Mindell / Susan K Buchanan /   要旨: The folding and insertion of β-barrel proteins into the mitochondrial outer membrane is facilitated by the sorting and assembly machinery (SAM) complex. Here we report two 2.8 Å cryo-EM ...The folding and insertion of β-barrel proteins into the mitochondrial outer membrane is facilitated by the sorting and assembly machinery (SAM) complex. Here we report two 2.8 Å cryo-EM structures of the Thermothelomyces thermophilus SAM complex in the absence of substrate in which the Sam50 lateral gate adopts two different conformations: the first is a closed lateral gate as observed in previously published structures, while the second contains a Sam50 with the first four β-strands rotated outwards by approximately 45°, resulting in an open lateral gate. The observed monomeric open conformation contrasts our previous work where the open conformation was adopted by non-physiological up-down dimers. To understand how these lateral gate dynamics are influenced by substrate, we studied the interaction of the SAM complex with a β-signal peptide mimic, darobactin A. Darobactin A binds to the SAM complex with nanomolar affinity and inhibits the import and assembly of mitochondrial β-barrel proteins in vitro. Lastly, we solved a 3.0 Å cryo-EM structure of the Thermothelomyces thermophilus SAM complex bound to darobactin A, which reveals that darobactin A stabilizes the Sam50 lateral gate similar to the open conformation by binding to strand β1, therefore blocking β-barrel biogenesis. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  9nk8.cif.gz 9nk8.cif.gz | 241.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb9nk8.ent.gz pdb9nk8.ent.gz | 188.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  9nk8.json.gz 9nk8.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  9nk8_validation.pdf.gz 9nk8_validation.pdf.gz | 1.8 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  9nk8_full_validation.pdf.gz 9nk8_full_validation.pdf.gz | 1.8 MB | 表示 | |
| XML形式データ |  9nk8_validation.xml.gz 9nk8_validation.xml.gz | 45 KB | 表示 | |
| CIF形式データ |  9nk8_validation.cif.gz 9nk8_validation.cif.gz | 65.7 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/nk/9nk8 https://data.pdbj.org/pub/pdb/validation_reports/nk/9nk8 ftp://data.pdbj.org/pub/pdb/validation_reports/nk/9nk8 ftp://data.pdbj.org/pub/pdb/validation_reports/nk/9nk8 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  49496MC  9nk6C  9nk7C M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 36882.680 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  Thermothelomyces thermophilus (菌類) Thermothelomyces thermophilus (菌類)遺伝子: MYCTH_2142789 / 発現宿主:  | ||||
|---|---|---|---|---|---|
| #2: タンパク質 | 分子量: 53468.043 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  Thermothelomyces thermophilus (菌類) Thermothelomyces thermophilus (菌類)遺伝子: MYCTH_2094326 / 発現宿主:  | ||||
| #3: タンパク質 | 分子量: 49327.566 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  Thermothelomyces thermophilus (菌類) Thermothelomyces thermophilus (菌類)遺伝子: MYCTH_2293977 / 発現宿主:  | ||||
| #4: タンパク質・ペプチド | | ||||
| #5: 化合物 | ChemComp-ERG / 研究の焦点であるリガンドがあるか | Y | Has protein modification | Y | |
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Thermothelomyces thermophilus SAM complex bound to darobactin A タイプ: COMPLEX / Entity ID: #1-#4 / 由来: MULTIPLE SOURCES |
|---|---|
| 由来(天然) | 生物種:  Thermothelomyces thermophilus (菌類) Thermothelomyces thermophilus (菌類) |
| 由来(組換発現) | 生物種:  |
| 緩衝液 | pH: 8 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 試料支持 | グリッドの材料: COPPER / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: Quantifoil R1.2/1.3 |
| 急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 295 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: TFS KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2000 nm / 最小 デフォーカス(公称値): 700 nm / アライメント法: COMA FREE |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 電子線照射量: 54.4 e/Å2 フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) |
- 解析
解析
| EMソフトウェア | 名称: PHENIX / バージョン: 1.21rc1_5084 / カテゴリ: モデル精密化 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 163391 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 精密化 | 最高解像度: 3 Å 立体化学のターゲット値: REAL-SPACE (WEIGHTED MAP SUM AT ATOM CENTERS) | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー





 PDBj
PDBj