[English] 日本語
 Yorodumi
Yorodumi- PDB-9mmr: Cryo-EM structure of CRAF/MEK1/14-3-3 complex (open monomer confo... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 9mmr | |||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Title | Cryo-EM structure of CRAF/MEK1/14-3-3 complex (open monomer conformation, CRAF Y340D/Y341D mutant) | |||||||||||||||||||||||||||
 Components Components |
| |||||||||||||||||||||||||||
 Keywords Keywords | TRANSFERASE / CRAF-MEK1-14-3-3 complex / MAPK pathway | |||||||||||||||||||||||||||
| Function / homology |  Function and homology information Function and homology informationdeath-inducing signaling complex assembly / epithelial cell proliferation involved in lung morphogenesis / positive regulation of endodermal cell differentiation / negative regulation of homotypic cell-cell adhesion / negative regulation of hypoxia-induced intrinsic apoptotic signaling pathway / regulation of vascular associated smooth muscle contraction / intermediate filament cytoskeleton organization / regulation of axon regeneration / mitogen-activated protein kinase kinase / positive regulation of muscle contraction ...death-inducing signaling complex assembly / epithelial cell proliferation involved in lung morphogenesis / positive regulation of endodermal cell differentiation / negative regulation of homotypic cell-cell adhesion / negative regulation of hypoxia-induced intrinsic apoptotic signaling pathway / regulation of vascular associated smooth muscle contraction / intermediate filament cytoskeleton organization / regulation of axon regeneration / mitogen-activated protein kinase kinase / positive regulation of muscle contraction / Golgi inheritance / placenta blood vessel development / MAP-kinase scaffold activity / cerebellar cortex formation / labyrinthine layer development / regulation of Rho protein signal transduction / melanosome transport / type B pancreatic cell proliferation / Signaling by MAP2K mutants / SHOC2 M1731 mutant abolishes MRAS complex function / Gain-of-function MRAS complexes activate RAF signaling / Rap1 signalling / vesicle transport along microtubule / positive regulation of Ras protein signal transduction / insulin secretion involved in cellular response to glucose stimulus / regulation of Golgi inheritance / central nervous system neuron differentiation / mitogen-activated protein kinase kinase kinase binding / positive regulation of axonogenesis / trachea formation / triglyceride homeostasis / Negative feedback regulation of MAPK pathway / regulation of early endosome to late endosome transport / IFNG signaling activates MAPKs / regulation of stress-activated MAPK cascade / Frs2-mediated activation / GP1b-IX-V activation signalling / MAPK3 (ERK1) activation / ERBB2-ERBB3 signaling pathway / neurotrophin TRK receptor signaling pathway / face development / regulation of neurotransmitter receptor localization to postsynaptic specialization membrane / endodermal cell differentiation / pseudopodium / MAP kinase kinase activity / Bergmann glial cell differentiation / positive regulation of ATP biosynthetic process / regulation of cell differentiation / thyroid gland development / Uptake and function of anthrax toxins / positive regulation of protein serine/threonine kinase activity / extrinsic apoptotic signaling pathway via death domain receptors / somatic stem cell population maintenance / positive regulation of peptidyl-serine phosphorylation / MAP kinase kinase kinase activity / protein kinase activator activity / type II interferon-mediated signaling pathway / Schwann cell development / response to axon injury / negative regulation of extrinsic apoptotic signaling pathway via death domain receptors / negative regulation of protein-containing complex assembly / keratinocyte differentiation / neuron projection morphogenesis / response to muscle stretch / ERK1 and ERK2 cascade / myelination / protein serine/threonine/tyrosine kinase activity / positive regulation of autophagy / CD209 (DC-SIGN) signaling / insulin-like growth factor receptor signaling pathway / dendrite cytoplasm / response to glucocorticoid / thymus development / adenylate cyclase activator activity / MAP3K8 (TPL2)-dependent MAPK1/3 activation / protein serine/threonine kinase activator activity / Signal transduction by L1 / cell motility / positive regulation of transcription elongation by RNA polymerase II / wound healing / RAF activation / Signaling by high-kinase activity BRAF mutants / MAP2K and MAPK activation / small GTPase binding / Stimuli-sensing channels / chemotaxis / neuron differentiation / Negative regulation of MAPK pathway / Signaling by RAF1 mutants / Signaling by moderate kinase activity BRAF mutants / Paradoxical activation of RAF signaling by kinase inactive BRAF / Signaling downstream of RAS mutants / cellular senescence / Signaling by BRAF and RAF1 fusions / insulin receptor signaling pathway / late endosome / MAPK cascade / heart development / response to oxidative stress / protein tyrosine kinase activity Similarity search - Function | |||||||||||||||||||||||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) Spodoptera exigua (beet armyworm) Spodoptera exigua (beet armyworm) | |||||||||||||||||||||||||||
| Method | ELECTRON MICROSCOPY / single particle reconstruction / cryo EM / Resolution: 3.9 Å | |||||||||||||||||||||||||||
 Authors Authors | Jang, D.M. / Jeon, H. / Eck, M.J. | |||||||||||||||||||||||||||
| Funding support |  United States, 1items United States, 1items
| |||||||||||||||||||||||||||
 Citation Citation |  Journal: Nat Commun / Year: 2025 Journal: Nat Commun / Year: 2025Title: Cryo-EM structures of CRAF/MEK1/14-3-3 complexes in autoinhibited and open-monomer states reveal features of RAF regulation. Authors: Dong Man Jang / Kayla Boxer / Byung Hak Ha / Emre Tkacik / Talya Levitz / Shaun Rawson / Rebecca J Metivier / Anna Schmoker / Hyesung Jeon / Michael J Eck /  Abstract: CRAF (RAF1) is one of three RAF-family kinases that initiate MAP kinase signaling in response to activated RAS and is essential for oncogenic signaling from mutant KRAS. Like BRAF, CRAF is regulated ...CRAF (RAF1) is one of three RAF-family kinases that initiate MAP kinase signaling in response to activated RAS and is essential for oncogenic signaling from mutant KRAS. Like BRAF, CRAF is regulated by 14-3-3 engagement and by intramolecular autoinhibitory interactions of its N-terminal regulatory region. Unlike BRAF, it is thought to require tyrosine phosphorylation in its N-terminal acidic (NtA) motif for full catalytic activation. Here we describe cryo-EM reconstructions of full-length CRAF in complex with MEK1 and a 14-3-3 dimer. These structures reveal a fully autoinhibited conformation analogous to that observed for BRAF and two "open monomer" states in which the inhibitory interactions of the CRD and 14-3-3 dimer are released or rearranged, but the kinase domain remains inactive. Structure-function studies of the NtA motif indicate that phosphorylation or acidic mutations in this segment increase catalytic activity by destabilizing the inactive conformation of the kinase domain. Collectively, these studies provide a structural foundation for understanding the shared and unique regulatory features of CRAF and will inform efforts to selectively block CRAF signaling in cancer. | |||||||||||||||||||||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  9mmr.cif.gz 9mmr.cif.gz | 208.9 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb9mmr.ent.gz pdb9mmr.ent.gz | 154.7 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  9mmr.json.gz 9mmr.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/mm/9mmr https://data.pdbj.org/pub/pdb/validation_reports/mm/9mmr ftp://data.pdbj.org/pub/pdb/validation_reports/mm/9mmr ftp://data.pdbj.org/pub/pdb/validation_reports/mm/9mmr | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  48401MC  9mmpC  9mmqC  9mmsC M: map data used to model this data C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
|
|---|---|
| 1 |
|
- Components
Components
-Protein , 3 types, 4 molecules ABCD
| #1: Protein | Mass: 76961.586 Da / Num. of mol.: 1 / Mutation: Q156R, Y340D, Y341D, D587E Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: RAF1, RAF / Production host: Homo sapiens (human) / Gene: RAF1, RAF / Production host:  References: UniProt: P04049, non-specific serine/threonine protein kinase |
|---|---|
| #2: Protein | Mass: 45934.543 Da / Num. of mol.: 1 / Mutation: S218A, S222A Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: MAP2K1, MEK1, PRKMK1 / Production host: Homo sapiens (human) / Gene: MAP2K1, MEK1, PRKMK1 / Production host:  References: UniProt: Q02750, mitogen-activated protein kinase kinase |
| #3: Protein | Mass: 28108.514 Da / Num. of mol.: 2 / Source method: isolated from a natural source / Source: (natural)  Spodoptera exigua (beet armyworm) / References: UniProt: V9P4T4 Spodoptera exigua (beet armyworm) / References: UniProt: V9P4T4 |
-Non-polymers , 3 types, 5 molecules 

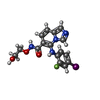


| #4: Chemical | | #5: Chemical | #6: Chemical | ChemComp-LCJ / | |
|---|
-Details
| Has ligand of interest | Y |
|---|---|
| Has protein modification | Y |
-Experimental details
-Experiment
| Experiment | Method: ELECTRON MICROSCOPY |
|---|---|
| EM experiment | Aggregation state: PARTICLE / 3D reconstruction method: single particle reconstruction |
- Sample preparation
Sample preparation
| Component |
| ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Molecular weight |
| ||||||||||||||||||||||||||||||
| Source (natural) |
| ||||||||||||||||||||||||||||||
| Source (recombinant) |
| ||||||||||||||||||||||||||||||
| Buffer solution | pH: 7.5 Details: 50 mM Tris pH 7.5, 150 mM NaCl, 2 mM MgCl2, 0.5 mM TCEP, 2 uM ATPgS, 1 uM GDC0623 | ||||||||||||||||||||||||||||||
| Specimen | Conc.: 0.4 mg/ml / Embedding applied: NO / Shadowing applied: NO / Staining applied: NO / Vitrification applied: YES | ||||||||||||||||||||||||||||||
| Vitrification | Instrument: LEICA EM GP / Cryogen name: ETHANE / Humidity: 95 % |
- Electron microscopy imaging
Electron microscopy imaging
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
|---|---|
| Microscopy | Model: TFS KRIOS |
| Electron gun | Electron source:  FIELD EMISSION GUN / Accelerating voltage: 300 kV / Illumination mode: FLOOD BEAM FIELD EMISSION GUN / Accelerating voltage: 300 kV / Illumination mode: FLOOD BEAM |
| Electron lens | Mode: OTHER / Nominal magnification: 165000 X / Nominal defocus max: 1800 nm / Nominal defocus min: 800 nm |
| Image recording | Electron dose: 56.65 e/Å2 / Film or detector model: FEI FALCON I (4k x 4k) / Num. of grids imaged: 1 / Num. of real images: 6060 / Details: tilt by 30 degree |
| EM imaging optics | Energyfilter name: TFS Selectris / Energyfilter slit width: 10 eV |
- Processing
Processing
| Software | Name: PHENIX / Version: 1.21.1_5286 / Classification: refinement | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EM software | Name: PHENIX / Version: 1.21.1_5286 / Category: model refinement | ||||||||||||||||||||||||
| CTF correction | Type: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||
| Symmetry | Point symmetry: C1 (asymmetric) | ||||||||||||||||||||||||
| 3D reconstruction | Resolution: 3.9 Å / Resolution method: FSC 0.143 CUT-OFF / Num. of particles: 131061 / Details: The final resolution was calculated without a mask / Symmetry type: POINT | ||||||||||||||||||||||||
| Atomic model building | Space: REAL | ||||||||||||||||||||||||
| Refinement | Cross valid method: NONE Stereochemistry target values: GeoStd + Monomer Library + CDL v1.2 | ||||||||||||||||||||||||
| Displacement parameters | Biso mean: 183.87 Å2 | ||||||||||||||||||||||||
| Refine LS restraints |
|
 Movie
Movie Controller
Controller





 PDBj
PDBj














