+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 9iol | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the complex of DNA, Ku70/80, and laXLF. | |||||||||||||||||||||
 要素 要素 |
| |||||||||||||||||||||
 キーワード キーワード | DNA BINDING PROTEIN/DNA / DNA repair / NHEJ / Complex / Lactylation / DNA BINDING PROTEIN / DNA BINDING PROTEIN-DNA complex | |||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of ligase activity / DNA ligase IV complex / Ku70:Ku80 complex / negative regulation of t-circle formation / DNA end binding / small-subunit processome assembly / positive regulation of lymphocyte differentiation / DNA-dependent protein kinase complex / DNA-dependent protein kinase-DNA ligase 4 complex / nonhomologous end joining complex ...positive regulation of ligase activity / DNA ligase IV complex / Ku70:Ku80 complex / negative regulation of t-circle formation / DNA end binding / small-subunit processome assembly / positive regulation of lymphocyte differentiation / DNA-dependent protein kinase complex / DNA-dependent protein kinase-DNA ligase 4 complex / nonhomologous end joining complex / immunoglobulin V(D)J recombination / regulation of smooth muscle cell proliferation / cellular response to X-ray / nuclear telomere cap complex / double-strand break repair via classical nonhomologous end joining / Cytosolic sensors of pathogen-associated DNA / IRF3-mediated induction of type I IFN / positive regulation of neurogenesis / regulation of telomere maintenance / recombinational repair / U3 snoRNA binding / protein localization to chromosome, telomeric region / response to ionizing radiation / cellular hyperosmotic salinity response / 2-LTR circle formation / telomeric DNA binding / hematopoietic stem cell proliferation / positive regulation of protein kinase activity / site of DNA damage / 付加脱離酵素(リアーゼ); 炭素-酸素リアーゼ類; その他の炭素-酸素リアーゼ / T cell differentiation / 5'-deoxyribose-5-phosphate lyase activity / hematopoietic stem cell differentiation / ATP-dependent activity, acting on DNA / telomere maintenance via telomerase / DNA polymerase binding / neurogenesis / telomere maintenance / activation of innate immune response / DNA helicase activity / cyclin binding / cellular response to leukemia inhibitory factor / B cell differentiation / central nervous system development / small-subunit processome / Nonhomologous End-Joining (NHEJ) / enzyme activator activity / cellular response to gamma radiation / protein-DNA complex / double-strand break repair via nonhomologous end joining / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / fibrillar center / site of double-strand break / double-strand break repair / double-stranded DNA binding / scaffold protein binding / secretory granule lumen / DNA recombination / transcription regulator complex / damaged DNA binding / ficolin-1-rich granule lumen / chromosome, telomeric region / transcription cis-regulatory region binding / ribonucleoprotein complex / innate immune response / negative regulation of DNA-templated transcription / ubiquitin protein ligase binding / DNA damage response / Neutrophil degranulation / positive regulation of DNA-templated transcription / protein-containing complex binding / nucleolus / positive regulation of transcription by RNA polymerase II / protein-containing complex / ATP hydrolysis activity / DNA binding / RNA binding / extracellular region / nucleoplasm / ATP binding / nucleus / membrane / plasma membrane / cytosol 類似検索 - 分子機能 | |||||||||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.46 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Liang, S. | |||||||||||||||||||||
| 資金援助 |  香港, 1件 香港, 1件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2025 ジャーナル: Mol Cell / 年: 2025タイトル: Lactylation of XLF promotes non-homologous end-joining repair and chemoresistance in cancer. 著者: Mingpeng Jin / Bingsong Huang / Xiaoning Yang / Shuyang Wang / Jinhuan Wu / Yiming He / Xin Ding / Xuanhe Wang / Zhe Wang / Jie Yang / Rui Li / Xuan Zhou / Qianwen Wang / Yunhui Li / Lei Li / ...著者: Mingpeng Jin / Bingsong Huang / Xiaoning Yang / Shuyang Wang / Jinhuan Wu / Yiming He / Xin Ding / Xuanhe Wang / Zhe Wang / Jie Yang / Rui Li / Xuan Zhou / Qianwen Wang / Yunhui Li / Lei Li / Wen Zheng / Zhikai Zeng / Chenxi Zhao / Jiaqi Liu / Qian Zhu / Zhihua Kang / Ke Li / Shikang Liang / Yuping Chen / Jian Yuan /  要旨: Metabolic reprogramming and DNA damage repair are essential in tumorigenesis and chemoresistance, yet their link remains elusive. Here, we show that LDHA deficiency impairs NHEJ and class switch ...Metabolic reprogramming and DNA damage repair are essential in tumorigenesis and chemoresistance, yet their link remains elusive. Here, we show that LDHA deficiency impairs NHEJ and class switch recombination. Additionally, glycolysis-derived lactate promotes XLF lactylation at K288 within its Ku-binding motif (X-KBM) to regulate NHEJ. Mechanistically, DNA damage triggers ATM-mediated GCN5 phosphorylation to increase GCN5-XLF interaction and XLF lactylation, enhancing XLF-Ku80 binding, XLF recruitment to DSBs, and NHEJ efficiency. Cryo-EM structural analysis demonstrates that lactylated X-KBM (laX-KBM) forms a more extensive interface with Ku70/80, inducing conformational changes in the Ku80 vWA domain. XLF lactylation deficiency impairs NHEJ and sensitizes cancer cells to chemotherapy. A specific XLF K288 lactylation peptide inhibitor plus 5-fluorouracil synergistically kills colorectal cancer cells in PDX models with XLF hyperlactylation. These findings highlight that the GCN5-XLF lactylation axis is a critical NHEJ regulator and that targeting XLF lactylation can improve chemotherapy efficiency. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  9iol.cif.gz 9iol.cif.gz | 223.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb9iol.ent.gz pdb9iol.ent.gz | 169.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  9iol.json.gz 9iol.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  9iol_validation.pdf.gz 9iol_validation.pdf.gz | 1.2 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  9iol_full_validation.pdf.gz 9iol_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  9iol_validation.xml.gz 9iol_validation.xml.gz | 37.5 KB | 表示 | |
| CIF形式データ |  9iol_validation.cif.gz 9iol_validation.cif.gz | 58 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/io/9iol https://data.pdbj.org/pub/pdb/validation_reports/io/9iol ftp://data.pdbj.org/pub/pdb/validation_reports/io/9iol ftp://data.pdbj.org/pub/pdb/validation_reports/io/9iol | HTTPS FTP |
-関連構造データ
| 関連構造データ |  60744MC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-X-ray repair cross-complementing protein ... , 2種, 2分子 AB
| #1: タンパク質 | 分子量: 82812.438 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: XRCC5, G22P2 Homo sapiens (ヒト) / 遺伝子: XRCC5, G22P2発現宿主:  参照: UniProt: P13010, 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 |
|---|---|
| #2: タンパク質 | 分子量: 69945.039 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: XRCC6, G22P1 Homo sapiens (ヒト) / 遺伝子: XRCC6, G22P1発現宿主:  参照: UniProt: P12956, 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与, 付加脱離酵素(リアーゼ); 炭素- ...参照: UniProt: P12956, 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与, 付加脱離酵素(リアーゼ); 炭素-酸素リアーゼ類; その他の炭素-酸素リアーゼ |
-DNA鎖 , 2種, 2分子 CD
| #3: DNA鎖 | 分子量: 6969.472 Da / 分子数: 1 / 由来タイプ: 合成 / 由来: (合成)  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|---|
| #4: DNA鎖 | 分子量: 7156.611 Da / 分子数: 1 / 由来タイプ: 合成 / 由来: (合成)  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-タンパク質・ペプチド , 1種, 1分子 M
| #5: タンパク質・ペプチド | 分子量: 1536.907 Da / 分子数: 1 / 由来タイプ: 合成 / 由来: (合成)  Homo sapiens (ヒト) / 参照: UniProt: Q9H9Q4 Homo sapiens (ヒト) / 参照: UniProt: Q9H9Q4 |
|---|
-非ポリマー , 2種, 2分子 
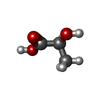

| #6: 化合物 | ChemComp-IHP / |
|---|---|
| #7: 化合物 | ChemComp-2OP / ( |
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|---|
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: The complex of DNA, Ku70/80, and laXLF / タイプ: COMPLEX / Entity ID: #1-#5 / 由来: MULTIPLE SOURCES |
|---|---|
| 分子量 | 値: 0.168 MDa / 実験値: NO |
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 由来(組換発現) | 生物種:  |
| 緩衝液 | pH: 7.6 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: TFS KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2500 nm / 最小 デフォーカス(公称値): 1000 nm |
| 撮影 | 電子線照射量: 50 e/Å2 フィルム・検出器のモデル: TFS FALCON 4i (4k x 4k) |
- 解析
解析
| EMソフトウェア | 名称: cryoSPARC / カテゴリ: CTF補正 |
|---|---|
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION |
| 3次元再構成 | 解像度: 3.46 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 298238 / 対称性のタイプ: POINT |
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj
















































