+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 9c9m | |||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | HIV-1 intasome core bound with DTG | |||||||||||||||||||||||||||||||||||||||||||||
 要素 要素 |
| |||||||||||||||||||||||||||||||||||||||||||||
 キーワード キーワード | VIRAL PROTEIN / HIV-1 / integrase / nucleoprotein complexes / INSTI / intasome / CryoEM | |||||||||||||||||||||||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報HIV-1 retropepsin / symbiont-mediated activation of host apoptosis / retroviral ribonuclease H / exoribonuclease H / exoribonuclease H activity / DNA integration / viral genome integration into host DNA / RNA-directed DNA polymerase / establishment of integrated proviral latency / RNA stem-loop binding ...HIV-1 retropepsin / symbiont-mediated activation of host apoptosis / retroviral ribonuclease H / exoribonuclease H / exoribonuclease H activity / DNA integration / viral genome integration into host DNA / RNA-directed DNA polymerase / establishment of integrated proviral latency / RNA stem-loop binding / host multivesicular body / viral penetration into host nucleus / RNA-directed DNA polymerase activity / RNA-DNA hybrid ribonuclease activity / 転移酵素; リンを含む基を移すもの; 核酸を移すもの / host cell / viral nucleocapsid / DNA recombination / DNA-directed DNA polymerase / aspartic-type endopeptidase activity / 加水分解酵素; エステル加水分解酵素 / DNA-directed DNA polymerase activity / symbiont-mediated suppression of host gene expression / viral translational frameshifting / lipid binding / symbiont entry into host cell / host cell nucleus / host cell plasma membrane / virion membrane / structural molecule activity / proteolysis / DNA binding / zinc ion binding / membrane 類似検索 - 分子機能 | |||||||||||||||||||||||||||||||||||||||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) | |||||||||||||||||||||||||||||||||||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.01 Å | |||||||||||||||||||||||||||||||||||||||||||||
 データ登録者 データ登録者 | Li, M. / Craigie, R. | |||||||||||||||||||||||||||||||||||||||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||||||||||||||||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Viruses / 年: 2024 ジャーナル: Viruses / 年: 2024タイトル: HIV-1 Intasomes Assembled with Excess Integrase C-Terminal Domain Protein Facilitate Structural Studies by Cryo-EM and Reveal the Role of the Integrase C-Terminal Tail in HIV-1 Integration. 著者: Min Li / Zhen Li / Xuemin Chen / Yanxiang Cui / Alan N Engelman / Robert Craigie /   要旨: Retroviral integration is mediated by intasome nucleoprotein complexes wherein a pair of viral DNA ends are bridged together by a multimer of integrase (IN). Atomic-resolution structures of HIV-1 ...Retroviral integration is mediated by intasome nucleoprotein complexes wherein a pair of viral DNA ends are bridged together by a multimer of integrase (IN). Atomic-resolution structures of HIV-1 intasomes provide detailed insights into the mechanism of integration and inhibition by clinical IN inhibitors. However, previously described HIV-1 intasomes are highly heterogeneous and have the tendency to form stacks, which is a limiting factor in determining high-resolution cryo-EM maps. We have assembled HIV-1 intasomes in the presence of excess IN C-terminal domain protein, which was readily incorporated into the intasomes. The purified intasomes were largely homogeneous and exhibited minimal stacking tendencies. The cryo-EM map resolution was further improved to 2.01 Å, which will greatly facilitate structural studies of IN inhibitor action and drug resistance mechanisms. The C-terminal 18 residues of HIV-1 IN, which are critical for virus replication and integration in vitro, have not been well resolved in previous intasome structures, and its function remains unclear. We show that the C-terminal tail participates in intasome assembly, resides within the intasome core, and forms a small alpha helix (residues 271-276). Mutations that disrupt alpha helix integrity impede IN activity in vitro and disrupt HIV-1 infection at the step of viral DNA integration. | |||||||||||||||||||||||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  9c9m.cif.gz 9c9m.cif.gz | 313.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb9c9m.ent.gz pdb9c9m.ent.gz | 238.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  9c9m.json.gz 9c9m.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  9c9m_validation.pdf.gz 9c9m_validation.pdf.gz | 1.3 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  9c9m_full_validation.pdf.gz 9c9m_full_validation.pdf.gz | 1.3 MB | 表示 | |
| XML形式データ |  9c9m_validation.xml.gz 9c9m_validation.xml.gz | 50.8 KB | 表示 | |
| CIF形式データ |  9c9m_validation.cif.gz 9c9m_validation.cif.gz | 77.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/c9/9c9m https://data.pdbj.org/pub/pdb/validation_reports/c9/9c9m ftp://data.pdbj.org/pub/pdb/validation_reports/c9/9c9m ftp://data.pdbj.org/pub/pdb/validation_reports/c9/9c9m | HTTPS FTP |
-関連構造データ
| 関連構造データ |  45364MC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-タンパク質 , 2種, 8分子 ABIKLCDM
| #1: タンパク質 | 分子量: 39864.641 Da / 分子数: 6 / 由来タイプ: 組換発現 由来: (組換発現)   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)遺伝子: gag-pol / 発現宿主:  参照: UniProt: P12497, 転移酵素; リンを含む基を移すもの; 核酸を移すもの, 加水分解酵素; エステル加水分解酵素 #2: タンパク質 | 分子量: 12101.872 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)遺伝子: gag-pol / 発現宿主:  参照: UniProt: P12497, 転移酵素; リンを含む基を移すもの; 核酸を移すもの, 加水分解酵素; エステル加水分解酵素 |
|---|
-DNA鎖 , 2種, 4分子 ENFO
| #3: DNA鎖 | 分子量: 8188.271 Da / 分子数: 2 / 由来タイプ: 合成 由来: (合成)   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)#4: DNA鎖 | 分子量: 7773.023 Da / 分子数: 2 / 由来タイプ: 合成 由来: (合成)   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
|---|
-非ポリマー , 4種, 89分子 

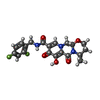




| #5: 化合物 | ChemComp-MG / #6: 化合物 | #7: 化合物 | #8: 水 | ChemComp-HOH / | |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|---|
| Has protein modification | N |
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: HIV-1 intasomes Core / タイプ: COMPLEX 詳細: HIV-1 intasomes assembled with full length integrase and integrase c-terminal domain Entity ID: #1-#4 / 由来: RECOMBINANT |
|---|---|
| 分子量 | 実験値: NO |
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 由来(組換発現) | 生物種:  |
| 緩衝液 | pH: 6.2 |
| 試料 | 濃度: 0.4 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 105000 X / 最大 デフォーカス(公称値): 2600 nm / 最小 デフォーカス(公称値): 600 nm / Cs: 2.7 mm / C2レンズ絞り径: 70 µm / アライメント法: COMA FREE |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 電子線照射量: 54 e/Å2 / 検出モード: SUPER-RESOLUTION フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) |
| 電子光学装置 | エネルギーフィルター名称: GIF Bioquantum / エネルギーフィルタースリット幅: 20 eV |
- 解析
解析
| EMソフトウェア |
| |||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: NONE | |||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 940736 | |||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 2.01 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 428779 / 対称性のタイプ: POINT | |||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: RIGID BODY FIT / 空間: REAL | |||||||||||||||||||||||||||
| 原子モデル構築 | PDB-ID: 5U1C Accession code: 5U1C / Source name: PDB / タイプ: experimental model |
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj











































