+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8yn0 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of NRG1C in complex with EDS1-SAG101-(ADPr-ATP) | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | PLANT PROTEIN/HYDROLASE / EDS1 / SAG101 / ATP-ADPR / NRG1C / PLANT PROTEIN / PLANT PROTEIN-HYDROLASE complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of defense response to oomycetes / positive regulation of leaf senescence / aerenchyma formation / EDS1 disease-resistance complex / leaf abscission / systemic acquired resistance, salicylic acid mediated signaling pathway / systemic acquired resistance / plant-type hypersensitive response / defense response to other organism / response to singlet oxygen ...positive regulation of defense response to oomycetes / positive regulation of leaf senescence / aerenchyma formation / EDS1 disease-resistance complex / leaf abscission / systemic acquired resistance, salicylic acid mediated signaling pathway / systemic acquired resistance / plant-type hypersensitive response / defense response to other organism / response to singlet oxygen / positive regulation of defense response to bacterium / lipase activity / carboxylesterase / regulation of hydrogen peroxide metabolic process / carboxylesterase activity / carboxylic ester hydrolase activity / lipid catabolic process / positive regulation of defense response to virus by host / chloroplast / lipid metabolic process / defense response to Gram-negative bacterium / response to hypoxia / endoplasmic reticulum / protein homodimerization activity / nucleus / membrane / cytosol / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.49 Å 分子置換 / 解像度: 2.49 Å | ||||||
 データ登録者 データ登録者 | Huang, S. / Xiao, Y. / Chai, J. | ||||||
| 資金援助 |  中国, 1件 中国, 1件
| ||||||
 引用 引用 |  ジャーナル: Nature / 年: 2025 ジャーナル: Nature / 年: 2025タイトル: Balanced plant helper NLR activation by a modified host protein complex. 著者: Shijia Huang / Junli Wang / Ridan Song / Aolin Jia / Yu Xiao / Yue Sun / Lin Wang / Dennis Mahr / Zhongshou Wu / Zhifu Han / Xin Li / Jane E Parker / Jijie Chai /    要旨: Nucleotide-binding leucine-rich repeat (NLR) receptors play crucial roles in plant immunity by sensing pathogen effectors. In Arabidopsis, certain sensor NLRs function as NADases to catalyse the ...Nucleotide-binding leucine-rich repeat (NLR) receptors play crucial roles in plant immunity by sensing pathogen effectors. In Arabidopsis, certain sensor NLRs function as NADases to catalyse the production of second messengers, which can be recognized by enhanced disease susceptibility 1 (EDS1) with its partner senescence-associated gene 101 (SAG101), to activate helper NLR N requirement gene 1 (NRG1). A cryoelectron microscopy structure shows that second-messenger-activated EDS1-SAG101 mainly contacts the leucine-rich repeat domain of NRG1A to mediate the formation of an induced EDS1-SAG101-NRG1A complex. Structural comparisons show that binding of a second messenger induces conformational changes in EDS1-SAG101, which are recognized by NRG1A, leading to its allosteric activation. We further show that an inhibitory NRG1 family member, NRG1C, efficiently outcompetes NRG1A for binding to second-messenger-activated EDS1-SAG101. These findings uncover mechanisms for NRG1A activation through its recognition of a modified host EDS1-SAG101 complex, and NRG1A inhibition by NRG1C through sequestration of the activated EDS1-SAG101, thus shedding light on the activation and constraint of a central plant immune response system. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8yn0.cif.gz 8yn0.cif.gz | 651 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8yn0.ent.gz pdb8yn0.ent.gz | 525.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8yn0.json.gz 8yn0.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/yn/8yn0 https://data.pdbj.org/pub/pdb/validation_reports/yn/8yn0 ftp://data.pdbj.org/pub/pdb/validation_reports/yn/8yn0 ftp://data.pdbj.org/pub/pdb/validation_reports/yn/8yn0 | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  8yn1C C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 3種, 6分子 BAECHD
| #1: タンパク質 | 分子量: 71093.438 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: EDS1, EDS1-90, EDS1A, At3g48090, T17F15.40 発現宿主:  参照: UniProt: Q9SU72 #2: タンパク質 | 分子量: 62022.195 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: SAG101, At5g14930, F2G14.50 発現宿主:  参照: UniProt: Q4F883, carboxylesterase #3: タンパク質 | 分子量: 47602.734 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)  遺伝子: At5g66890, MUD21.15 発現宿主:  参照: UniProt: Q9FKZ2 |
|---|
-非ポリマー , 3種, 708分子 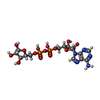




| #4: 化合物 | | #5: 化合物 | #6: 水 | ChemComp-HOH / | |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|---|
| Has protein modification | N |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.38 Å3/Da / 溶媒含有率: 63.64 % |
|---|---|
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, ハンギングドロップ法 詳細: 5% v/v (+/-)-2-Methyl-2,4-pentanediol, 0.1 M HEPES pH 7.5, 10 % w/v Polyethylene glycol 10000 |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SSRF SSRF  / ビームライン: BL19U1 / 波長: 0.979 Å / ビームライン: BL19U1 / 波長: 0.979 Å |
| 検出器 | タイプ: DECTRIS PILATUS 6M / 検出器: PIXEL / 日付: 2022年1月1日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.979 Å / 相対比: 1 |
| 反射 | 解像度: 2.49→50 Å / Num. obs: 159460 / % possible obs: 96.1 % / 冗長度: 3 % / Rrim(I) all: 0.15 / Net I/σ(I): 9.1 |
| 反射 シェル | 解像度: 2.49→2.54 Å / Mean I/σ(I) obs: 1.3 / Num. unique obs: 7943 / Rrim(I) all: 0.99 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 / 解像度: 2.49→39.77 Å / SU ML: 0.29 / 交差検証法: FREE R-VALUE / σ(F): 1.35 / 位相誤差: 25.27 / 立体化学のターゲット値: ML 分子置換 / 解像度: 2.49→39.77 Å / SU ML: 0.29 / 交差検証法: FREE R-VALUE / σ(F): 1.35 / 位相誤差: 25.27 / 立体化学のターゲット値: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.49→39.77 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
|
 ムービー
ムービー コントローラー
コントローラー




 PDBj
PDBj


