| 登録情報 | データベース: PDB / ID: 8xsb
|
|---|
| タイトル | Crystal structure of the DNA-bound AHR-ARNT heterodimer in complex with Indirubin |
|---|
 要素 要素 | - (Aryl hydrocarbon ...) x 2
- DNAF
- DNAR
|
|---|
 キーワード キーワード | TRANSCRIPTION / Aryl hydrocarbon receptor / Indirubin / bHLH-PAS |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
Phase I - Functionalization of compounds / Endogenous sterols / Xenobiotics / Aryl hydrocarbon receptor signalling / regulation of B cell proliferation / cellular response to 2,3,7,8-tetrachlorodibenzodioxine / nuclear aryl hydrocarbon receptor complex / cellular response to molecule of bacterial origin / positive regulation of hormone biosynthetic process / Aryl hydrocarbon receptor signalling ...Phase I - Functionalization of compounds / Endogenous sterols / Xenobiotics / Aryl hydrocarbon receptor signalling / regulation of B cell proliferation / cellular response to 2,3,7,8-tetrachlorodibenzodioxine / nuclear aryl hydrocarbon receptor complex / cellular response to molecule of bacterial origin / positive regulation of hormone biosynthetic process / Aryl hydrocarbon receptor signalling / aryl hydrocarbon receptor complex / negative regulation of T cell mediated immune response to tumor cell / positive regulation of protein sumoylation / Xenobiotics / Phase I - Functionalization of compounds / positive regulation of vascular endothelial growth factor receptor signaling pathway / Regulation of gene expression by Hypoxia-inducible Factor / aryl hydrocarbon receptor binding / TFIID-class transcription factor complex binding / positive regulation of vascular endothelial growth factor production / Endogenous sterols / embryonic placenta development / cis-regulatory region sequence-specific DNA binding / NPAS4 regulates expression of target genes / positive regulation of endothelial cell proliferation / cellular response to forskolin / xenobiotic metabolic process / TBP-class protein binding / positive regulation of erythrocyte differentiation / cellular response to cAMP / positive regulation of glycolytic process / Hsp90 protein binding / PPARA activates gene expression / response to toxic substance / negative regulation of inflammatory response / RNA polymerase II transcription regulator complex / transcription coactivator binding / nuclear receptor activity / sequence-specific double-stranded DNA binding / rhythmic process / cellular response to oxidative stress / sequence-specific DNA binding / RNA polymerase II-specific DNA-binding transcription factor binding / DNA-binding transcription factor activity, RNA polymerase II-specific / cell differentiation / response to hypoxia / transcription cis-regulatory region binding / nuclear body / RNA polymerase II cis-regulatory region sequence-specific DNA binding / DNA-binding transcription factor activity / protein heterodimerization activity / regulation of transcription by RNA polymerase II / chromatin / protein homodimerization activity / positive regulation of transcription by RNA polymerase II / nucleoplasm / nucleus / cytoplasm / cytosol類似検索 - 分子機能 Aryl hydrocarbon receptor / Aryl hydrocarbon receptor/Aryl hydrocarbon receptor repressor / : / Nuclear translocator / PAS fold-3 / PAS fold / Helix-loop-helix DNA-binding domain / PAC motif / Motif C-terminal to PAS motifs (likely to contribute to PAS structural domain) / PAS domain ...Aryl hydrocarbon receptor / Aryl hydrocarbon receptor/Aryl hydrocarbon receptor repressor / : / Nuclear translocator / PAS fold-3 / PAS fold / Helix-loop-helix DNA-binding domain / PAC motif / Motif C-terminal to PAS motifs (likely to contribute to PAS structural domain) / PAS domain / helix loop helix domain / Myc-type, basic helix-loop-helix (bHLH) domain / Myc-type, basic helix-loop-helix (bHLH) domain profile. / Helix-loop-helix DNA-binding domain superfamily / PAS fold / PAS fold / PAS domain / PAS repeat profile. / PAS domain / PAS domain superfamily類似検索 - ドメイン・相同性 Chem-JY6 / DNA / DNA (> 10) / Aryl hydrocarbon receptor / Aryl hydrocarbon receptor nuclear translocator類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト)
  Sus scrofa (ブタ) Sus scrofa (ブタ) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 3.06 Å 分子置換 / 解像度: 3.06 Å |
|---|
 データ登録者 データ登録者 | Diao, X. / Shang, Q. / Wu, D. |
|---|
| 資金援助 |  中国, 1件 中国, 1件 | 組織 | 認可番号 | 国 |
|---|
| National Natural Science Foundation of China (NSFC) | 82373876 |  中国 中国 |
|
|---|
 引用 引用 |  ジャーナル: Nat Commun / 年: 2025 ジャーナル: Nat Commun / 年: 2025
タイトル: Structural basis for the ligand-dependent activation of heterodimeric AHR-ARNT complex.
著者: Diao, X. / Shang, Q. / Guo, M. / Huang, Y. / Zhang, M. / Chen, X. / Liang, Y. / Sun, X. / Zhou, F. / Zhuang, J. / Liu, S.J. / Vogel, C.F.A. / Rastinejad, F. / Wu, D. |
|---|
| 履歴 | | 登録 | 2024年1月9日 | 登録サイト: PDBJ / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2025年1月15日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2025年2月19日 | Group: Database references / カテゴリ: citation / citation_author
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 3.06 Å
分子置換 / 解像度: 3.06 Å  データ登録者
データ登録者 中国, 1件
中国, 1件  引用
引用 ジャーナル: Nat Commun / 年: 2025
ジャーナル: Nat Commun / 年: 2025 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 8xsb.cif.gz
8xsb.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb8xsb.ent.gz
pdb8xsb.ent.gz PDB形式
PDB形式 8xsb.json.gz
8xsb.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 8xsb_validation.pdf.gz
8xsb_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 8xsb_full_validation.pdf.gz
8xsb_full_validation.pdf.gz 8xsb_validation.xml.gz
8xsb_validation.xml.gz 8xsb_validation.cif.gz
8xsb_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/xs/8xsb
https://data.pdbj.org/pub/pdb/validation_reports/xs/8xsb ftp://data.pdbj.org/pub/pdb/validation_reports/xs/8xsb
ftp://data.pdbj.org/pub/pdb/validation_reports/xs/8xsb リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: ARNT / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: ARNT / 発現宿主: 


 Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト)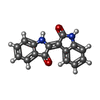


 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SSRF
SSRF  / ビームライン: BL18U1 / 波長: 0.979 Å
/ ビームライン: BL18U1 / 波長: 0.979 Å 解析
解析 分子置換 / 解像度: 3.06→36 Å / SU ML: 0.43 / 交差検証法: FREE R-VALUE / σ(F): 1.35 / 位相誤差: 26.21 / 立体化学のターゲット値: ML
分子置換 / 解像度: 3.06→36 Å / SU ML: 0.43 / 交差検証法: FREE R-VALUE / σ(F): 1.35 / 位相誤差: 26.21 / 立体化学のターゲット値: ML ムービー
ムービー コントローラー
コントローラー








 PDBj
PDBj











































