+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8xls | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | PSI-FCPI of the diatom Thalassiosira pseudonana CCMP1335 | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | PHOTOSYNTHESIS / Photosystem I / ELECTRON TRANSPORT | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報light-harvesting complex / photosynthesis, light harvesting in photosystem I / photosynthesis, light harvesting / photosystem I reaction center / photosystem I / photosynthetic electron transport in photosystem I / photosystem I / chlorophyll binding / chloroplast thylakoid membrane / response to light stimulus ...light-harvesting complex / photosynthesis, light harvesting in photosystem I / photosynthesis, light harvesting / photosystem I reaction center / photosystem I / photosynthetic electron transport in photosystem I / photosystem I / chlorophyll binding / chloroplast thylakoid membrane / response to light stimulus / photosynthesis / chloroplast / 4 iron, 4 sulfur cluster binding / electron transfer activity / oxidoreductase activity / magnesium ion binding / metal ion binding / membrane 類似検索 - 分子機能 | ||||||
| 生物種 |  Thalassiosira pseudonana CCMP1335 (珪藻) Thalassiosira pseudonana CCMP1335 (珪藻) | ||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.3 Å | ||||||
 データ登録者 データ登録者 | Kato, K. / Nakajima, Y. / Shen, J.R. / Nagao, R. | ||||||
| 資金援助 | 1件
| ||||||
 引用 引用 |  ジャーナル: Elife / 年: 2024 ジャーナル: Elife / 年: 2024タイトル: Structural basis for molecular assembly of fucoxanthin chlorophyll /-binding proteins in a diatom photosystem I supercomplex. 著者: Koji Kato / Yoshiki Nakajima / Jian Xing / Minoru Kumazawa / Haruya Ogawa / Jian-Ren Shen / Kentaro Ifuku / Ryo Nagao /  要旨: Photosynthetic organisms exhibit remarkable diversity in their light-harvesting complexes (LHCs). LHCs are associated with photosystem I (PSI), forming a PSI-LHCI supercomplex. The number of LHCI ...Photosynthetic organisms exhibit remarkable diversity in their light-harvesting complexes (LHCs). LHCs are associated with photosystem I (PSI), forming a PSI-LHCI supercomplex. The number of LHCI subunits, along with their protein sequences and pigment compositions, has been found to differ greatly among the PSI-LHCI structures. However, the mechanisms by which LHCIs recognize their specific binding sites within the PSI core remain unclear. In this study, we determined the cryo-electron microscopy structure of a PSI supercomplex incorporating fucoxanthin chlorophyll /-binding proteins (FCPs), designated as PSI-FCPI, isolated from the diatom CCMP1335. Structural analysis of PSI-FCPI revealed five FCPI subunits associated with a PSI monomer; these subunits were identified as RedCAP, Lhcr3, Lhcq10, Lhcf10, and Lhcq8. Through structural and sequence analyses, we identified specific protein-protein interactions at the interfaces between FCPI and PSI subunits, as well as among FCPI subunits themselves. Comparative structural analyses of PSI-FCPI supercomplexes, combined with phylogenetic analysis of FCPs from and the diatom , underscore the evolutionary conservation of protein motifs crucial for the selective binding of individual FCPI subunits. These findings provide significant insights into the molecular mechanisms underlying the assembly and selective binding of FCPIs in diatoms. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8xls.cif.gz 8xls.cif.gz | 959.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8xls.ent.gz pdb8xls.ent.gz | 829.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8xls.json.gz 8xls.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  8xls_validation.pdf.gz 8xls_validation.pdf.gz | 10.9 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  8xls_full_validation.pdf.gz 8xls_full_validation.pdf.gz | 12.5 MB | 表示 | |
| XML形式データ |  8xls_validation.xml.gz 8xls_validation.xml.gz | 237.2 KB | 表示 | |
| CIF形式データ |  8xls_validation.cif.gz 8xls_validation.cif.gz | 293.1 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/xl/8xls https://data.pdbj.org/pub/pdb/validation_reports/xl/8xls ftp://data.pdbj.org/pub/pdb/validation_reports/xl/8xls ftp://data.pdbj.org/pub/pdb/validation_reports/xl/8xls | HTTPS FTP |
-関連構造データ
| 関連構造データ |  38457MC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-Photosystem I P700 chlorophyll a apoprotein ... , 2種, 2分子 AB
| #1: タンパク質 | 分子量: 83725.828 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Thalassiosira pseudonana CCMP1335 (珪藻) / 参照: UniProt: A0T0M8, photosystem I Thalassiosira pseudonana CCMP1335 (珪藻) / 参照: UniProt: A0T0M8, photosystem I |
|---|---|
| #2: タンパク質 | 分子量: 82173.875 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Thalassiosira pseudonana CCMP1335 (珪藻) / 参照: UniProt: A0T0M9, photosystem I Thalassiosira pseudonana CCMP1335 (珪藻) / 参照: UniProt: A0T0M9, photosystem I |
-タンパク質 , 3種, 3分子 Cu3
| #3: タンパク質 | 分子量: 8807.169 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Thalassiosira pseudonana CCMP1335 (珪藻) / 参照: UniProt: A0T0W4, photosystem I Thalassiosira pseudonana CCMP1335 (珪藻) / 参照: UniProt: A0T0W4, photosystem I |
|---|---|
| #12: タンパク質 | 分子量: 7166.825 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Thalassiosira pseudonana CCMP1335 (珪藻) Thalassiosira pseudonana CCMP1335 (珪藻) |
| #15: タンパク質 | 分子量: 21210.223 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Thalassiosira pseudonana CCMP1335 (珪藻) / 参照: UniProt: B8CEQ3 Thalassiosira pseudonana CCMP1335 (珪藻) / 参照: UniProt: B8CEQ3 |
-Photosystem I reaction center subunit ... , 8種, 8分子 DEFIJLMW
| #4: タンパク質 | 分子量: 15535.668 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Thalassiosira pseudonana CCMP1335 (珪藻) / 参照: UniProt: A0T0T5 Thalassiosira pseudonana CCMP1335 (珪藻) / 参照: UniProt: A0T0T5 |
|---|---|
| #5: タンパク質 | 分子量: 7520.497 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Thalassiosira pseudonana CCMP1335 (珪藻) / 参照: UniProt: A0T0P9 Thalassiosira pseudonana CCMP1335 (珪藻) / 参照: UniProt: A0T0P9 |
| #6: タンパク質 | 分子量: 20382.461 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Thalassiosira pseudonana CCMP1335 (珪藻) / 参照: UniProt: A0T0V0 Thalassiosira pseudonana CCMP1335 (珪藻) / 参照: UniProt: A0T0V0 |
| #7: タンパク質・ペプチド | 分子量: 3919.688 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Thalassiosira pseudonana CCMP1335 (珪藻) / 参照: UniProt: A0T0S9 Thalassiosira pseudonana CCMP1335 (珪藻) / 参照: UniProt: A0T0S9 |
| #8: タンパク質・ペプチド | 分子量: 4854.705 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Thalassiosira pseudonana CCMP1335 (珪藻) / 参照: UniProt: A0T0V1 Thalassiosira pseudonana CCMP1335 (珪藻) / 参照: UniProt: A0T0V1 |
| #9: タンパク質 | 分子量: 15715.207 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Thalassiosira pseudonana CCMP1335 (珪藻) / 参照: UniProt: A0T0U5 Thalassiosira pseudonana CCMP1335 (珪藻) / 参照: UniProt: A0T0U5 |
| #10: タンパク質・ペプチド | 分子量: 3271.954 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Thalassiosira pseudonana CCMP1335 (珪藻) / 参照: UniProt: A0T0S1 Thalassiosira pseudonana CCMP1335 (珪藻) / 参照: UniProt: A0T0S1 |
| #11: タンパク質 | 分子量: 20048.312 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Thalassiosira pseudonana CCMP1335 (珪藻) / 参照: UniProt: B8BUW3 Thalassiosira pseudonana CCMP1335 (珪藻) / 参照: UniProt: B8BUW3 |
-Fucoxanthin chlorophyll a/c-binding protein ... , 2種, 2分子 15
| #13: タンパク質 | 分子量: 23695.039 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Thalassiosira pseudonana CCMP1335 (珪藻) Thalassiosira pseudonana CCMP1335 (珪藻) |
|---|---|
| #17: タンパク質 | 分子量: 21112.090 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Thalassiosira pseudonana CCMP1335 (珪藻) / 参照: UniProt: B8C770 Thalassiosira pseudonana CCMP1335 (珪藻) / 参照: UniProt: B8C770 |
-Fucoxanthin chl a/c light-harvesting ... , 2種, 2分子 24
| #14: タンパク質 | 分子量: 21438.559 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Thalassiosira pseudonana CCMP1335 (珪藻) / 参照: UniProt: B8C2K6 Thalassiosira pseudonana CCMP1335 (珪藻) / 参照: UniProt: B8C2K6 |
|---|---|
| #16: タンパク質 | 分子量: 21502.492 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Thalassiosira pseudonana CCMP1335 (珪藻) / 参照: UniProt: B5YM25 Thalassiosira pseudonana CCMP1335 (珪藻) / 参照: UniProt: B5YM25 |
-糖 , 2種, 5分子 


| #24: 糖 | ChemComp-LMT / #26: 糖 | ChemComp-DGD / | |
|---|
-非ポリマー , 13種, 1213分子 







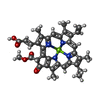
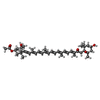











| #18: 化合物 | ChemComp-CL0 / | ||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| #19: 化合物 | ChemComp-CLA / #20: 化合物 | #21: 化合物 | #22: 化合物 | ChemComp-BCR / #23: 化合物 | ChemComp-LHG / #25: 化合物 | ChemComp-UNL / 分子数: 89 / 由来タイプ: 合成 #27: 化合物 | ChemComp-5X6 / | 分子量: 568.871 Da / 分子数: 1 / 由来タイプ: 合成 / 式: C40H56O2 #28: 化合物 | ChemComp-DD6 / ( #29: 化合物 | ChemComp-LMG / | #30: 化合物 | ChemComp-KC1 / #31: 化合物 | ChemComp-A86 / ( #32: 水 | ChemComp-HOH / | |
-詳細
| 研究の焦点であるリガンドがあるか | N |
|---|---|
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: PSI-FCPI / タイプ: COMPLEX / Entity ID: #1-#17 / 由来: NATURAL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 値: 0.55 MDa / 実験値: NO | |||||||||||||||
| 由来(天然) | 生物種:  Thalassiosira pseudonana CCMP1335 (珪藻) Thalassiosira pseudonana CCMP1335 (珪藻) | |||||||||||||||
| 緩衝液 | pH: 6.5 | |||||||||||||||
| 緩衝液成分 |
| |||||||||||||||
| 試料 | 濃度: 3 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES | |||||||||||||||
| 試料支持 | グリッドの材料: COPPER / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: Quantifoil R1.2/1.3 | |||||||||||||||
| 急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 277 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 顕微鏡 | モデル: JEOL CRYO ARM 300 |
|---|---|
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 60000 X / 最大 デフォーカス(公称値): 1800 nm / 最小 デフォーカス(公称値): 1200 nm |
| 撮影 | 電子線照射量: 50 e/Å2 / 検出モード: COUNTING / フィルム・検出器のモデル: GATAN K3 (6k x 4k) |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 2733572 | ||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性: C1 (非対称) | ||||||||||||||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 2.3 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 75667 / アルゴリズム: FOURIER SPACE / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: FLEXIBLE FIT / 空間: RECIPROCAL | ||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | 詳細: ModelAngelo / Source name: Other / タイプ: experimental model |
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj






















