[English] 日本語
 Yorodumi
Yorodumi- PDB-8ttw: Cryo-EM structure of BG505 SOSIP.664 HIV-1 Env trimer in complex ... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 8ttw | |||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Title | Cryo-EM structure of BG505 SOSIP.664 HIV-1 Env trimer in complex with temsavir, 8ANC195, and 10-1074 | |||||||||||||||||||||||||||||||||||||||
 Components Components |
| |||||||||||||||||||||||||||||||||||||||
 Keywords Keywords | VIRAL PROTEIN/INHIBITOR/IMMUNE SYSTEM / BG505 SOSIP / 8ANC195 / 10-1074 / BMS-626529 / temsavir / VIRAL PROTEIN-INHIBITOR-IMMUNE SYSTEM complex | |||||||||||||||||||||||||||||||||||||||
| Function / homology |  Function and homology information Function and homology informationsymbiont-mediated perturbation of host defense response / positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell ...symbiont-mediated perturbation of host defense response / positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / host cell plasma membrane / virion membrane / structural molecule activity / membrane / identical protein binding Similarity search - Function | |||||||||||||||||||||||||||||||||||||||
| Biological species |   Human immunodeficiency virus 1 Human immunodeficiency virus 1 Homo sapiens (human) Homo sapiens (human) | |||||||||||||||||||||||||||||||||||||||
| Method | ELECTRON MICROSCOPY / single particle reconstruction / cryo EM / Resolution: 2.96 Å | |||||||||||||||||||||||||||||||||||||||
 Authors Authors | Tolbert, W.D. / Pozharski, E. / Pazgier, M. | |||||||||||||||||||||||||||||||||||||||
| Funding support |  United States, 2items United States, 2items
| |||||||||||||||||||||||||||||||||||||||
 Citation Citation |  Journal: Nat Commun / Year: 2023 Journal: Nat Commun / Year: 2023Title: Structure-function analyses reveal key molecular determinants of HIV-1 CRF01_AE resistance to the entry inhibitor temsavir. Authors: Jérémie Prévost / Yaozong Chen / Fei Zhou / William D Tolbert / Romain Gasser / Halima Medjahed / Manon Nayrac / Dung N Nguyen / Suneetha Gottumukkala / Ann J Hessell / Venigalla B Rao / ...Authors: Jérémie Prévost / Yaozong Chen / Fei Zhou / William D Tolbert / Romain Gasser / Halima Medjahed / Manon Nayrac / Dung N Nguyen / Suneetha Gottumukkala / Ann J Hessell / Venigalla B Rao / Edwin Pozharski / Rick K Huang / Doreen Matthies / Andrés Finzi / Marzena Pazgier /   Abstract: The HIV-1 entry inhibitor temsavir prevents the viral receptor CD4 (cluster of differentiation 4) from interacting with the envelope glycoprotein (Env) and blocks its conformational changes. To do ...The HIV-1 entry inhibitor temsavir prevents the viral receptor CD4 (cluster of differentiation 4) from interacting with the envelope glycoprotein (Env) and blocks its conformational changes. To do this, temsavir relies on the presence of a residue with small side chain at position 375 in Env and is unable to neutralize viral strains like CRF01_AE carrying His375. Here we investigate the mechanism of temsavir resistance and show that residue 375 is not the sole determinant of resistance. At least six additional residues within the gp120 inner domain layers, including five distant from the drug-binding pocket, contribute to resistance. A detailed structure-function analysis using engineered viruses and soluble trimer variants reveals that the molecular basis of resistance is mediated by crosstalk between His375 and the inner domain layers. Furthermore, our data confirm that temsavir can adjust its binding mode to accommodate changes in Env conformation, a property that likely contributes to its broad antiviral activity. | |||||||||||||||||||||||||||||||||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  8ttw.cif.gz 8ttw.cif.gz | 787.9 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb8ttw.ent.gz pdb8ttw.ent.gz | 645.1 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  8ttw.json.gz 8ttw.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/tt/8ttw https://data.pdbj.org/pub/pdb/validation_reports/tt/8ttw ftp://data.pdbj.org/pub/pdb/validation_reports/tt/8ttw ftp://data.pdbj.org/pub/pdb/validation_reports/tt/8ttw | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  41613MC  8czzC  8dokC  8g6uC M: map data used to model this data C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
|
|---|---|
| 1 |
|
- Components
Components
-Protein , 2 types, 6 molecules AEIBFJ
| #1: Protein | Mass: 54064.277 Da / Num. of mol.: 3 / Mutation: A501C T332N E509R K510R A512R V513R Source method: isolated from a genetically manipulated source Source: (gene. exp.)   Human immunodeficiency virus 1 / Gene: env / Cell line (production host): HEK 293 GnT1- / Production host: Human immunodeficiency virus 1 / Gene: env / Cell line (production host): HEK 293 GnT1- / Production host:  Homo sapiens (human) / References: UniProt: Q2N0S6 Homo sapiens (human) / References: UniProt: Q2N0S6#2: Protein | Mass: 17146.482 Da / Num. of mol.: 3 / Mutation: I559P T605C Source method: isolated from a genetically manipulated source Source: (gene. exp.)   Human immunodeficiency virus 1 / Gene: env / Cell line (production host): HEK 293 GNT1- / Production host: Human immunodeficiency virus 1 / Gene: env / Cell line (production host): HEK 293 GNT1- / Production host:  Homo sapiens (human) / References: UniProt: Q2N0S5 Homo sapiens (human) / References: UniProt: Q2N0S5 |
|---|
-Antibody , 4 types, 12 molecules CGKDHLMOQNPR
| #3: Antibody | Mass: 25324.402 Da / Num. of mol.: 3 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Cell line (production host): HEK293 / Production host: Homo sapiens (human) / Cell line (production host): HEK293 / Production host:  Homo sapiens (human) Homo sapiens (human)#4: Antibody | Mass: 23460.047 Da / Num. of mol.: 3 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Cell line (production host): HEK293 / Production host: Homo sapiens (human) / Cell line (production host): HEK293 / Production host:  Homo sapiens (human) Homo sapiens (human)#5: Antibody | Mass: 25661.688 Da / Num. of mol.: 3 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Cell line (production host): HEK293 / Production host: Homo sapiens (human) / Cell line (production host): HEK293 / Production host:  Homo sapiens (human) Homo sapiens (human)#6: Antibody | Mass: 23180.760 Da / Num. of mol.: 3 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Cell line (production host): HEK293 / Production host: Homo sapiens (human) / Cell line (production host): HEK293 / Production host:  Homo sapiens (human) Homo sapiens (human) |
|---|
-Sugars , 5 types, 63 molecules 
| #7: Polysaccharide | 2-acetamido-2-deoxy-beta-D-glucopyranose-(1-4)-2-acetamido-2-deoxy-beta-D-glucopyranose Source method: isolated from a genetically manipulated source #8: Polysaccharide | Source method: isolated from a genetically manipulated source #9: Polysaccharide | Source method: isolated from a genetically manipulated source #10: Polysaccharide | Source method: isolated from a genetically manipulated source #11: Sugar | ChemComp-NAG / |
|---|
-Non-polymers , 1 types, 3 molecules 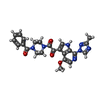
| #12: Chemical |
|---|
-Details
| Has ligand of interest | Y |
|---|---|
| Has protein modification | Y |
-Experimental details
-Experiment
| Experiment | Method: ELECTRON MICROSCOPY |
|---|---|
| EM experiment | Aggregation state: PARTICLE / 3D reconstruction method: single particle reconstruction |
- Sample preparation
Sample preparation
| Component | Name: Complex of BG505 SOSIP.664 with temsavir and the antibody Fabs of 8anc195 and 10-1074 Type: COMPLEX Details: Fab fragments were generated by papain digest of their respective IgGs. Entity ID: #1-#6 / Source: MULTIPLE SOURCES |
|---|---|
| Molecular weight | Experimental value: NO |
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Source (recombinant) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Buffer solution | pH: 7.2 |
| Specimen | Conc.: 4 mg/ml / Embedding applied: NO / Shadowing applied: NO / Staining applied: NO / Vitrification applied: YES |
| Vitrification | Instrument: FEI VITROBOT MARK IV / Cryogen name: ETHANE |
- Electron microscopy imaging
Electron microscopy imaging
| Microscopy | Model: TFS GLACIOS |
|---|---|
| Electron gun | Electron source:  FIELD EMISSION GUN / Accelerating voltage: 200 kV / Illumination mode: FLOOD BEAM FIELD EMISSION GUN / Accelerating voltage: 200 kV / Illumination mode: FLOOD BEAM |
| Electron lens | Mode: BRIGHT FIELD / Nominal defocus max: 2700 nm / Nominal defocus min: 500 nm / Cs: 2.7 mm |
| Specimen holder | Cryogen: NITROGEN |
| Image recording | Average exposure time: 2.5 sec. / Electron dose: 43.8 e/Å2 / Film or detector model: GATAN K3 (6k x 4k) |
- Processing
Processing
| EM software |
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF correction | Type: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||
| Symmetry | Point symmetry: C3 (3 fold cyclic) | ||||||||||||||||||||||||
| 3D reconstruction | Resolution: 2.96 Å / Resolution method: FSC 0.143 CUT-OFF / Num. of particles: 319575 / Symmetry type: POINT | ||||||||||||||||||||||||
| Refine LS restraints |
|
 Movie
Movie Controller
Controller





 PDBj
PDBj






