+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8pqh | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | PDGFRA T674I mutant kinase domain in complex with avapritinib | |||||||||||||||
 要素 要素 | Platelet-derived growth factor receptor alpha | |||||||||||||||
 キーワード キーワード | TRANSFERASE / PDGFRA protein kinase / avapritinib / inhibitor / tyrosine kinase / transmembrane receptor / platelet-derived growth factor receptor alpha / PDGF / platelet-derived growth factor / GIST / gastrointestinal stromal tumors / gatekeeper mutant / P16234 | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Imatinib-resistant PDGFR mutants / Sunitinib-resistant PDGFR mutants / Regorafenib-resistant PDGFR mutants / Sorafenib-resistant PDGFR mutants / PDGFR mutants bind TKIs / platelet-derived growth factor receptor-alpha signaling pathway / platelet-derived growth factor receptor-ligand complex / platelet-derived growth factor alpha-receptor activity / metanephric glomerular capillary formation / regulation of mesenchymal stem cell differentiation ...Imatinib-resistant PDGFR mutants / Sunitinib-resistant PDGFR mutants / Regorafenib-resistant PDGFR mutants / Sorafenib-resistant PDGFR mutants / PDGFR mutants bind TKIs / platelet-derived growth factor receptor-alpha signaling pathway / platelet-derived growth factor receptor-ligand complex / platelet-derived growth factor alpha-receptor activity / metanephric glomerular capillary formation / regulation of mesenchymal stem cell differentiation / luteinization / positive regulation of cell proliferation by VEGF-activated platelet derived growth factor receptor signaling pathway / platelet-derived growth factor binding / vascular endothelial growth factor binding / embryonic skeletal system morphogenesis / retina vasculature development in camera-type eye / vascular endothelial growth factor receptor activity / embryonic digestive tract morphogenesis / cardiac myofibril assembly / Leydig cell differentiation / cell activation / Signaling by PDGF / positive regulation of chemotaxis / male genitalia development / embryonic cranial skeleton morphogenesis / phospholipase C activator activity / platelet-derived growth factor receptor binding / estrogen metabolic process / face morphogenesis / signal transduction involved in regulation of gene expression / adrenal gland development / platelet-derived growth factor receptor signaling pathway / microvillus / odontogenesis of dentin-containing tooth / roof of mouth development / white fat cell differentiation / negative regulation of platelet activation / hematopoietic progenitor cell differentiation / Signaling by PDGFRA transmembrane, juxtamembrane and kinase domain mutants / Signaling by PDGFRA extracellular domain mutants / extracellular matrix organization / transmembrane receptor protein tyrosine kinase activity / Downstream signal transduction / positive regulation of calcium-mediated signaling / lung development / cell surface receptor protein tyrosine kinase signaling pathway / peptidyl-tyrosine phosphorylation / cell chemotaxis / cellular response to reactive oxygen species / regulation of actin cytoskeleton organization / cellular response to amino acid stimulus / wound healing / receptor protein-tyrosine kinase / platelet aggregation / positive regulation of fibroblast proliferation / Constitutive Signaling by Aberrant PI3K in Cancer / cell junction / cell migration / PIP3 activates AKT signaling / protein autophosphorylation / PI5P, PP2A and IER3 Regulate PI3K/AKT Signaling / RAF/MAP kinase cascade / in utero embryonic development / positive regulation of ERK1 and ERK2 cascade / protein kinase activity / receptor complex / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / nuclear body / cilium / positive regulation of cell migration / external side of plasma membrane / positive regulation of cell population proliferation / endoplasmic reticulum membrane / protein-containing complex binding / Golgi apparatus / protein homodimerization activity / protein-containing complex / nucleoplasm / ATP binding / nucleus / membrane / plasma membrane / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.5 Å 分子置換 / 解像度: 2.5 Å | |||||||||||||||
 データ登録者 データ登録者 | Teuber, A. / Kleinboelting, S. / Mueller, M.P. / Rauh, D. | |||||||||||||||
| 資金援助 | European Union,  ドイツ, 4件 ドイツ, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Avapritinib-based SAR studies unveil a binding pocket in KIT and PDGFRA. 著者: Teuber, A. / Schulz, T. / Fletcher, B.S. / Gontla, R. / Muhlenberg, T. / Zischinsky, M.L. / Niggenaber, J. / Weisner, J. / Kleinbolting, S.B. / Lategahn, J. / Sievers, S. / Muller, M.P. / Bauer, S. / Rauh, D. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8pqh.cif.gz 8pqh.cif.gz | 167.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8pqh.ent.gz pdb8pqh.ent.gz | 108.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8pqh.json.gz 8pqh.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  8pqh_validation.pdf.gz 8pqh_validation.pdf.gz | 708.2 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  8pqh_full_validation.pdf.gz 8pqh_full_validation.pdf.gz | 709.5 KB | 表示 | |
| XML形式データ |  8pqh_validation.xml.gz 8pqh_validation.xml.gz | 13.4 KB | 表示 | |
| CIF形式データ |  8pqh_validation.cif.gz 8pqh_validation.cif.gz | 17.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/pq/8pqh https://data.pdbj.org/pub/pdb/validation_reports/pq/8pqh ftp://data.pdbj.org/pub/pdb/validation_reports/pq/8pqh ftp://data.pdbj.org/pub/pdb/validation_reports/pq/8pqh | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8pq9C  8pqaC  8pqbC  8pqcC  8pqdC  8pqeC  8pqfC  8pqgC  8pqiC  8pqjC  8pqkC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
| 実験データセット #1 | データ参照:  10.18430/m38pqh / データの種類: diffraction image data / 詳細: https://www.proteindiffraction.org/project/8PQH/ 10.18430/m38pqh / データの種類: diffraction image data / 詳細: https://www.proteindiffraction.org/project/8PQH/ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 40226.371 Da / 分子数: 1 / 変異: T674I / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: PDGFRA, PDGFR2, RHEPDGFRA Homo sapiens (ヒト) / 遺伝子: PDGFRA, PDGFR2, RHEPDGFRA発現宿主:  参照: UniProt: P16234, receptor protein-tyrosine kinase |
|---|---|
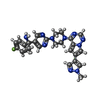
| #2: 化合物 | ChemComp-9JI / |
| #3: 化合物 | ChemComp-PEG / |
| #4: 水 | ChemComp-HOH / |
| 研究の焦点であるリガンドがあるか | Y |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.02 Å3/Da / 溶媒含有率: 39.24 % |
|---|---|
| 結晶化 | 温度: 293.15 K / 手法: 蒸気拡散法, ハンギングドロップ法 / 詳細: 10 mg/mL, 13% PEG3350, 200 mM KCl |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SLS SLS  / ビームライン: X10SA / 波長: 1 Å / ビームライン: X10SA / 波長: 1 Å |
| 検出器 | タイプ: DECTRIS EIGER2 X 16M / 検出器: PIXEL / 日付: 2021年2月17日 |
| 放射 | モノクロメーター: Si(111) / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1 Å / 相対比: 1 |
| 反射 | 解像度: 2.5→46.75 Å / Num. obs: 14185 / % possible obs: 100 % / 冗長度: 13.09 % / Biso Wilson estimate: 71.45 Å2 / CC1/2: 0.999 / Rrim(I) all: 0.08 / Net I/σ(I): 18.79 |
| 反射 シェル | 解像度: 2.5→2.6 Å / 冗長度: 13.67 % / Mean I/σ(I) obs: 2.06 / Num. unique obs: 1529 / CC1/2: 0.77 / Rrim(I) all: 1.499 / % possible all: 100 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 / 解像度: 2.5→46.57 Å / SU ML: 0.399 / 交差検証法: FREE R-VALUE / σ(F): 1.36 / 位相誤差: 30.9463 分子置換 / 解像度: 2.5→46.57 Å / SU ML: 0.399 / 交差検証法: FREE R-VALUE / σ(F): 1.36 / 位相誤差: 30.9463 立体化学のターゲット値: GeoStd + Monomer Library + CDL v1.2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 78.98 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.5→46.57 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ | Refine-ID: X-RAY DIFFRACTION / Auth asym-ID: A / Label asym-ID: A
|
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj