+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8hin | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Structure of human SGLT2-MAP17 complex with Phlorizin | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | TRANSPORT PROTEIN / Ion transport / Sodium transport / Sugar transport / Symport / Transport | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報low-affinity D-glucose:sodium symporter activity / Defective SLC5A2 causes renal glucosuria (GLYS1) / alpha-glucoside transport / alpha-glucoside transmembrane transporter activity / D-glucose:sodium symporter activity / renal D-glucose absorption / hexose transmembrane transport / D-glucose import across plasma membrane / Cellular hexose transport / D-glucose transmembrane transporter activity ...low-affinity D-glucose:sodium symporter activity / Defective SLC5A2 causes renal glucosuria (GLYS1) / alpha-glucoside transport / alpha-glucoside transmembrane transporter activity / D-glucose:sodium symporter activity / renal D-glucose absorption / hexose transmembrane transport / D-glucose import across plasma membrane / Cellular hexose transport / D-glucose transmembrane transporter activity / sodium ion import across plasma membrane / sodium ion transport / carbohydrate metabolic process / apical plasma membrane / extracellular exosome / metal ion binding / membrane / plasma membrane 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | ||||||
 データ登録者 データ登録者 | Hiraizumi, M. / Kishida, H. / Miyaguchi, I. / Nureki, O. | ||||||
| 資金援助 | 1件
| ||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2024 ジャーナル: Nat Struct Mol Biol / 年: 2024タイトル: Transport and inhibition mechanism of the human SGLT2-MAP17 glucose transporter. 著者: Masahiro Hiraizumi / Tomoya Akashi / Kouta Murasaki / Hiroyuki Kishida / Taichi Kumanomidou / Nao Torimoto / Osamu Nureki / Ikuko Miyaguchi /  要旨: Sodium-glucose cotransporter 2 (SGLT2) is imporant in glucose reabsorption. SGLT2 inhibitors suppress renal glucose reabsorption, therefore reducing blood glucose levels in patients with type 2 ...Sodium-glucose cotransporter 2 (SGLT2) is imporant in glucose reabsorption. SGLT2 inhibitors suppress renal glucose reabsorption, therefore reducing blood glucose levels in patients with type 2 diabetes. We and others have developed several SGLT2 inhibitors starting from phlorizin, a natural product. Using cryo-electron microscopy, we present the structures of human (h)SGLT2-MAP17 complexed with five natural or synthetic inhibitors. The four synthetic inhibitors (including canagliflozin) bind the transporter in the outward conformations, while phlorizin binds it in the inward conformation. The phlorizin-hSGLT2 interaction exhibits biphasic kinetics, suggesting that phlorizin alternately binds to the extracellular and intracellular sides. The Na-bound outward-facing and unbound inward-open structures of hSGLT2-MAP17 suggest that the MAP17-associated bundle domain functions as a scaffold, with the hash domain rotating around the Na-binding site. Thus, Na binding stabilizes the outward-facing conformation, and its release promotes state transition to inward-open conformation, exhibiting a role of Na in symport mechanism. These results provide structural evidence for the Na-coupled alternating-access mechanism proposed for the transporter family. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8hin.cif.gz 8hin.cif.gz | 138.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8hin.ent.gz pdb8hin.ent.gz | 102.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8hin.json.gz 8hin.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  8hin_validation.pdf.gz 8hin_validation.pdf.gz | 1.1 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  8hin_full_validation.pdf.gz 8hin_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  8hin_validation.xml.gz 8hin_validation.xml.gz | 29.7 KB | 表示 | |
| CIF形式データ |  8hin_validation.cif.gz 8hin_validation.cif.gz | 42.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/hi/8hin https://data.pdbj.org/pub/pdb/validation_reports/hi/8hin ftp://data.pdbj.org/pub/pdb/validation_reports/hi/8hin ftp://data.pdbj.org/pub/pdb/validation_reports/hi/8hin | HTTPS FTP |
-関連構造データ
| 関連構造データ |  34823MC  8hb0C  8hdhC  8hezC  8hg7C M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 73247.703 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: SLC5A2, SGLT2 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: SLC5A2, SGLT2 / 発現宿主:  Homo sapiens (ヒト) / 参照: UniProt: P31639 Homo sapiens (ヒト) / 参照: UniProt: P31639 |
|---|---|
| #2: タンパク質 | 分子量: 12235.000 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: MAP17 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: MAP17 / 発現宿主:  Homo sapiens (ヒト) / 参照: UniProt: Q13113 Homo sapiens (ヒト) / 参照: UniProt: Q13113 |
| #3: 糖 | ChemComp-NAG / |
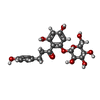
| #4: 化合物 | ChemComp-LN9 / |
| 研究の焦点であるリガンドがあるか | Y |
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Binary complex of Sodium/glucose cotransporter 2 with PDZK1-interacting protein 1. タイプ: COMPLEX 詳細: Electrogenic Na+-coupled sugar simporter that actively transports D-glucose at the plasma membrane, with a Na+ to sugar coupling ratio of 1:1. Transporter activity is driven by a ...詳細: Electrogenic Na+-coupled sugar simporter that actively transports D-glucose at the plasma membrane, with a Na+ to sugar coupling ratio of 1:1. Transporter activity is driven by a transmembrane Na+ electrochemical gradient set by the Na+/K+ pump Entity ID: #1-#2 / 由来: RECOMBINANT |
|---|---|
| 分子量 | 実験値: NO |
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 由来(組換発現) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 緩衝液 | pH: 7.5 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: OTHER FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: OTHER |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1600 nm / 最小 デフォーカス(公称値): 800 nm |
| 撮影 | 電子線照射量: 64 e/Å2 フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) |
- 解析
解析
| ソフトウェア | 名称: REFMAC / バージョン: 5.8.0267 / 分類: 精密化 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3.3 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 76485 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | 解像度: 3.3→3.3 Å / Cor.coef. Fo:Fc: 0.879 / SU B: 11.542 / SU ML: 0.193 / ESU R: 0.323 立体化学のターゲット値: MAXIMUM LIKELIHOOD WITH PHASES 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: PARAMETERS FOR MASK CACLULATION | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 115.573 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: 1 / 合計: 4788 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー







 PDBj
PDBj