| Entry | Database: PDB / ID: 8gtw
|
|---|
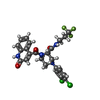
| Title | SARS-CoV-2 3CL protease (3CLpro) in complex with compound JZD-26 |
|---|
 Components Components | 3C-like proteinase |
|---|
 Keywords Keywords | VIRAL PROTEIN/INHIBITOR / Mpro / VIRAL PROTEIN-INHIBITOR COMPLEX |
|---|
| Function / homology |  Function and homology information Function and homology information
viral genome replication / methyltransferase activity / regulation of autophagy / Assembly of the SARS-CoV-2 Replication-Transcription Complex (RTC) / Maturation of replicase proteins / ISG15-specific peptidase activity / Transcription of SARS-CoV-2 sgRNAs / endonuclease activity / Translation of Replicase and Assembly of the Replication Transcription Complex / Replication of the SARS-CoV-2 genome ...viral genome replication / methyltransferase activity / regulation of autophagy / Assembly of the SARS-CoV-2 Replication-Transcription Complex (RTC) / Maturation of replicase proteins / ISG15-specific peptidase activity / Transcription of SARS-CoV-2 sgRNAs / endonuclease activity / Translation of Replicase and Assembly of the Replication Transcription Complex / Replication of the SARS-CoV-2 genome / double membrane vesicle viral factory outer membrane / SARS coronavirus main proteinase / methylation / host cell endosome / symbiont-mediated degradation of host mRNA / mRNA guanylyltransferase / symbiont-mediated suppression of host ISG15-protein conjugation / G-quadruplex RNA binding / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of IRF3 activity / omega peptidase activity / SARS-CoV-2 modulates host translation machinery / host cell Golgi apparatus / symbiont-mediated perturbation of host ubiquitin-like protein modification / ubiquitinyl hydrolase 1 / cysteine-type deubiquitinase activity / Hydrolases; Acting on peptide bonds (peptidases); Cysteine endopeptidases / single-stranded RNA binding / viral protein processing / host cell perinuclear region of cytoplasm / host cell endoplasmic reticulum membrane / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / symbiont-mediated suppression of host gene expression / symbiont-mediated activation of host autophagy / viral translational frameshifting / cysteine-type endopeptidase activity / lipid binding / host cell nucleus / SARS-CoV-2 activates/modulates innate and adaptive immune responses / proteolysis / zinc ion binding / membraneSimilarity search - Function Coronavirus replicase NSP2, C-terminal / main proteinase (3clpro) structure, domain 3 / main proteinase (3clpro) structure, domain 3 / Trypsin-like serine proteases / Non-structural protein NSP3, SUD-N (Mac2) domain, betacoronavirus / Sarbecovirus Nsp3c-N domain profile. / Non-structural protein NSP3, N-terminal, betacoronavirus / Polyprotein cleavage domain PL2pro superfamily, betacoronavirus / Non-structural protein NSP3, SUD-N (Mac2) domain superfamily, betacoronavirus / Betacoronavirus SUD-C domain ...Coronavirus replicase NSP2, C-terminal / main proteinase (3clpro) structure, domain 3 / main proteinase (3clpro) structure, domain 3 / Trypsin-like serine proteases / Non-structural protein NSP3, SUD-N (Mac2) domain, betacoronavirus / Sarbecovirus Nsp3c-N domain profile. / Non-structural protein NSP3, N-terminal, betacoronavirus / Polyprotein cleavage domain PL2pro superfamily, betacoronavirus / Non-structural protein NSP3, SUD-N (Mac2) domain superfamily, betacoronavirus / Betacoronavirus SUD-C domain / Betacoronavirus replicase NSP3, N-terminal / NSP1 globular domain superfamily, betacoronavirus / Non-structural protein 2, SARS-CoV-like / Thrombin, subunit H / Betacoronavirus single-stranded poly(A) binding domain / Betacoronavirus Nsp3c-M domain profile. / NSP1, globular domain, betacoronavirus / Non-structural protein NSP3, SUD-M domain, betacoronavirus / Non-structural protein NSP3, SUD-M domain superfamily, betacoronavirus / Betacoronavirus replicase NSP1 / NSP1, C-terminal domain, betacoronavirus / Betacoronavirus (BetaCoV) Nsp1 C-terminal domain profile. / : / Betacoronavirus Nsp3e group 2-specific marker (G2M) domain profile. / Betacoronavirus Nsp3c-C domain profile. / Betacoronavirus Nsp3e nucleic acid-binding (NAB) domain profile. / DPUP/SUD, C-terminal, betacoronavirus / Non-structural protein NSP3, nucleic acid-binding domain, betacoronavirus / Non-structural protein NSP3A domain-like superfamily / Non-structural protein NSP3, nucleic acid-binding domain superfamily, betacoronavirus / Non-structural protein 6, betacoronavirus / Betacoronavirus nucleic acid-binding (NAB) / Papain-like viral protease, palm and finger domains, coronavirus / Papain-like protease, N-terminal domain superfamily, coronavirus / Carbamoyl-phosphate synthase subdomain signature 2. / Coronavirus replicase NSP2, N-terminal / : / Coronavirus (CoV) Nsp2 middle domain profile. / NSP1, globular domain, alpha/betacoronavirus / Coronavirus (CoV) Nsp1 globular domain profile. / Coronavirus (CoV) Nsp2 N-terminal domain profile. / Coronavirus (CoV) Nsp2 C-terminal domain profile. / Nonstructural protein 2, N-terminal domain, coronavirus / Non-structural protein 2, C-terminal domain, coronavirus / NSP3, second ubiquitin-like (Ubl) domain, coronavirus / Coronavirus Nsp3a Ubl domain profile. / Coronavirus Nsp3d Ubl domain profile. / NSP3, first ubiquitin-like (Ubl) domain, coronavirus / Peptidase family C16 domain profile. / Coronavirus replicase NSP7 / : / : / Coronavirus Nsp3 ectodomain (3Ecto) profile. / Coronavirus RNA-dependent RNA polymerase (RdRp) Nsp7 cofactor domain profile. / Coronavirus RNA-dependent RNA polymerase (RdRp) Nsp8 cofactor domain profile. / Coronavirus Nsp9 single-stranded RNA (ssRNA)-binding domain profile. / Coronavirus (CoV) ExoN/MTase coactivator domain profile. / Coronavirus (CoV) Nsp3 Y domain profile. / Coronavirus Nsp4 C-terminal (Nsp4C) domain profile. / Coronavirus main protease (M-pro) domain profile. / Peptidase C30, coronavirus / Peptidase C16, coronavirus / Non-structural protein NSP9, coronavirus / Non-structural protein NSP7, coronavirus / Non-structural protein NSP8, coronavirus / RNA synthesis protein NSP10, coronavirus / Non-structural protein NSP4, C-terminal, coronavirus / RNA synthesis protein NSP10 superfamily, coronavirus / Non-structural protein NSP9 superfamily, coronavirus / Non-structural protein NSP7 superfamily, coronavirus / Non-structural protein NSP8 superfamily, coronavirus / Non-structural protein NSP4, C-terminal superfamily, coronavirus / Papain-like protease, thumb domain superfamily, coronavirus / Peptidase C30, domain 3, coronavirus / Non-structural protein 6, coronavirus / Coronavirus replicase NSP3, C-terminal / Non-structural protein NSP4, N-terminal, coronavirus / Coronavirus endopeptidase C30 / Coronavirus papain-like peptidase / Coronavirus replicase NSP8 / Coronavirus RNA synthesis protein NSP10 / Coronavirus replicase NSP4, C-terminal / Coronavirus replicase NSP6 / Coronavirus replicase NSP4, N-terminal / Coronavirus replicase NSP3, C-terminal / Coronavirus replicase NSP9 / Non-structural protein 3, X-domain-like / Appr-1"-p processing enzyme / Macro domain / Macro domain profile. / Macro domain / Macro domain-like / Peptidase S1, PA clan, chymotrypsin-like fold / Peptidase S1, PA clan / Beta Barrel / Orthogonal Bundle / Mainly Beta / Mainly AlphaSimilarity search - Domain/homology |
|---|
| Biological species |   Severe acute respiratory syndrome coronavirus 2 Severe acute respiratory syndrome coronavirus 2 |
|---|
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / MOLECULAR REPLACEMENT /  molecular replacement / Resolution: 1.85 Å molecular replacement / Resolution: 1.85 Å |
|---|
 Authors Authors | Su, H.X. / Nie, T.Q. / Xie, H. / Li, M.J. / Xu, Y.C. |
|---|
| Funding support | 1items | Organization | Grant number | Country |
|---|
| Not funded | | |
|
|---|
 Citation Citation |  Journal: J.Med.Chem. / Year: 2024 Journal: J.Med.Chem. / Year: 2024
Title: Discovery of Novel Nonpeptidic and Noncovalent Small Molecule 3CL pro Inhibitors as anti-SARS-CoV-2 Drug Candidate.
Authors: Jiang, Z. / Feng, B. / Chen, L. / Nie, T. / Chen, S. / Wang, L. / Liu, H. / Yu, T. / Zhang, Y. / Zheng, M. / Xu, Y. / Liu, H. / Zang, Y. / Su, H. / Zhang, L. / Li, J. / Zhou, Y. |
|---|
| History | | Deposition | Sep 8, 2022 | Deposition site: PDBJ / Processing site: PDBJ |
|---|
| Revision 1.0 | Sep 13, 2023 | Provider: repository / Type: Initial release |
|---|
| Revision 1.1 | Dec 24, 2025 | Group: Database references / Structure summary / Category: citation / citation_author / pdbx_entry_details
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_ASTM / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year / _pdbx_entry_details.has_protein_modification |
|---|
|
|---|
 Open data
Open data Basic information
Basic information Components
Components Keywords
Keywords Function and homology information
Function and homology information
 X-RAY DIFFRACTION /
X-RAY DIFFRACTION /  SYNCHROTRON /
SYNCHROTRON /  MOLECULAR REPLACEMENT /
MOLECULAR REPLACEMENT /  molecular replacement / Resolution: 1.85 Å
molecular replacement / Resolution: 1.85 Å  Authors
Authors Citation
Citation Journal: J.Med.Chem. / Year: 2024
Journal: J.Med.Chem. / Year: 2024 Structure visualization
Structure visualization Molmil
Molmil Jmol/JSmol
Jmol/JSmol Downloads & links
Downloads & links Download
Download 8gtw.cif.gz
8gtw.cif.gz PDBx/mmCIF format
PDBx/mmCIF format pdb8gtw.ent.gz
pdb8gtw.ent.gz PDB format
PDB format 8gtw.json.gz
8gtw.json.gz PDBx/mmJSON format
PDBx/mmJSON format Other downloads
Other downloads https://data.pdbj.org/pub/pdb/validation_reports/gt/8gtw
https://data.pdbj.org/pub/pdb/validation_reports/gt/8gtw ftp://data.pdbj.org/pub/pdb/validation_reports/gt/8gtw
ftp://data.pdbj.org/pub/pdb/validation_reports/gt/8gtw



 F&H Search
F&H Search Links
Links Assembly
Assembly

 Components
Components

 X-RAY DIFFRACTION / Number of used crystals: 1
X-RAY DIFFRACTION / Number of used crystals: 1  Sample preparation
Sample preparation SYNCHROTRON / Site:
SYNCHROTRON / Site:  SSRF
SSRF  / Beamline: BL19U1 / Wavelength: 0.9785 Å
/ Beamline: BL19U1 / Wavelength: 0.9785 Å molecular replacement
molecular replacement Processing
Processing MOLECULAR REPLACEMENT
MOLECULAR REPLACEMENT Movie
Movie Controller
Controller



 PDBj
PDBj