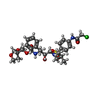
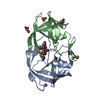
登録情報 データベース : PDB / ID : 8f0fタイトル HIV-1 wild type protease with GRL-110-19A, a chloroacetamide derivative based on Darunavir as P2' group Protease キーワード / / / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / 生物種 手法 / / / 解像度 : 1.29 Å データ登録者 Wang, Y.-F. / Agniswamy, J. / Ghosh, A.K. / Weber, I.T. 資金援助 組織 認可番号 国 National Institutes of Health/National Institute Of Allergy and Infectious Diseases (NIH/NIAID) AI150466 National Institutes of Health/National Institute Of Allergy and Infectious Diseases (NIH/NIAID) AI150461 Department of Energy (DOE, United States) W-31-109-Eng-38 National Institutes of Health/National Cancer Institute (NIH/NCI) Intramural Research Program of the Center for Cancer Research Japan Agency for Medical Research and Development (AMED) JP15fk0410001 Japan Agency for Medical Research and Development (AMED) JP16fk0410101 Ministry of Education, Culture, Sports, Science and Technology (Japan) a Grant-in-Aid for Scientific Research (Priority Areas)
ジャーナル : Bioorg.Med.Chem.Lett. / 年 : 2023タイトル : Evaluation of darunavir-derived HIV-1 protease inhibitors incorporating P2' amide-derivatives: Synthesis, biological evaluation and structural studies.著者 : Ghosh, A.K. / Shahabi, D. / Kipfmiller, M. / Ghosh, A.K. / Johnson, M. / Wang, Y.F. / Agniswamy, J. / Amano, M. / Weber, I.T. / Mitsuya, H. 履歴 登録 2022年11月2日 登録サイト / 処理サイト 改定 1.0 2023年2月15日 Provider / タイプ 改定 1.1 2023年3月8日 Group / カテゴリ / Item / _citation.title改定 1.2 2023年10月25日 Group / Refinement descriptionカテゴリ / chem_comp_bond / pdbx_initial_refinement_model
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)
Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.29 Å
分子置換 / 解像度: 1.29 Å  データ登録者
データ登録者 米国,
米国,  日本, 7件
日本, 7件  引用
引用 ジャーナル: Bioorg.Med.Chem.Lett. / 年: 2023
ジャーナル: Bioorg.Med.Chem.Lett. / 年: 2023 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 8f0f.cif.gz
8f0f.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb8f0f.ent.gz
pdb8f0f.ent.gz PDB形式
PDB形式 8f0f.json.gz
8f0f.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 8f0f_validation.pdf.gz
8f0f_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 8f0f_full_validation.pdf.gz
8f0f_full_validation.pdf.gz 8f0f_validation.xml.gz
8f0f_validation.xml.gz 8f0f_validation.cif.gz
8f0f_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/f0/8f0f
https://data.pdbj.org/pub/pdb/validation_reports/f0/8f0f ftp://data.pdbj.org/pub/pdb/validation_reports/f0/8f0f
ftp://data.pdbj.org/pub/pdb/validation_reports/f0/8f0f
 F&H 検索
F&H 検索 リンク
リンク 集合体
集合体
 要素
要素
 Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)
Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)








 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 22-BM / 波長: 1 Å
/ ビームライン: 22-BM / 波長: 1 Å 分子置換
分子置換 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー




 PDBj
PDBj




