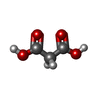
登録構造単位
D: Disks large-like protein 1
A: Disks large-like protein 1
B: Disks large-like protein 1
C: Disks large-like protein 1
E: Vang-like protein 1
F: Vang-like protein 1
G: Vang-like protein 1
H: Vang-like protein 1
ヘテロ分子 概要 構成要素の詳細
分子量 (理論値) 分子数 合計 (水以外) 43,452 11 ポリマ- 43,140 8 非ポリマー 312 3 水 2,432 135
1
D: Disks large-like protein 1
E: Vang-like protein 1 概要 構成要素の詳細 対称操作
分子量 (理論値) 分子数 合計 (水以外) 10,785 2 ポリマ- 10,785 2 非ポリマー 0 0 水 36 2
タイプ 名称 対称操作 数 identity operation 1_555 x,y,z 1
2
A: Disks large-like protein 1
F: Vang-like protein 1 概要 構成要素の詳細 対称操作
分子量 (理論値) 分子数 合計 (水以外) 10,785 2 ポリマ- 10,785 2 非ポリマー 0 0 水 36 2
タイプ 名称 対称操作 数 identity operation 1_555 x,y,z 1
3
B: Disks large-like protein 1
G: Vang-like protein 1
ヘテロ分子 概要 構成要素の詳細 対称操作
分子量 (理論値) 分子数 合計 (水以外) 10,993 4 ポリマ- 10,785 2 非ポリマー 208 2 水 36 2
タイプ 名称 対称操作 数 identity operation 1_555 x,y,z 1
4
C: Disks large-like protein 1
H: Vang-like protein 1
ヘテロ分子 概要 構成要素の詳細 対称操作
分子量 (理論値) 分子数 合計 (水以外) 10,889 3 ポリマ- 10,785 2 非ポリマー 104 1 水 36 2
タイプ 名称 対称操作 数 identity operation 1_555 x,y,z 1
単位格子 Length a, b, c (Å) 34.696, 47.953, 50.830 Angle α, β, γ (deg.) 85.250, 72.080, 69.170 Int Tables number 1 Space group name H-M P1 Space group name Hall P1 Symmetry operation #1 : x,y,z
非結晶学的対称性 (NCS) NCSドメイン ID Ens-ID 詳細 d_1ens_1(chain "A" and (resid 1 through 10 or (resid 11...d_2ens_1(chain "B" and (resid 1 through 10 or (resid 11...d_3ens_1(chain "C" and (resid 1 through 23 or (resid 24...d_4ens_1(chain "D" and (resid 1 through 10 or (resid 11...d_1ens_2(chain "E" and (resid 116 through 122 or (resid 123...d_2ens_2chain "F"d_3ens_2(chain "G" and (resid 48 through 54 or (resid 55...
NCSドメイン領域 Dom-ID Component-ID Ens-ID Beg label comp-ID End label comp-ID Label asym-ID Label seq-ID d_11 ens_1GLNSERB2 - 79 d_12 ens_1GLYARGB81 - 90 d_21 ens_1GLNSERC2 - 79 d_22 ens_1GLYARGC81 - 90 d_31 ens_1GLNSERF1 - 78 d_32 ens_1GLYARGF80 - 89 d_41 ens_1GLNARGA2 - 89 d_11 ens_2ASNVALH1 - 8 d_21 ens_2ASNVALI1 - 8 d_31 ens_2ASNVALJ1 - 8
NCSアンサンブル NCS oper ID Code Matrix ベクター 1 given(-0.92674991047, 0.214976796485, 0.308090214737), (-0.0347581556908, 0.767506107294, -0.640098621994), (-0.374067472599, -0.603919988276, -0.703814019258)11.9104237503, -9.76434247486, 14.98388166022 given(-0.927876811756, 0.229921027989, 0.293565909284), (0.0102413100797, -0.771268345651, 0.636427729255), (0.372746210944, 0.593533031841, 0.713287321029)29.3612730665, -8.07453369715, 4.303159878033 given(0.999867876545, 0.0161926596162, 0.00142380745098), (0.0161505445344, -0.999536485976, 0.025806454871), (0.00184102263588, -0.0257800499674, -0.999665944033)17.5108845343, -18.0106583031, 19.05272932374 given(0.999829474351, 0.00034320317063, -0.0184635974629), (0.000675087911356, -0.999838265231, 0.0179718567562), (-0.018454443259, -0.0179812566451, -0.999667998854)-16.6554112967, -18.3765611596, 20.22610152665 given(-0.730186026261, -0.330123065579, -0.598203250264), (0.158646416697, -0.933518099265, 0.321520252573), (-0.664574812585, 0.139866793478, 0.734014712768)33.089278161, -30.0029115125, 27.459253049
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.2 Å
分子置換 / 解像度: 2.2 Å  データ登録者
データ登録者 オーストラリア, 2件
オーストラリア, 2件  引用
引用 ジャーナル: To be published
ジャーナル: To be published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 8c1t.cif.gz
8c1t.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb8c1t.ent.gz
pdb8c1t.ent.gz PDB形式
PDB形式 8c1t.json.gz
8c1t.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 8c1t_validation.pdf.gz
8c1t_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 8c1t_full_validation.pdf.gz
8c1t_full_validation.pdf.gz 8c1t_validation.xml.gz
8c1t_validation.xml.gz 8c1t_validation.cif.gz
8c1t_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/c1/8c1t
https://data.pdbj.org/pub/pdb/validation_reports/c1/8c1t ftp://data.pdbj.org/pub/pdb/validation_reports/c1/8c1t
ftp://data.pdbj.org/pub/pdb/validation_reports/c1/8c1t
 F&H 検索
F&H 検索 リンク
リンク 集合体
集合体




 要素
要素


 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  Australian Synchrotron
Australian Synchrotron  / ビームライン: MX2 / 波長: 0.95372 Å
/ ビームライン: MX2 / 波長: 0.95372 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj