+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8ae7 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The strucuture of Compound 15 bound to CK2alpha | |||||||||
 要素 要素 | Casein kinase II subunit alpha | |||||||||
 キーワード キーワード | TRANSFERASE / Fragment based drug discovery | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of chromosome separation / positive regulation of aggrephagy / WNT mediated activation of DVL / Condensation of Prometaphase Chromosomes / protein kinase CK2 complex / symbiont-mediated disruption of host cell PML body / Receptor Mediated Mitophagy / Synthesis of PC / Sin3-type complex / Maturation of hRSV A proteins ...regulation of chromosome separation / positive regulation of aggrephagy / WNT mediated activation of DVL / Condensation of Prometaphase Chromosomes / protein kinase CK2 complex / symbiont-mediated disruption of host cell PML body / Receptor Mediated Mitophagy / Synthesis of PC / Sin3-type complex / Maturation of hRSV A proteins / RUNX1 interacts with co-factors whose precise effect on RUNX1 targets is not known / negative regulation of signal transduction by p53 class mediator / negative regulation of apoptotic signaling pathway / positive regulation of Wnt signaling pathway / negative regulation of double-strand break repair via homologous recombination / : / negative regulation of proteasomal ubiquitin-dependent protein catabolic process / Signal transduction by L1 / Hsp90 protein binding / PML body / Regulation of PTEN stability and activity / Wnt signaling pathway / positive regulation of protein catabolic process / kinase activity / KEAP1-NFE2L2 pathway / rhythmic process / Cooperation of PDCL (PhLP1) and TRiC/CCT in G-protein beta folding / double-strand break repair / positive regulation of cell growth / Regulation of TP53 Activity through Phosphorylation / non-specific serine/threonine protein kinase / regulation of cell cycle / negative regulation of translation / protein stabilization / protein serine kinase activity / protein serine/threonine kinase activity / positive regulation of cell population proliferation / apoptotic process / DNA damage response / signal transduction / nucleoplasm / ATP binding / identical protein binding / nucleus / plasma membrane / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.28 Å 分子置換 / 解像度: 1.28 Å | |||||||||
 データ登録者 データ登録者 | Brear, P. / De Fusco, C. / Atkinson, E. / Frances, N. / Iegre, J. / Venkitaraman, A. / Hyvonen, M. / Spring, D. | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Rsc Med Chem / 年: 2022 ジャーナル: Rsc Med Chem / 年: 2022タイトル: A fragment-based approach leading to the discovery of inhibitors of CK2 alpha with a novel mechanism of action. 著者: Brear, P. / De Fusco, C. / Atkinson, E.L. / Iegre, J. / Francis-Newton, N.J. / Venkitaraman, A.R. / Hyvonen, M. / Spring, D.R. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8ae7.cif.gz 8ae7.cif.gz | 95.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8ae7.ent.gz pdb8ae7.ent.gz | 69.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8ae7.json.gz 8ae7.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ae/8ae7 https://data.pdbj.org/pub/pdb/validation_reports/ae/8ae7 ftp://data.pdbj.org/pub/pdb/validation_reports/ae/8ae7 ftp://data.pdbj.org/pub/pdb/validation_reports/ae/8ae7 | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  7zy0C  7zy2C  7zy5C  7zy8C  7zydC  7zykC  7zyoC  7zyrC  8aecC  8aekC  8aemC  3pe1S S: 精密化の開始モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 1分子 A
| #1: タンパク質 | 分子量: 39031.391 Da / 分子数: 1 / 変異: K74A, K75A, K76A, R21S / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: CSNK2A1, CK2A1 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: CSNK2A1, CK2A1 / 発現宿主:  参照: UniProt: P68400, non-specific serine/threonine protein kinase |
|---|
-非ポリマー , 6種, 344分子 

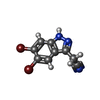








| #2: 化合物 | ChemComp-GOL / |
|---|---|
| #3: 化合物 | ChemComp-ACT / |
| #4: 化合物 | ChemComp-LVU / |
| #5: 化合物 | ChemComp-ADP / |
| #6: 化合物 | ChemComp-MG / |
| #7: 水 | ChemComp-HOH / |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.03 Å3/Da / 溶媒含有率: 39.49 % |
|---|---|
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, シッティングドロップ法 詳細: 112.5mM Mes pH 6.5, 35% glycerol ethoxylate, 180 mM ammonium acetate |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  Diamond Diamond  / ビームライン: I04 / 波長: 0.9184 Å / ビームライン: I04 / 波長: 0.9184 Å | ||||||||||||||||||||||||||||||
| 検出器 | タイプ: DECTRIS PILATUS 6M-F / 検出器: PIXEL / 日付: 2016年9月16日 | ||||||||||||||||||||||||||||||
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | ||||||||||||||||||||||||||||||
| 放射波長 | 波長: 0.9184 Å / 相対比: 1 | ||||||||||||||||||||||||||||||
| 反射 | 解像度: 1.28→59.02 Å / Num. obs: 80979 / % possible obs: 100 % / 冗長度: 6.4 % / Biso Wilson estimate: 12.98 Å2 / CC1/2: 0.999 / Rmerge(I) obs: 0.074 / Rpim(I) all: 0.032 / Rrim(I) all: 0.081 / Net I/σ(I): 10.3 / Num. measured all: 517476 / Scaling rejects: 628 | ||||||||||||||||||||||||||||||
| 反射 シェル | Diffraction-ID: 1
|
-位相決定
| 位相決定 | 手法:  分子置換 分子置換 |
|---|
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 3PE1 解像度: 1.28→54.47 Å / Cor.coef. Fo:Fc: 0.9601 / Cor.coef. Fo:Fc free: 0.9493 / SU R Cruickshank DPI: 0.057 / 交差検証法: THROUGHOUT / σ(F): 0 / SU R Blow DPI: 0.058 / SU Rfree Blow DPI: 0.06 / SU Rfree Cruickshank DPI: 0.059
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 107.38 Å2 / Biso mean: 20.2 Å2 / Biso min: 5.18 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.196 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: final / 解像度: 1.28→54.47 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 1.28→1.31 Å / Rfactor Rfree error: 0 / Total num. of bins used: 20
|
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj









