| 登録情報 | データベース: PDB / ID: 7x2e
|
|---|
| タイトル | Structure of USH1C PDZ2 and coiled-coil in complex with CDHR2 C-terminal tail |
|---|
 要素 要素 | - Cadherin-related family member 2
- Harmonin
|
|---|
 キーワード キーワード | PROTEIN BINDING / CELL ADHESION / PEPTIDE BINDING PROTEIN |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
negative regulation of cell growth involved in contact inhibition / intermicrovillar adhesion / protein localization to microvillus / brush border assembly / stereocilia ankle link complex / regulation of microvillus length / parallel actin filament bundle assembly / equilibrioception / sensory perception of light stimulus / retinal cone cell development ...negative regulation of cell growth involved in contact inhibition / intermicrovillar adhesion / protein localization to microvillus / brush border assembly / stereocilia ankle link complex / regulation of microvillus length / parallel actin filament bundle assembly / equilibrioception / sensory perception of light stimulus / retinal cone cell development / stereocilium tip / auditory receptor cell morphogenesis / inner ear receptor cell stereocilium organization / inner ear auditory receptor cell differentiation / cell-cell adhesion mediated by cadherin / stereocilium / photoreceptor cell maintenance / Sensory processing of sound by outer hair cells of the cochlea / Sensory processing of sound by inner hair cells of the cochlea / anchoring junction / inner ear morphogenesis / spectrin binding / microvillus membrane / microvillus / homophilic cell-cell adhesion / brush border / actin filament bundle assembly / photoreceptor outer segment / cell adhesion molecule binding / epithelial cell differentiation / photoreceptor inner segment / brush border membrane / sensory perception of sound / G2/M transition of mitotic cell cycle / apical part of cell / protein-containing complex assembly / cytoskeleton / cilium / apical plasma membrane / calcium ion binding / synapse / plasma membrane / cytosol / cytoplasm類似検索 - 分子機能 Harmonin / Harmonin, N-terminal domain / : / Cadherin conserved site / Cadherin domain signature. / Cadherin repeats. / Cadherin domain / Cadherin-like / Cadherins domain profile. / Cadherin-like superfamily ...Harmonin / Harmonin, N-terminal domain / : / Cadherin conserved site / Cadherin domain signature. / Cadherin repeats. / Cadherin domain / Cadherin-like / Cadherins domain profile. / Cadherin-like superfamily / PDZ domain / Pdz3 Domain / PDZ domain / PDZ domain profile. / Domain present in PSD-95, Dlg, and ZO-1/2. / PDZ domain / PDZ superfamily / Roll / Mainly Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト)
  Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.85 Å 分子置換 / 解像度: 1.85 Å |
|---|
 データ登録者 データ登録者 | Yan, W. / Chen, G. / Li, J. |
|---|
| 資金援助 |  中国, 1件 中国, 1件 | 組織 | 認可番号 | 国 |
|---|
| National Natural Science Foundation of China (NSFC) | |  中国 中国 |
|
|---|
 引用 引用 |  ジャーナル: Faseb J. / 年: 2022 ジャーナル: Faseb J. / 年: 2022
タイトル: Structure of the Harmonin PDZ2 and coiled-coil domains in a complex with CDHR2 tail and its implications.
著者: Yan, W. / Chen, G. / Li, J. |
|---|
| 履歴 | | 登録 | 2022年2月25日 | 登録サイト: PDBJ / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2022年7月6日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 2.0 | 2023年11月29日 | Group: Atomic model / Data collection ...Atomic model / Data collection / Derived calculations / Refinement description
カテゴリ: atom_site / chem_comp_atom ...atom_site / chem_comp_atom / chem_comp_bond / pdbx_initial_refinement_model / pdbx_nonpoly_scheme / pdbx_struct_assembly / pdbx_struct_assembly_gen / pdbx_struct_assembly_prop / pdbx_struct_oper_list
Item: _atom_site.B_iso_or_equiv / _atom_site.Cartn_x ..._atom_site.B_iso_or_equiv / _atom_site.Cartn_x / _atom_site.Cartn_y / _atom_site.Cartn_z / _pdbx_nonpoly_scheme.auth_seq_num / _pdbx_struct_assembly.oligomeric_count / _pdbx_struct_assembly.oligomeric_details / _pdbx_struct_assembly_gen.oper_expression / _pdbx_struct_assembly_prop.value |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.85 Å
分子置換 / 解像度: 1.85 Å  データ登録者
データ登録者 中国, 1件
中国, 1件  引用
引用 ジャーナル: Faseb J. / 年: 2022
ジャーナル: Faseb J. / 年: 2022 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 7x2e.cif.gz
7x2e.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb7x2e.ent.gz
pdb7x2e.ent.gz PDB形式
PDB形式 7x2e.json.gz
7x2e.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 7x2e_validation.pdf.gz
7x2e_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 7x2e_full_validation.pdf.gz
7x2e_full_validation.pdf.gz 7x2e_validation.xml.gz
7x2e_validation.xml.gz 7x2e_validation.cif.gz
7x2e_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/x2/7x2e
https://data.pdbj.org/pub/pdb/validation_reports/x2/7x2e ftp://data.pdbj.org/pub/pdb/validation_reports/x2/7x2e
ftp://data.pdbj.org/pub/pdb/validation_reports/x2/7x2e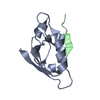
 F&H 検索
F&H 検索 リンク
リンク 集合体
集合体

 要素
要素 Homo sapiens (ヒト) / 遺伝子: USH1C / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: USH1C / 発現宿主: 

 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SSRF
SSRF  / ビームライン: BL18U1 / 波長: 0.97915 Å
/ ビームライン: BL18U1 / 波長: 0.97915 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj





