ソフトウェア 名称 バージョン 分類 5.8.0267精密化 データ削減 データスケーリング 位相決定
精密化 構造決定の手法 開始モデル 解像度 / Cor.coef. Fo :Fc / Cor.coef. Fo :Fc free / SU B / SU ML / 交差検証法 / ESU R / ESU R Free / 詳細 Rfactor 反射数 %反射 Rfree 0.2916 239 5.14 % Rwork 0.2139 4411 - all 0.218 - - obs - 4650 86.399 %
溶媒の処理 イオンプローブ半径 / 減衰半径 / VDWプローブ半径 / 溶媒モデル 原子変位パラメータ Biso mean 2 Baniso -1 Baniso -2 Baniso -3 1- 0.03 Å2 0.015 Å2 0 Å2 2- - 0.03 Å2 -0 Å2 3- - - -0.098 Å2
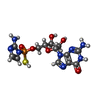
精密化ステップ サイクル / 解像度 タンパク質 核酸 リガンド 溶媒 全体 原子数 0 378 334 49 761
拘束条件 大きな表を表示 (5 x 23) 大きな表を隠す Refine-ID タイプ Dev ideal Dev ideal target 数 X-RAY DIFFRACTION r_bond_refined_d0.018 0.019 796 X-RAY DIFFRACTION r_bond_other_d0.029 0.024 358 X-RAY DIFFRACTION r_angle_refined_deg2.603 2.369 1204 X-RAY DIFFRACTION r_angle_other_deg3.537 2.604 840 X-RAY DIFFRACTION r_chiral_restr0.168 0.2 152 X-RAY DIFFRACTION r_chiral_restr_other1.454 0.2 26 X-RAY DIFFRACTION r_gen_planes_refined0.012 0.021 418 X-RAY DIFFRACTION r_gen_planes_other0.001 0.023 136 X-RAY DIFFRACTION r_nbd_refined0.11 0.2 86 X-RAY DIFFRACTION r_symmetry_nbd_other0.237 0.2 460 X-RAY DIFFRACTION r_nbtor_refined0.247 0.2 315 X-RAY DIFFRACTION r_symmetry_nbtor_other0.277 0.2 195 X-RAY DIFFRACTION r_xyhbond_nbd_refined0.19 0.2 40 X-RAY DIFFRACTION r_symmetry_xyhbond_nbd_other0.019 0.2 1 X-RAY DIFFRACTION r_symmetry_nbd_refined0.17 0.2 16 X-RAY DIFFRACTION r_nbd_other0.217 0.2 62 X-RAY DIFFRACTION r_symmetry_xyhbond_nbd_refined0.243 0.2 13 X-RAY DIFFRACTION r_scbond_it2.764 2.326 794 X-RAY DIFFRACTION r_scbond_other2.762 2.317 787 X-RAY DIFFRACTION r_scangle_it4.778 3.468 1204 X-RAY DIFFRACTION r_scangle_other4.788 3.445 1193 X-RAY DIFFRACTION r_lrange_it11.144 20.633 1085 X-RAY DIFFRACTION r_lrange_other11.16 20.574 1078
LS精密化 シェル 大きな表を表示 (7 x 20) 大きな表を隠す 解像度 (Å)Rfactor Rfree Num. reflection Rfree Rfactor Rwork Num. reflection Rwork Refine-ID % reflection obs (%)2.14-2.196 0.248 6 0.241 157 X-RAY DIFFRACTION 45.0276 2.196-2.256 0.262 17 0.278 181 X-RAY DIFFRACTION 50 2.256-2.321 0.388 18 0.25 193 X-RAY DIFFRACTION 58.7744 2.321-2.393 0.321 15 0.241 228 X-RAY DIFFRACTION 67.313 2.393-2.471 0.315 26 0.234 254 X-RAY DIFFRACTION 82.3529 2.471-2.558 0.392 21 0.25 316 X-RAY DIFFRACTION 99.7041 2.558-2.654 0.422 23 0.243 308 X-RAY DIFFRACTION 99.6988 2.654-2.762 0.396 20 0.247 286 X-RAY DIFFRACTION 100 2.762-2.885 0.261 14 0.236 294 X-RAY DIFFRACTION 100 2.885-3.026 0.254 9 0.226 278 X-RAY DIFFRACTION 100 3.026-3.189 0.269 9 0.226 263 X-RAY DIFFRACTION 98.9091 3.189-3.382 0.228 5 0.221 259 X-RAY DIFFRACTION 99.6226 3.382-3.616 0.179 1 0.183 246 X-RAY DIFFRACTION 100 3.616-3.905 0.291 5 0.171 239 X-RAY DIFFRACTION 99.5918 3.905-4.276 0.181 16 0.16 198 X-RAY DIFFRACTION 100 4.276-4.78 0.118 8 0.172 193 X-RAY DIFFRACTION 100 4.78-5.516 0.214 7 0.166 169 X-RAY DIFFRACTION 100 5.516-6.749 0.274 10 0.234 146 X-RAY DIFFRACTION 100 6.749-9.515 0.422 6 0.231 122 X-RAY DIFFRACTION 100 9.515-42.948 1.324 3 0.265 81 X-RAY DIFFRACTION 97.6744
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.14 Å
分子置換 / 解像度: 2.14 Å  データ登録者
データ登録者 米国, 2件
米国, 2件  引用
引用 ジャーナル: J.Am.Chem.Soc. / 年: 2024
ジャーナル: J.Am.Chem.Soc. / 年: 2024 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 7u88.cif.gz
7u88.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb7u88.ent.gz
pdb7u88.ent.gz PDB形式
PDB形式 7u88.json.gz
7u88.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 7u88_validation.pdf.gz
7u88_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 7u88_full_validation.pdf.gz
7u88_full_validation.pdf.gz 7u88_validation.xml.gz
7u88_validation.xml.gz 7u88_validation.cif.gz
7u88_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/u8/7u88
https://data.pdbj.org/pub/pdb/validation_reports/u8/7u88 ftp://data.pdbj.org/pub/pdb/validation_reports/u8/7u88
ftp://data.pdbj.org/pub/pdb/validation_reports/u8/7u88 リンク
リンク 集合体
集合体
 要素
要素 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ALS
ALS  / ビームライン: 8.2.2 / 波長: 1 Å
/ ビームライン: 8.2.2 / 波長: 1 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー






 PDBj
PDBj