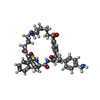
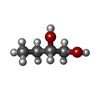
| 登録情報 | データベース: PDB / ID: 7ths
|
|---|
| タイトル | Macrocyclic plasmin inhibitor |
|---|
 要素 要素 | Plasminogen |
|---|
 キーワード キーワード | BLOOD CLOTTING / Hydrolase/Inhibitor / protease / inhibitor / Hydrolase-Inhibitor complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
plasmin / trans-synaptic signaling by BDNF, modulating synaptic transmission / trophoblast giant cell differentiation / tissue remodeling / tissue regeneration / Signaling by PDGF / mononuclear cell migration / positive regulation of fibrinolysis / negative regulation of cell-cell adhesion mediated by cadherin / protein antigen binding ...plasmin / trans-synaptic signaling by BDNF, modulating synaptic transmission / trophoblast giant cell differentiation / tissue remodeling / tissue regeneration / Signaling by PDGF / mononuclear cell migration / positive regulation of fibrinolysis / negative regulation of cell-cell adhesion mediated by cadherin / protein antigen binding / Dissolution of Fibrin Clot / myoblast differentiation / biological process involved in interaction with symbiont / labyrinthine layer blood vessel development / muscle cell cellular homeostasis / Activation of Matrix Metalloproteinases / apolipoprotein binding / extracellular matrix disassembly / negative regulation of cell-substrate adhesion / positive regulation of blood vessel endothelial cell migration / negative regulation of fibrinolysis / fibrinolysis / Degradation of the extracellular matrix / serine-type peptidase activity / platelet alpha granule lumen / protein processing / kinase binding / Schaffer collateral - CA1 synapse / Regulation of Insulin-like Growth Factor (IGF) transport and uptake by Insulin-like Growth Factor Binding Proteins (IGFBPs) / blood coagulation / Platelet degranulation / protein-folding chaperone binding / : / protease binding / blood microparticle / endopeptidase activity / protein domain specific binding / signaling receptor binding / negative regulation of cell population proliferation / serine-type endopeptidase activity / external side of plasma membrane / glutamatergic synapse / enzyme binding / cell surface / proteolysis / extracellular space / extracellular exosome / extracellular region / plasma membrane類似検索 - 分子機能 Peptidase S1A, plasmin / divergent subfamily of APPLE domains / : / PAN/Apple domain profile. / PAN domain / PAN/Apple domain / Kringle domain / Kringle / Kringle, conserved site / Kringle superfamily ...Peptidase S1A, plasmin / divergent subfamily of APPLE domains / : / PAN/Apple domain profile. / PAN domain / PAN/Apple domain / Kringle domain / Kringle / Kringle, conserved site / Kringle superfamily / Kringle domain signature. / Kringle domain profile. / Kringle domain / Kringle-like fold / Serine proteases, trypsin family, histidine active site / Serine proteases, trypsin family, serine active site / Serine proteases, trypsin family, histidine active site. / Peptidase S1A, chymotrypsin family / Serine proteases, trypsin family, serine active site. / Serine proteases, trypsin domain profile. / Trypsin-like serine protease / Serine proteases, trypsin domain / Trypsin / Peptidase S1, PA clan, chymotrypsin-like fold / Peptidase S1, PA clan類似検索 - ドメイン・相同性 (2S)-butane-1,2-diol / Chem-I5Y / Plasminogen類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.8 Å 分子置換 / 解像度: 1.8 Å |
|---|
 データ登録者 データ登録者 | Guojie, W. |
|---|
| 資金援助 |  オーストラリア, 1件 オーストラリア, 1件 | 組織 | 認可番号 | 国 |
|---|
| National Health and Medical Research Council (NHMRC, Australia) | APP1127593 |  オーストラリア オーストラリア |
|
|---|
 引用 引用 |  ジャーナル: Chemmedchem / 年: 2023 ジャーナル: Chemmedchem / 年: 2023
タイトル: Synthesis and Structural Characterization of Macrocyclic Plasmin Inhibitors.
著者: Wiedemeyer, S.J.A. / Wu, G. / Pham, T.L.P. / Lang-Henkel, H. / Perez Urzua, B. / Whisstock, J.C. / Law, R.H.P. / Steinmetzer, T. |
|---|
| 履歴 | | 登録 | 2022年1月12日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2023年1月18日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2023年8月2日 | Group: Database references / Source and taxonomy / カテゴリ: citation / citation_author / entity_src_gen
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year / _entity_src_gen.pdbx_host_org_ncbi_taxonomy_id / _entity_src_gen.pdbx_host_org_scientific_name / _entity_src_gen.pdbx_host_org_strain |
|---|
| 改定 1.2 | 2023年10月25日 | Group: Data collection / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond / pdbx_initial_refinement_model |
|---|
| 改定 1.3 | 2024年10月30日 | Group: Structure summary
カテゴリ: pdbx_entry_details / pdbx_modification_feature
Item: _pdbx_entry_details.has_protein_modification |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.8 Å
分子置換 / 解像度: 1.8 Å  データ登録者
データ登録者 オーストラリア, 1件
オーストラリア, 1件  引用
引用 ジャーナル: Chemmedchem / 年: 2023
ジャーナル: Chemmedchem / 年: 2023 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 7ths.cif.gz
7ths.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb7ths.ent.gz
pdb7ths.ent.gz PDB形式
PDB形式 7ths.json.gz
7ths.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 7ths_validation.pdf.gz
7ths_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 7ths_full_validation.pdf.gz
7ths_full_validation.pdf.gz 7ths_validation.xml.gz
7ths_validation.xml.gz 7ths_validation.cif.gz
7ths_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/th/7ths
https://data.pdbj.org/pub/pdb/validation_reports/th/7ths ftp://data.pdbj.org/pub/pdb/validation_reports/th/7ths
ftp://data.pdbj.org/pub/pdb/validation_reports/th/7ths

 F&H 検索
F&H 検索 リンク
リンク 集合体
集合体


 要素
要素 Homo sapiens (ヒト) / 遺伝子: PLG / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: PLG / 発現宿主:  Homo sapiens (ヒト) / 参照: UniProt: P00747, plasmin
Homo sapiens (ヒト) / 参照: UniProt: P00747, plasmin X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  Australian Synchrotron
Australian Synchrotron  / ビームライン: MX2 / 波長: 0.9537 Å
/ ビームライン: MX2 / 波長: 0.9537 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj