+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7t1p | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Solution structure of 7SK stem-loop 1 with HIV-1 Tat Finland Arginine Rich Motif | |||||||||
 要素 要素 |
| |||||||||
 キーワード キーワード | TRANSCRIPTION / Tat / 7SK / HIV-1 | |||||||||
| 機能・相同性 | RNA / RNA (> 10) 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) HIV-1 06TG.HT008 (ヒト免疫不全ウイルス) HIV-1 06TG.HT008 (ヒト免疫不全ウイルス) | |||||||||
| 手法 | 溶液NMR /  溶液散乱 / simulated annealing 溶液散乱 / simulated annealing | |||||||||
 データ登録者 データ登録者 | Pham, V.V. / Gao, M. / D'Souza, V.M. | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2022 ジャーナル: Commun Biol / 年: 2022タイトル: A structure-based mechanism for displacement of the HEXIM adapter from 7SK small nuclear RNA. 著者: Pham, V.V. / Gao, M. / Meagher, J.L. / Smith, J.L. / D'Souza, V.M. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7t1p.cif.gz 7t1p.cif.gz | 409.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7t1p.ent.gz pdb7t1p.ent.gz | 342.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7t1p.json.gz 7t1p.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/t1/7t1p https://data.pdbj.org/pub/pdb/validation_reports/t1/7t1p ftp://data.pdbj.org/pub/pdb/validation_reports/t1/7t1p ftp://data.pdbj.org/pub/pdb/validation_reports/t1/7t1p | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  7t1nC  7t1oC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
| その他のデータベース |
|
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 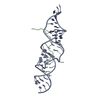
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: RNA鎖 | 分子量: 17986.635 Da / 分子数: 1 / 由来タイプ: 合成 / 由来: (合成)  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|---|
| #2: タンパク質・ペプチド | 分子量: 2144.537 Da / 分子数: 1 / 由来タイプ: 合成 由来: (合成)  HIV-1 06TG.HT008 (ヒト免疫不全ウイルス) HIV-1 06TG.HT008 (ヒト免疫不全ウイルス) |
-実験情報
-実験
| 実験 |
| ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 |
| ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料 |
| ||||||||||||||||||||
| 試料状態 |
|
-データ収集
| NMRスペクトロメーター | タイプ: Bruker DMX / 製造業者: Bruker / モデル: DMX / 磁場強度: 700 MHz |
|---|---|
| Soln scatter | タイプ: neutron / Buffer name: 10 mM phosphate, 70 mM NaCl, 0.1 mM EDTA / Data analysis software list: Scatter / Data reduction software list: ATSAS / Detector specific: Dectris / 検出器タイプ: Pilatus3 X 2M detector / Mean guiner radius: 2.5 nm / Num. of time frames: 10 / Sample pH: 5.6 / Source beamline: 12.3.1 / Source beamline instrument: SIBYLS / Source class: Y / Source type: 12.3.1 (SIBYLS) beam line / 温度: 283 K |
- 解析
解析
| NMR software |
| |||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: simulated annealing / ソフトェア番号: 8 | |||||||||||||||||||||||||||
| 代表構造 | 選択基準: lowest energy | |||||||||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: structures with the lowest energy 計算したコンフォーマーの数: 100 / 登録したコンフォーマーの数: 10 | |||||||||||||||||||||||||||
| Soln scatter model | 手法: This structure was calculated with XPLOR-NIH with a CYANA input and minimized with SAXS. |
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj






























