+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 7sku | ||||||
|---|---|---|---|---|---|---|---|
| Title | Nipah virus matrix protein in complex with PI(4,5)P2 | ||||||
 Components Components | Matrix protein | ||||||
 Keywords Keywords | VIRAL PROTEIN / Matrix / Dimer / Viral assembly / lipid complex | ||||||
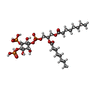
| Function / homology |  Function and homology information Function and homology informationvirion assembly / virion component / structural constituent of virion / host cell cytoplasm / host cell nucleus / host cell plasma membrane Similarity search - Function | ||||||
| Biological species |  Nipah virus Nipah virus | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 2.117 Å MOLECULAR REPLACEMENT / Resolution: 2.117 Å | ||||||
 Authors Authors | Norris, M.J. / Saphire, E.O. | ||||||
| Funding support |  United States, 1items United States, 1items
| ||||||
 Citation Citation |  Journal: Sci Adv / Year: 2022 Journal: Sci Adv / Year: 2022Title: Measles and Nipah virus assembly: Specific lipid binding drives matrix polymerization. Authors: Norris, M.J. / Husby, M.L. / Kiosses, W.B. / Yin, J. / Saxena, R. / Rennick, L.J. / Heiner, A. / Harkins, S.S. / Pokhrel, R. / Schendel, S.L. / Hastie, K.M. / Landeras-Bueno, S. / Salie, Z.L. ...Authors: Norris, M.J. / Husby, M.L. / Kiosses, W.B. / Yin, J. / Saxena, R. / Rennick, L.J. / Heiner, A. / Harkins, S.S. / Pokhrel, R. / Schendel, S.L. / Hastie, K.M. / Landeras-Bueno, S. / Salie, Z.L. / Lee, B. / Chapagain, P.P. / Maisner, A. / Duprex, W.P. / Stahelin, R.V. / Saphire, E.O. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  7sku.cif.gz 7sku.cif.gz | 367 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb7sku.ent.gz pdb7sku.ent.gz | 301.7 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  7sku.json.gz 7sku.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/sk/7sku https://data.pdbj.org/pub/pdb/validation_reports/sk/7sku ftp://data.pdbj.org/pub/pdb/validation_reports/sk/7sku ftp://data.pdbj.org/pub/pdb/validation_reports/sk/7sku | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  7sksC  7sktSC S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| Unit cell |
|
- Components
Components
| #1: Protein | Mass: 43961.277 Da / Num. of mol.: 2 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Nipah virus / Production host: Nipah virus / Production host:  #2: Chemical | #3: Chemical | ChemComp-SO4 / #4: Water | ChemComp-HOH / | Has ligand of interest | Y | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.31 Å3/Da / Density % sol: 46.69 % |
|---|---|
| Crystal grow | Temperature: 293 K / Method: vapor diffusion, sitting drop / pH: 7.5 / Details: 0.1 M HEPES pH 7.5, 2.0 M ammonium sulfate |
-Data collection
| Diffraction | Mean temperature: 100 K / Serial crystal experiment: N | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  APS APS  / Beamline: 23-ID-B / Wavelength: 1.03317 Å / Beamline: 23-ID-B / Wavelength: 1.03317 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Detector | Type: DECTRIS EIGER X 16M / Detector: PIXEL / Date: Aug 9, 2019 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation wavelength | Wavelength: 1.03317 Å / Relative weight: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection | Resolution: 2.117→75.18 Å / Num. obs: 45637 / % possible obs: 96.9 % / Redundancy: 12.71 % Details: Some remarks regarding the mmCIF items written, the PDB Exchange Dictionary (PDBx/mmCIF) Version 5.0 supporting the data files in the current PDB archive (dictionary version 5.325, last ...Details: Some remarks regarding the mmCIF items written, the PDB Exchange Dictionary (PDBx/mmCIF) Version 5.0 supporting the data files in the current PDB archive (dictionary version 5.325, last updated 2020-04-13: http://mmcif.wwpdb.org/dictionaries/mmcif_pdbx_v50.dic/Index/) and the actual quantities provided by MRFANA (https://github.com/githubgphl/MRFANA) from the autoPROC package (https://www.globalphasing.com/autoproc/). In general, the mmCIF categories here should provide items that are currently used in the PDB archive. If there are alternatives, the one recommended by the PDB developers has been selected. The distinction between *_all and *_obs quantities is not always clear: often only one version is actively used within the PDB archive (or is the one recommended by PDB developers). The intention of distinguishing between classes of reflections before and after some kind of observation criterion was applied, can in principle be useful - but such criteria change in various ways throughout the data processing steps (rejection of overloaded or too partial reflections, outlier/misfit rejections during scaling etc) and there is no retrospect computation of data scaling/merging statistics for the reflections used in the final refinement (where another observation criterion might have been applied). Typical data processing will usually only provide one version of statistics at various stages and these are given in the recommended item here, irrespective of the "_all" and "_obs" connotation, see e.g. the use of _reflns.pdbx_Rmerge_I_obs, _reflns.pdbx_Rrim_I_all and _reflns.pdbx_Rpim_I_all. Please note that all statistics related to "merged intensities" (or "merging") are based on inverse-variance weighting of the individual measurements making up a symmetry-unique reflection. This is standard for several decades now, even if some of the dictionary definitions seem to suggest that a simple "mean" or "average" intensity is being used instead. R-values are always given for all symmetry-equivalent reflections following Friedel's law, i.e. Bijvoet pairs are not treated separately (since we want to describe the overall mean intensity and not the mean I(+) and I(-) here). The Rrim metric is identical to the Rmeas R-value and only differs in name. _reflns.pdbx_number_measured_all is the number of measured intensities just before the final merging step (at which point no additional rejection takes place). _reflns.number_obs is the number of symmetry-unique observations, i.e. the result of merging those measurements via inverse-variance weighting. _reflns.pdbx_netI_over_sigmaI is based on the merged intensities (_reflns.number_obs) as expected. _reflns.pdbx_redundancy is synonymous with "multiplicity". The per-shell item _reflns_shell.number_measured_all corresponds to the overall value _reflns.pdbx_number_measured_all. The per-shell item _reflns_shell.number_unique_all corresponds to the overall value _reflns.number_obs. The per-shell item _reflns_shell.percent_possible_all corresponds to the overall value _reflns.percent_possible_obs. The per-shell item _reflns_shell.meanI_over_sigI_obs corresponds to the overall value given as _reflns.pdbx_netI_over_sigmaI. But be aware of the incorrect definition of the former in the current dictionary! CC1/2: 0.998 / CC1/2 anomalous: -0.076 / Rmerge(I) obs: 0.2011 / Rpim(I) all: 0.0579 / Rrim(I) all: 0.2095 / AbsDiff over sigma anomalous: 0.755 / Net I/σ(I): 9.95 / Num. measured all: 579838 / % possible anomalous: 96.8 / Redundancy anomalous: 6.7 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection shell |
|
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 7SKT Resolution: 2.117→75.18 Å / Cor.coef. Fo:Fc: 0.934 / Cor.coef. Fo:Fc free: 0.925 / SU R Cruickshank DPI: 0.451 / Cross valid method: THROUGHOUT / SU R Blow DPI: 0.232 / SU Rfree Blow DPI: 0.192 / SU Rfree Cruickshank DPI: 0.189
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso mean: 53.95 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.33 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 2.117→75.18 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Resolution: 2.12→2.13 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller



 PDBj
PDBj