| 登録情報 | データベース: PDB / ID: 7sie
|
|---|
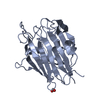
| タイトル | Structure of AAP A-domain (residues 351-605) from Staphylococcus epidermidis |
|---|
 要素 要素 | Accumulation associated protein |
|---|
 キーワード キーワード | CELL ADHESION / L-type lectin / Staphylococcus epidermidis |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
E domain / E domain / G5 domain / G5 domain / G5 domain profile. / G5 / : / YSIRK type signal peptide / YSIRK Gram-positive signal peptide / LPXTG cell wall anchor motif ...E domain / E domain / G5 domain / G5 domain / G5 domain profile. / G5 / : / YSIRK type signal peptide / YSIRK Gram-positive signal peptide / LPXTG cell wall anchor motif / Gram-positive cocci surface proteins LPxTG motif profile. / LPXTG cell wall anchor domain類似検索 - ドメイン・相同性 |
|---|
| 生物種 |   Staphylococcus epidermidis (表皮ブドウ球菌) Staphylococcus epidermidis (表皮ブドウ球菌) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  単波長異常分散 / 解像度: 1.3 Å 単波長異常分散 / 解像度: 1.3 Å |
|---|
 データ登録者 データ登録者 | Atkin, K.E. / Brentnall, A.S. / Dodson, E.J. / Whelan, F. / Clark, L. / Turkenburg, J.P. / Potts, J.R. |
|---|
| 資金援助 |  英国, 3件 英国, 3件 | 組織 | 認可番号 | 国 |
|---|
| Biotechnology and Biological Sciences Research Council (BBSRC) | BB/J005029/ |  英国 英国 | | British Heart Foundation | FS/12/36/29588 |  英国 英国 | | British Heart Foundation | PG/17/19/32862 |  英国 英国 |
|
|---|
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2023 ジャーナル: J.Biol.Chem. / 年: 2023
タイトル: Staphylococcal Periscope proteins Aap, SasG, and Pls project noncanonical legume-like lectin adhesin domains from the bacterial surface.
著者: Clark, L.C. / Atkin, K.E. / Whelan, F. / Brentnall, A.S. / Harris, G. / Towell, A.M. / Turkenburg, J.P. / Liu, Y. / Feizi, T. / Griffiths, S.C. / Geoghegan, J.A. / Potts, J.R. |
|---|
| 履歴 | | 登録 | 2021年10月13日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 置き換え | 2022年10月19日 | ID: 4CCI |
|---|
| 改定 1.0 | 2022年10月19日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2023年5月3日 | Group: Database references / カテゴリ: citation / citation_author
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_ASTM / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year |
|---|
| 改定 1.2 | 2024年5月22日 | Group: Data collection / カテゴリ: chem_comp_atom / chem_comp_bond |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  単波長異常分散 / 解像度: 1.3 Å
単波長異常分散 / 解像度: 1.3 Å  データ登録者
データ登録者 英国, 3件
英国, 3件  引用
引用 ジャーナル: J.Biol.Chem. / 年: 2023
ジャーナル: J.Biol.Chem. / 年: 2023 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 7sie.cif.gz
7sie.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb7sie.ent.gz
pdb7sie.ent.gz PDB形式
PDB形式 7sie.json.gz
7sie.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 7sie_validation.pdf.gz
7sie_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 7sie_full_validation.pdf.gz
7sie_full_validation.pdf.gz 7sie_validation.xml.gz
7sie_validation.xml.gz 7sie_validation.cif.gz
7sie_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/si/7sie
https://data.pdbj.org/pub/pdb/validation_reports/si/7sie ftp://data.pdbj.org/pub/pdb/validation_reports/si/7sie
ftp://data.pdbj.org/pub/pdb/validation_reports/si/7sie

 F&H 検索
F&H 検索 リンク
リンク 集合体
集合体
 要素
要素 Staphylococcus epidermidis (strain ATCC 35984 / RP62A) (表皮ブドウ球菌)
Staphylococcus epidermidis (strain ATCC 35984 / RP62A) (表皮ブドウ球菌)
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  Diamond
Diamond  / ビームライン: I03 / 波長: 0.9762 Å
/ ビームライン: I03 / 波長: 0.9762 Å 解析
解析 単波長異常分散 / 解像度: 1.3→16.53 Å / Cor.coef. Fo:Fc: 0.98 / Cor.coef. Fo:Fc free: 0.976 / SU B: 0.769 / SU ML: 0.015 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.033 / ESU R Free: 0.033 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD
単波長異常分散 / 解像度: 1.3→16.53 Å / Cor.coef. Fo:Fc: 0.98 / Cor.coef. Fo:Fc free: 0.976 / SU B: 0.769 / SU ML: 0.015 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.033 / ESU R Free: 0.033 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj




