| 登録情報 | データベース: PDB / ID: 7pb0
|
|---|
| タイトル | Structure of the human heterotetrameric cis-prenyltransferase complex in complex with magnesium, GGsPP and IsPP |
|---|
 要素 要素 | (Dehydrodolichyl diphosphate synthase complex subunit ...) x 2 |
|---|
 キーワード キーワード | TRANSFERASE / DHDDS / NgBR / hcit / cis-prenyltransferase / dolichol |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
dolichyl diphosphate biosynthetic process / protein mannosylation / Defective DHDDS causes RP59 / : / dehydrodolichyl diphosphate synthase complex / Synthesis of dolichyl-phosphate / regulation of intracellular cholesterol transport / ditrans,polycis-polyprenyl diphosphate synthase [(2E,6E)-farnesyl diphosphate specific] / ditrans,polycis-polyprenyl diphosphate synthase [(2E,6E)-farnesyl diphosphate specific] activity / polyprenol biosynthetic process ...dolichyl diphosphate biosynthetic process / protein mannosylation / Defective DHDDS causes RP59 / : / dehydrodolichyl diphosphate synthase complex / Synthesis of dolichyl-phosphate / regulation of intracellular cholesterol transport / ditrans,polycis-polyprenyl diphosphate synthase [(2E,6E)-farnesyl diphosphate specific] / ditrans,polycis-polyprenyl diphosphate synthase [(2E,6E)-farnesyl diphosphate specific] activity / polyprenol biosynthetic process / vascular endothelial growth factor signaling pathway / protein glycosylation / positive regulation of blood vessel endothelial cell migration / positive regulation of nitric-oxide synthase activity / cholesterol homeostasis / angiogenesis / cell differentiation / endoplasmic reticulum membrane / endoplasmic reticulum / metal ion binding類似検索 - 分子機能 Dehydrodolichyl diphosphate synthase complex subunit Nus1 / Di-trans-poly-cis-decaprenylcistransferase-like, conserved site / Undecaprenyl pyrophosphate synthase family signature. / Undecaprenyl pyrophosphate synthetase / Decaprenyl diphosphate synthase-like / Decaprenyl diphosphate synthase-like / Putative undecaprenyl diphosphate synthase / Decaprenyl diphosphate synthase-like superfamily / 3-Layer(aba) Sandwich / Alpha Beta類似検索 - ドメイン・相同性 Chem-GGS / Chem-ISY / Dehydrodolichyl diphosphate synthase complex subunit DHDDS / Dehydrodolichyl diphosphate synthase complex subunit NUS1類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.301 Å 分子置換 / 解像度: 2.301 Å |
|---|
 データ登録者 データ登録者 | Giladi, M. / Lisnyansky Bar-El, M. / Haitin, Y. |
|---|
| 資金援助 |  イスラエル, 7件 イスラエル, 7件 | 組織 | 認可番号 | 国 |
|---|
| Other private | ICRF-19202 |  イスラエル イスラエル | | Israel Science Foundation | 1721/16 |  イスラエル イスラエル | | Other private | Recanati Foundation for Medical Research |  イスラエル イスラエル | | German-Israeli Foundation for Research and Development | I2425418.13/2016 |  イスラエル イスラエル | | Other government | ICORE-1775/12 |  イスラエル イスラエル | | Other private | Tel Aviv Sourasky Medical Center Orion Project |  イスラエル イスラエル | | Other private | ICA-20200037 |  イスラエル イスラエル |
|
|---|
 引用 引用 |  ジャーナル: Sci Adv / 年: 2022 ジャーナル: Sci Adv / 年: 2022
タイトル: Structural basis for long-chain isoprenoid synthesis by cis -prenyltransferases.
著者: Giladi, M. / Lisnyansky Bar-El, M. / Vankova, P. / Ferofontov, A. / Melvin, E. / Alkaderi, S. / Kavan, D. / Redko, B. / Haimov, E. / Wiener, R. / Man, P. / Haitin, Y. |
|---|
| 履歴 | | 登録 | 2021年7月30日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2022年6月1日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2024年1月31日 | Group: Data collection / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond / pdbx_initial_refinement_model |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.301 Å
分子置換 / 解像度: 2.301 Å  データ登録者
データ登録者 イスラエル, 7件
イスラエル, 7件  引用
引用 ジャーナル: Sci Adv / 年: 2022
ジャーナル: Sci Adv / 年: 2022 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 7pb0.cif.gz
7pb0.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb7pb0.ent.gz
pdb7pb0.ent.gz PDB形式
PDB形式 7pb0.json.gz
7pb0.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/pb/7pb0
https://data.pdbj.org/pub/pdb/validation_reports/pb/7pb0 ftp://data.pdbj.org/pub/pdb/validation_reports/pb/7pb0
ftp://data.pdbj.org/pub/pdb/validation_reports/pb/7pb0 リンク
リンク 集合体
集合体

 要素
要素 Homo sapiens (ヒト) / 遺伝子: DHDDS, HDS / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: DHDDS, HDS / 発現宿主: 
 Homo sapiens (ヒト) / 遺伝子: NUS1, C6orf68, NGBR / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: NUS1, C6orf68, NGBR / 発現宿主: 
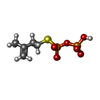
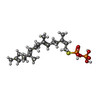





 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ESRF
ESRF  / ビームライン: ID30B / 波長: 1 Å
/ ビームライン: ID30B / 波長: 1 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー







 PDBj
PDBj


