+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7f4v | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of a primordial cyanobacterial photosystem I | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | PHOTOSYNTHESIS / Photosystem I / ELECTRON TRANSPORT | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報thylakoid membrane / photosystem I reaction center / photosystem I / photosynthetic electron transport in photosystem I / photosystem I / chlorophyll binding / photosynthesis / 4 iron, 4 sulfur cluster binding / electron transfer activity / oxidoreductase activity ...thylakoid membrane / photosystem I reaction center / photosystem I / photosynthetic electron transport in photosystem I / photosystem I / chlorophyll binding / photosynthesis / 4 iron, 4 sulfur cluster binding / electron transfer activity / oxidoreductase activity / magnesium ion binding / metal ion binding / plasma membrane 類似検索 - 分子機能 | ||||||
| 生物種 |  Gloeobacter violaceus (バクテリア) Gloeobacter violaceus (バクテリア) | ||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.04 Å | ||||||
 データ登録者 データ登録者 | Kato, K. / Hamaguchi, T. / Nagao, R. / Kawakami, K. / Yonekura, K. / Shen, J.R. | ||||||
 引用 引用 |  ジャーナル: Biorxiv / 年: 2022 ジャーナル: Biorxiv / 年: 2022タイトル: Structural basis for the absence of low-energy chlorophylls responsible for photoprotection from a primitive cyanobacterial PSI 著者: Kato, K. / Hamaguchi, T. / Nagao, R. / Kawakami, K. / Ueno, Y. / Suzuki, T. / Uchida, H. / Murakami, A. / Nakajima, Y. / Yokono, M. / Akimoto, S. / Dohmae, N. / Yonekura, K. / Shen, J.R. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7f4v.cif.gz 7f4v.cif.gz | 1.6 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7f4v.ent.gz pdb7f4v.ent.gz | 表示 |  PDB形式 PDB形式 | |
| PDBx/mmJSON形式 |  7f4v.json.gz 7f4v.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7f4v_validation.pdf.gz 7f4v_validation.pdf.gz | 18 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7f4v_full_validation.pdf.gz 7f4v_full_validation.pdf.gz | 19.5 MB | 表示 | |
| XML形式データ |  7f4v_validation.xml.gz 7f4v_validation.xml.gz | 339.6 KB | 表示 | |
| CIF形式データ |  7f4v_validation.cif.gz 7f4v_validation.cif.gz | 442.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/f4/7f4v https://data.pdbj.org/pub/pdb/validation_reports/f4/7f4v ftp://data.pdbj.org/pub/pdb/validation_reports/f4/7f4v ftp://data.pdbj.org/pub/pdb/validation_reports/f4/7f4v | HTTPS FTP |
-関連構造データ
| 関連構造データ |  31455MC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-Photosystem I P700 chlorophyll a apoprotein ... , 2種, 6分子 aAbAcAaBbBcB
| #1: タンパク質 | 分子量: 86505.219 Da / 分子数: 3 / 由来タイプ: 天然 由来: (天然)  Gloeobacter violaceus (strain ATCC 29082 / PCC 7421) (バクテリア) Gloeobacter violaceus (strain ATCC 29082 / PCC 7421) (バクテリア)株: ATCC 29082 / PCC 7421 / 参照: UniProt: Q7NFT6, photosystem I #2: タンパク質 | 分子量: 96166.758 Da / 分子数: 3 / 由来タイプ: 天然 由来: (天然)  Gloeobacter violaceus (strain ATCC 29082 / PCC 7421) (バクテリア) Gloeobacter violaceus (strain ATCC 29082 / PCC 7421) (バクテリア)株: ATCC 29082 / PCC 7421 / 参照: UniProt: Q7NFT5, photosystem I |
|---|
-Photosystem I reaction center subunit ... , 6種, 18分子 aDbDcDaEbEcEaFbFcFaIbIcIaLbLcLaMbMcM
| #4: タンパク質 | 分子量: 15952.242 Da / 分子数: 3 / 由来タイプ: 天然 由来: (天然)  Gloeobacter violaceus (strain ATCC 29082 / PCC 7421) (バクテリア) Gloeobacter violaceus (strain ATCC 29082 / PCC 7421) (バクテリア)株: ATCC 29082 / PCC 7421 / 参照: UniProt: Q7NF26 #5: タンパク質 | 分子量: 7468.415 Da / 分子数: 3 / 由来タイプ: 天然 由来: (天然)  Gloeobacter violaceus (strain ATCC 29082 / PCC 7421) (バクテリア) Gloeobacter violaceus (strain ATCC 29082 / PCC 7421) (バクテリア)株: ATCC 29082 / PCC 7421 / 参照: UniProt: Q7NFW6 #6: タンパク質 | 分子量: 19931.127 Da / 分子数: 3 / 由来タイプ: 天然 由来: (天然)  Gloeobacter violaceus (strain ATCC 29082 / PCC 7421) (バクテリア) Gloeobacter violaceus (strain ATCC 29082 / PCC 7421) (バクテリア)株: ATCC 29082 / PCC 7421 / 参照: UniProt: Q7NH05 #7: タンパク質・ペプチド | 分子量: 3853.611 Da / 分子数: 3 / 由来タイプ: 天然 由来: (天然)  Gloeobacter violaceus (strain ATCC 29082 / PCC 7421) (バクテリア) Gloeobacter violaceus (strain ATCC 29082 / PCC 7421) (バクテリア)株: ATCC 29082 / PCC 7421 / 参照: UniProt: P0C029 #9: タンパク質 | 分子量: 15806.324 Da / 分子数: 3 / 由来タイプ: 天然 由来: (天然)  Gloeobacter violaceus (strain ATCC 29082 / PCC 7421) (バクテリア) Gloeobacter violaceus (strain ATCC 29082 / PCC 7421) (バクテリア)株: ATCC 29082 / PCC 7421 / 参照: UniProt: Q7NIE7 #10: タンパク質・ペプチド | 分子量: 3212.907 Da / 分子数: 3 / 由来タイプ: 天然 由来: (天然)  Gloeobacter violaceus (strain ATCC 29082 / PCC 7421) (バクテリア) Gloeobacter violaceus (strain ATCC 29082 / PCC 7421) (バクテリア)株: ATCC 29082 / PCC 7421 / 参照: UniProt: Q7NHY3 |
|---|
-タンパク質 / タンパク質・ペプチド , 2種, 6分子 aCbCcCaJbJcJ
| #3: タンパク質 | 分子量: 8807.170 Da / 分子数: 3 / 由来タイプ: 天然 由来: (天然)  Gloeobacter violaceus (strain ATCC 29082 / PCC 7421) (バクテリア) Gloeobacter violaceus (strain ATCC 29082 / PCC 7421) (バクテリア)株: ATCC 29082 / PCC 7421 / 参照: UniProt: Q7NG86, photosystem I #8: タンパク質・ペプチド | 分子量: 2826.475 Da / 分子数: 3 / 由来タイプ: 天然 由来: (天然)  Gloeobacter violaceus (strain ATCC 29082 / PCC 7421) (バクテリア) Gloeobacter violaceus (strain ATCC 29082 / PCC 7421) (バクテリア)株: ATCC 29082 / PCC 7421 |
|---|
-非ポリマー , 8種, 756分子 

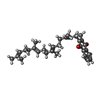












| #11: 化合物 | | #12: 化合物 | ChemComp-CLA / #13: 化合物 | ChemComp-1L3 / #14: 化合物 | ChemComp-SF4 / #15: 化合物 | ChemComp-BCR / #16: 化合物 | ChemComp-LHG / #17: 化合物 | #18: 水 | ChemComp-HOH / | |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | N |
|---|---|
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: PSI trimer / タイプ: COMPLEX / Entity ID: #1-#10 / 由来: NATURAL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 値: 1.1 MDa / 実験値: NO | ||||||||||||
| 由来(天然) | 生物種:  Gloeobacter violaceus PCC 7421 (バクテリア) Gloeobacter violaceus PCC 7421 (バクテリア) | ||||||||||||
| 緩衝液 | pH: 6.5 | ||||||||||||
| 緩衝液成分 |
| ||||||||||||
| 試料 | 濃度: 1.68 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES | ||||||||||||
| 試料支持 | グリッドの材料: COPPER / グリッドのサイズ: 200 divisions/in. / グリッドのタイプ: Quantifoil R1.2/1.3 | ||||||||||||
| 急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 277 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 顕微鏡 | モデル: JEOL CRYO ARM 300 |
|---|---|
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD |
| 撮影 | 電子線照射量: 70.22 e/Å2 / 検出モード: COUNTING / フィルム・検出器のモデル: GATAN K3 (6k x 4k) |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||
| 対称性 | 点対称性: C3 (3回回転対称) | ||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 2.04 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 261743 / アルゴリズム: FOURIER SPACE / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: FLEXIBLE FIT / 空間: REAL |
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj
















