+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7dpd | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Human MCM9 N-terminal domain | ||||||
 要素 要素 | DNA helicase MCM9 | ||||||
 キーワード キーワード | DNA BINDING PROTEIN / Zinc Finger / DNA binding | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報MutLbeta complex binding / MutSbeta complex binding / recombinational interstrand cross-link repair / MCM8-MCM9 complex / mismatch repair involved in maintenance of fidelity involved in DNA-dependent DNA replication / female gamete generation / MutSalpha complex binding / MCM complex / protein localization to chromatin / DNA helicase activity ...MutLbeta complex binding / MutSbeta complex binding / recombinational interstrand cross-link repair / MCM8-MCM9 complex / mismatch repair involved in maintenance of fidelity involved in DNA-dependent DNA replication / female gamete generation / MutSalpha complex binding / MCM complex / protein localization to chromatin / DNA helicase activity / double-strand break repair via homologous recombination / single-stranded DNA binding / chromosome / DNA helicase / DNA damage response / chromatin binding / protein-containing complex binding / enzyme binding / ATP hydrolysis activity / ATP binding / nucleus 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.55 Å 分子置換 / 解像度: 2.55 Å | ||||||
 データ登録者 データ登録者 | Li, J. / Liu, L. / Liu, Y. | ||||||
 引用 引用 |  ジャーナル: Structure / 年: 2021 ジャーナル: Structure / 年: 2021タイトル: Structural study of the N-terminal domain of human MCM8/9 complex. 著者: Jun Li / Daqi Yu / Lan Liu / Huanhuan Liang / Qi Ouyang / Yingfang Liu /  要旨: MCM8/9 is a complex involved in homologous recombination (HR) repair pathway. MCM8/9 dysfunction can cause genome instability and result in primary ovarian insufficiency (POI). However, the mechanism ...MCM8/9 is a complex involved in homologous recombination (HR) repair pathway. MCM8/9 dysfunction can cause genome instability and result in primary ovarian insufficiency (POI). However, the mechanism underlying these effects is largely unknown. Here, we report crystal structures of the N-terminal domains (NTDs) of MCM8 and MCM9, and build a ring-shaped NTD structure based on a 6.6 Å resolution cryoelectron microscopy map. This shows that the MCM8/9 complex forms a 3:3 heterohexamer in an alternating pattern. A positively charged DNA binding channel and a putative ssDNA exit pathway for fork DNA unwinding are revealed. Based on the atomic model, the potential effects of the clinical POI mutants are interpreted. Surprisingly, the zinc-finger motifs are found to be capable of binding an iron atom as well. Overall, our results provide a model for the formation of the MCM8/9 complex and provide a path for further studies. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7dpd.cif.gz 7dpd.cif.gz | 127.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7dpd.ent.gz pdb7dpd.ent.gz | 96.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7dpd.json.gz 7dpd.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7dpd_validation.pdf.gz 7dpd_validation.pdf.gz | 446.6 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7dpd_full_validation.pdf.gz 7dpd_full_validation.pdf.gz | 451.4 KB | 表示 | |
| XML形式データ |  7dpd_validation.xml.gz 7dpd_validation.xml.gz | 22.8 KB | 表示 | |
| CIF形式データ |  7dpd_validation.cif.gz 7dpd_validation.cif.gz | 32.2 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/dp/7dpd https://data.pdbj.org/pub/pdb/validation_reports/dp/7dpd ftp://data.pdbj.org/pub/pdb/validation_reports/dp/7dpd ftp://data.pdbj.org/pub/pdb/validation_reports/dp/7dpd | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 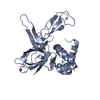
| ||||||||||||
| 2 | 
| ||||||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 33073.582 Da / 分子数: 2 / 断片: N-terminal domain / 変異: R168P,C257A / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: MCM9, C6orf61, MCMDC1 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: MCM9, C6orf61, MCMDC1 / 発現宿主:  #2: 化合物 | #3: 化合物 | ChemComp-NA / | #4: 水 | ChemComp-HOH / | 研究の焦点であるリガンドがあるか | N | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.83 Å3/Da / 溶媒含有率: 56.6 % |
|---|---|
| 結晶化 | 温度: 289 K / 手法: 蒸気拡散法, ハンギングドロップ法 詳細: 0.1M Li2SO4, 0.1M ADA pH 6.2-6.5, 13-16% (w/v) PEG 4000 |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SSRF SSRF  / ビームライン: BL18U1 / 波長: 0.97853 Å / ビームライン: BL18U1 / 波長: 0.97853 Å |
| 検出器 | タイプ: DECTRIS PILATUS3 6M / 検出器: PIXEL / 日付: 2019年6月6日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.97853 Å / 相対比: 1 |
| 反射 | 解像度: 2.55→50 Å / Num. obs: 25165 / % possible obs: 99.9 % / 冗長度: 5.6 % / Biso Wilson estimate: 38.08 Å2 / CC1/2: 0.987 / CC star: 0.997 / Rpim(I) all: 0.075 / Rrim(I) all: 0.183 / Rsym value: 0.166 / Χ2: 0.884 / Net I/σ(I): 9.62 |
| 反射 シェル | 解像度: 2.55→2.59 Å / 冗長度: 4.4 % / Mean I/σ(I) obs: 2.22 / Num. unique obs: 1250 / CC1/2: 0.806 / CC star: 0.945 / Rpim(I) all: 0.382 / Rrim(I) all: 0.821 / Rsym value: 0.723 / Χ2: 0.712 / % possible all: 99.9 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: Rossetta predicted model 解像度: 2.55→34.11 Å / SU ML: 0.3299 / 交差検証法: FREE R-VALUE / σ(F): 1.35 / 位相誤差: 28.0471 / 立体化学のターゲット値: CDL v1.2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 48.63 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.55→34.11 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
|
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj






