+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7aci | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | In meso structure of apolipoprotein N-acyltransferase, Lnt, from Escherichia coli in 9.8 monoacylglycerol | ||||||
 要素 要素 | Apolipoprotein N-acyltransferase | ||||||
 キーワード キーワード | MEMBRANE PROTEIN / Nitrilase fold / helical bundle | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報apolipoprotein N-acyltransferase / N-acyltransferase activity / lipoprotein biosynthetic process / outer membrane-bounded periplasmic space / plasma membrane 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.3 Å 分子置換 / 解像度: 2.3 Å | ||||||
 データ登録者 データ登録者 | Smithers, L. / van Dalsen, L. / Boland, C. / Caffrey, M. | ||||||
| 資金援助 |  アイルランド, 1件 アイルランド, 1件
| ||||||
 引用 引用 |  ジャーナル: Cryst.Growth Des. / 年: 2020 ジャーナル: Cryst.Growth Des. / 年: 2020タイトル: 9.8 MAG. A new host lipid for in meso (lipid cubic phase) crystallization of integral membrane proteins 著者: van Dalsen, L. / Smithers, L. / Boland, C. / Weichert, D. / Caffrey, M. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7aci.cif.gz 7aci.cif.gz | 366.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7aci.ent.gz pdb7aci.ent.gz | 251.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7aci.json.gz 7aci.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7aci_validation.pdf.gz 7aci_validation.pdf.gz | 1014.8 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7aci_full_validation.pdf.gz 7aci_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  7aci_validation.xml.gz 7aci_validation.xml.gz | 21.4 KB | 表示 | |
| CIF形式データ |  7aci_validation.cif.gz 7aci_validation.cif.gz | 29.2 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ac/7aci https://data.pdbj.org/pub/pdb/validation_reports/ac/7aci ftp://data.pdbj.org/pub/pdb/validation_reports/ac/7aci ftp://data.pdbj.org/pub/pdb/validation_reports/ac/7aci | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 59280.766 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  遺伝子: lnt, ACU57_00505, AM464_20560, AUQ13_21565, BMA87_17500, BUE81_17200, BvCms2454_02009, BvCmsHHP001_00880, BvCmsKSNP120_02778, BvCmsKSP076_04015, BW690_14780, C5N07_14455, C9Z39_12310, ...遺伝子: lnt, ACU57_00505, AM464_20560, AUQ13_21565, BMA87_17500, BUE81_17200, BvCms2454_02009, BvCmsHHP001_00880, BvCmsKSNP120_02778, BvCmsKSP076_04015, BW690_14780, C5N07_14455, C9Z39_12310, CA593_25875, CI694_20445, CIG45_13010, D0X26_17735, D2185_16075, D3821_19610, D4638_22450, D4718_15390, D9D20_13050, D9D44_16075, D9G69_11750, D9J52_13020, DBQ99_18215, DJ503_07085, DL326_16140, DT034_15925, E2119_09030, E4K55_13820, E4K60_15665, E4K61_12850, EA213_10835, EAI52_07270, EC3234A_6c00760, EC3426_01483, ECTO6_03389, ED307_12710, EEP23_08605, EI021_13075, EI028_12695, EI041_12995, EIZ93_02940, EL75_3125, EL79_3219, EL80_3175, ELT20_10985, EPT01_08450, EXX71_13910, EYD11_16175, FV293_12780, GHR40_20735, GKF74_06475, GKF86_07920, GKF89_05980, GP689_15750, GQE34_06850, GQM17_21560, GRW42_10840, NCTC8500_03946, NCTC9045_04089, NCTC9062_04723, NCTC9969_03714, PGD_02673, RK56_025370, SAMEA3472047_02047, SAMEA3472080_00307, SAMEA3484427_03116, SAMEA3484429_03266, SAMEA3752559_01009, SAMEA3753300_04108, SK85_00657 発現宿主:  参照: UniProt: A0A037YBN4, UniProt: P23930*PLUS, apolipoprotein N-acyltransferase | ||||||
|---|---|---|---|---|---|---|---|
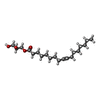
| #2: 化合物 | ChemComp-GOL / #3: 化合物 | ChemComp-LH9 / [( #4: 水 | ChemComp-HOH / | 研究の焦点であるリガンドがあるか | Y | |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.45 Å3/Da / 溶媒含有率: 49.85 % |
|---|---|
| 結晶化 | 温度: 293 K / 手法: 脂質キュービック相法 詳細: 0.1M MES pH 6.0, * %(v/v) MPD, 0.05-0.4 M sodium thiocyanate |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SLS SLS  / ビームライン: X06SA / 波長: 0.999 Å / ビームライン: X06SA / 波長: 0.999 Å |
| 検出器 | タイプ: DECTRIS EIGER X 16M / 検出器: PIXEL / 日付: 2020年6月13日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.999 Å / 相対比: 1 |
| 反射 | 解像度: 2.3→46.57 Å / Num. obs: 26629 / % possible obs: 99.34 % / 冗長度: 12.9 % / Biso Wilson estimate: 38.94 Å2 / CC1/2: 0.99 / Net I/σ(I): 7.5 |
| 反射 シェル | 解像度: 2.3→2.382 Å / Num. unique obs: 2578 / CC1/2: 0.6 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 5N6H 解像度: 2.3→46.57 Å / SU ML: 0.2367 / 交差検証法: FREE R-VALUE / σ(F): 1.34 / 位相誤差: 26.3107 立体化学のターゲット値: GeoStd + Monomer Library + CDL v1.2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 45.2 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.3→46.57 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj