+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6w6v | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Structure of yeast RNase MRP holoenzyme | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | HYDROLASE / ribozyme / RNP / ribonucleoprotein | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ribonuclease MRP activity / nuclear-transcribed mRNA catabolic process, RNase MRP-dependent / intronic box C/D snoRNA processing / nucleolar ribonuclease P complex / ribonuclease MRP complex / ribonuclease P RNA binding / ribonuclease P complex / plasmid partitioning / ribonuclease P / nuclease activity ...ribonuclease MRP activity / nuclear-transcribed mRNA catabolic process, RNase MRP-dependent / intronic box C/D snoRNA processing / nucleolar ribonuclease P complex / ribonuclease MRP complex / ribonuclease P RNA binding / ribonuclease P complex / plasmid partitioning / ribonuclease P / nuclease activity / rRNA primary transcript binding / ribonuclease P activity / tRNA 5'-leader removal / maturation of 5.8S rRNA from tricistronic rRNA transcript (SSU-rRNA, 5.8S rRNA, LSU-rRNA) / telomerase holoenzyme complex / tRNA processing / maturation of 5.8S rRNA / mRNA processing / rRNA processing / nucleolus / RNA binding / nucleus / membrane / cytosol / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3 Å | ||||||
 データ登録者 データ登録者 | Perederina, A. / Li, D. / Lee, H. / Bator, C. / Berezin, I. / Hafenstein, S.L. / Krasilnikov, A.S. | ||||||
| 資金援助 |  米国, 1件 米国, 1件
| ||||||
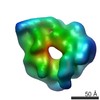
 引用 引用 |  ジャーナル: Nat Commun / 年: 2020 ジャーナル: Nat Commun / 年: 2020タイトル: Cryo-EM structure of catalytic ribonucleoprotein complex RNase MRP. 著者: Anna Perederina / Di Li / Hyunwook Lee / Carol Bator / Igor Berezin / Susan L Hafenstein / Andrey S Krasilnikov /  要旨: RNase MRP is an essential eukaryotic ribonucleoprotein complex involved in the maturation of rRNA and the regulation of the cell cycle. RNase MRP is related to the ribozyme-based RNase P, but it has ...RNase MRP is an essential eukaryotic ribonucleoprotein complex involved in the maturation of rRNA and the regulation of the cell cycle. RNase MRP is related to the ribozyme-based RNase P, but it has evolved to have distinct cellular roles. We report a cryo-EM structure of the S. cerevisiae RNase MRP holoenzyme solved to 3.0 Å. We describe the structure of this 450 kDa complex, interactions between its components, and the organization of its catalytic RNA. We show that some of the RNase MRP proteins shared with RNase P undergo an unexpected RNA-driven remodeling that allows them to bind to divergent RNAs. Further, we reveal how this RNA-driven protein remodeling, acting together with the introduction of new auxiliary elements, results in the functional diversification of RNase MRP and its progenitor, RNase P, and demonstrate structural underpinnings of the acquisition of new functions by catalytic RNPs. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6w6v.cif.gz 6w6v.cif.gz | 562.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6w6v.ent.gz pdb6w6v.ent.gz | 443.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6w6v.json.gz 6w6v.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  6w6v_validation.pdf.gz 6w6v_validation.pdf.gz | 1002.8 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  6w6v_full_validation.pdf.gz 6w6v_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  6w6v_validation.xml.gz 6w6v_validation.xml.gz | 67.2 KB | 表示 | |
| CIF形式データ |  6w6v_validation.cif.gz 6w6v_validation.cif.gz | 106.9 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/w6/6w6v https://data.pdbj.org/pub/pdb/validation_reports/w6/6w6v ftp://data.pdbj.org/pub/pdb/validation_reports/w6/6w6v ftp://data.pdbj.org/pub/pdb/validation_reports/w6/6w6v | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-Ribonucleases P/MRP protein subunit ... , 4種, 4分子 BFGH
| #2: タンパク質 | 分子量: 100559.555 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: P41812, ribonuclease P |
|---|---|
| #5: タンパク質 | 分子量: 18234.959 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: P53218, ribonuclease P |
| #6: タンパク質 | 分子量: 15844.284 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: P38291, ribonuclease P |
| #7: タンパク質 | 分子量: 15530.351 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: P38208, ribonuclease P |
-Ribonuclease P/MRP protein subunit ... , 2種, 3分子 EIJ
| #4: タンパク質 | 分子量: 19601.590 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: P28005, ribonuclease P |
|---|---|
| #8: タンパク質 | 分子量: 32270.262 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: P38786, ribonuclease P |
-Ribonuclease MRP protein subunit ... , 2種, 2分子 KL
| #9: タンパク質 | 分子量: 22578.955 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: P40993 |
|---|---|
| #10: タンパク質 | 分子量: 23657.668 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: Q12530 |
-RNA鎖 / タンパク質 , 2種, 2分子 AD
| #1: RNA鎖 | 分子量: 108767.664 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: GenBank: 4037 |
|---|---|
| #3: タンパク質 | 分子量: 32933.168 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  株: ATCC 204508 / S288c / 参照: UniProt: P38336 |
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: CELL / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Ribonuclease MRP / タイプ: COMPLEX / Entity ID: all / 由来: NATURAL |
|---|---|
| 分子量 | 値: 0.45 MDa / 実験値: NO |
| 由来(天然) | 生物種:  |
| 緩衝液 | pH: 8 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 試料支持 | 詳細: unspecified |
| 急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD |
| 撮影 | 電子線照射量: 39 e/Å2 / 検出モード: COUNTING フィルム・検出器のモデル: FEI FALCON III (4k x 4k) |
- 解析
解析
| ソフトウェア | 名称: PHENIX / バージョン: 1.18.1_3865: / 分類: 精密化 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア | 名称: PHENIX / バージョン: 1.18.1 / カテゴリ: モデル精密化 | ||||||||||||||||||||||||
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 155205 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj





























