| 登録情報 | データベース: PDB / ID: 6q2z
|
|---|
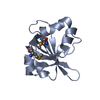
| タイトル | NMR solution structure of the HVO_2922 protein from Haloferax volcanii |
|---|
 要素 要素 | UPF0339 family protein |
|---|
 キーワード キーワード | UNKNOWN FUNCTION / conserved hypothetical protein |
|---|
| 機能・相同性 | YegP-like / Domain of unknown function DUF1508 / Domain of unknown function (DUF1508) / YegP-like superfamily / Double Stranded RNA Binding Domain / 2-Layer Sandwich / Alpha Beta / UPF0339 family protein 機能・相同性情報 機能・相同性情報 |
|---|
| 生物種 |  Haloferax volcanii (古細菌) Haloferax volcanii (古細菌) |
|---|
| 手法 | 溶液NMR / simulated annealing with torsion angle dynamics / cartesian angle dynamics / molecular dynamics |
|---|
 データ登録者 データ登録者 | Kubatova, N. / Jonker, H.R.A. / Saxena, K. / Richter, C. / Marchfelder, A. / Schwalbe, H. |
|---|
| 資金援助 |  ドイツ, 2件 ドイツ, 2件 | 組織 | 認可番号 | 国 |
|---|
| German Research Foundation | SPP 2002 priority program - 313752925 |  ドイツ ドイツ | | State of Hesse - BMRZ |  ドイツ ドイツ |
|
|---|
 引用 引用 |  ジャーナル: Chembiochem / 年: 2020 ジャーナル: Chembiochem / 年: 2020
タイトル: Solution Structure and Dynamics of the Small Protein HVO_2922 from Haloferax volcanii.
著者: Kubatova, N. / Jonker, H.R.A. / Saxena, K. / Richter, C. / Vogel, V. / Schreiber, S. / Marchfelder, A. / Schwalbe, H. |
|---|
| 履歴 | | 登録 | 2018年12月3日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2019年6月12日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2020年1月29日 | Group: Database references / カテゴリ: citation
Item: _citation.journal_volume / _citation.page_first ..._citation.journal_volume / _citation.page_first / _citation.page_last / _citation.title / _citation.year |
|---|
| 改定 1.2 | 2024年7月3日 | Group: Data collection / Database references / Other
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_database_status
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.status_code_nmr_data |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Haloferax volcanii (古細菌)
Haloferax volcanii (古細菌) データ登録者
データ登録者 ドイツ, 2件
ドイツ, 2件  引用
引用 ジャーナル: Chembiochem / 年: 2020
ジャーナル: Chembiochem / 年: 2020 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6q2z.cif.gz
6q2z.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6q2z.ent.gz
pdb6q2z.ent.gz PDB形式
PDB形式 6q2z.json.gz
6q2z.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/q2/6q2z
https://data.pdbj.org/pub/pdb/validation_reports/q2/6q2z ftp://data.pdbj.org/pub/pdb/validation_reports/q2/6q2z
ftp://data.pdbj.org/pub/pdb/validation_reports/q2/6q2z リンク
リンク 集合体
集合体
 要素
要素 Haloferax volcanii (strain ATCC 29605 / DSM 3757 / JCM 8879 / NBRC 14742 / NCIMB 2012 / VKM B-1768 / DS2) (古細菌)
Haloferax volcanii (strain ATCC 29605 / DSM 3757 / JCM 8879 / NBRC 14742 / NCIMB 2012 / VKM B-1768 / DS2) (古細菌)
 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj HSQC
HSQC